芦笋老茎堆肥中嗜热放线菌的分离和鉴定
2013-12-23陈婷婷王丽芳韩建荣
陈婷婷,王丽芳,王 琪,韩建荣
(1.山西大学生物技术研究所,山西太原030006;2.山西大学生命科学学院,山西太原030006)
芦笋(Asparagus officinalis L.)为百合科天门冬属多年生草本植物,又名石刁柏、龙须菜,原产于欧洲、北非和西亚[1-3]。因其较高的药食兼用价值而在我国各地广泛栽培,山西是全国最大的芦笋产区[4]。芦笋的可食部分是其嫩茎,每年过了收获季节以后,芦笋的地上茎仍要继续生长,可长到1.5~2.0 m,继续生长的地上茎称为芦笋老茎。芦笋老茎主要由纤维素(40.2%)、半纤维素(19.8%)和木质素(17.9%)构成[5]。与芦笋嫩茎相比,老茎由于木质纤维化程度较高,失去了食用价值,致使大量的芦笋老茎收割后没有被充分利用,只能任其自然腐败或直接焚烧,这样既造成资源的极大浪费,也带来了严重的环境污染。
针对如何利用芦笋老茎资源的问题,山西大学生命科学学院微生物实验室开展了一系列研究工作,已经证明了芦笋老茎经堆制发酵后可用来栽培姬松茸(Agaricus blazei)[6]。研究表明,嗜热放线菌是蘑菇堆肥高温期的主要优势菌群[7-8]。蘑菇堆肥是一个复杂而又独特的微生态系统,是一种由群落结构演替非常迅速的多个微生物群体共同作用有效分解有机物的动态生化过程[9]。目前,有关于双孢菇培养料(主要是麦秆)发酵过程中微生物群落结构的研究[10],还有针对猪粪堆肥[11]、污泥[12]、生活垃圾堆肥[13]等微生物群落的研究,而针对芦笋老茎堆肥的研究还未见报道。
本试验对芦笋老茎堆肥中的嗜热放线菌进行了初步的分离鉴定研究,旨在为制备芦笋老茎堆料发酵菌剂及调控堆料过程提供理论依据。
1 材料和方法
1.1 供试材料
1.1.1 堆肥配方 芦笋老茎70%,棉籽壳20%,豆饼7.5%,过磷酸钙1%,石膏粉1%,尿素0.5%(要求芦笋老茎及其他辅料无霉变,切成20 cm左右的小段,然后将芦笋老茎浸透水,预湿后与其他辅料搅拌建堆)。料堆一般底宽120~150 cm,上宽80~90 cm,高80~100 cm。
1.1.2 堆制过程 按2次发酵法[14-15]发酵堆料,室外前发酵18 d,共翻堆4次;移入室内进行后发酵,60℃保持10 h,50℃保持4 d。后发酵结束后,调整培养料的含水量到65%,pH值为7.0~7.5。
1.1.3 采样 以芦笋老茎堆肥的堆体四周及中心处为采样点,采样深度为20 cm左右,采用四分法采样。在前发酵每次翻堆前和后发酵前后进行采样,共采6次,即建堆后第4天(A)、第6天(B)、第9天(C)、第11天(D)、第15天(E)和第22天(F),于4℃保存。试验前将样品风干2 d,放入烘箱,置于120℃下1 h[16-17]。
1.1.4 培养基 高氏Ⅰ号培养基:可溶性淀粉20 g,NaCl 0.5 g,KNO31 g,K2HPO40.5 g,MgSO40.5 g,FeSO40.01 g,琼脂20 g,蒸馏水1 000 mL,pH值7.2~7.4。121℃灭菌20 min。倒平板时,每200 mL培养基添加10 mg的K2Cr2O7[18]作抑菌剂。
1.2 试验方法
1.2.1 菌株的分离 准确称取样品1 g,加入装有99 mL无菌水并带有玻璃珠的三角瓶中,振荡约30 min,吸取1 mL样品悬液加入装有9 mL无菌水的试管中,吹洗多次,让菌液混合均匀,即成10-3稀释液。依此类推,分别制成10-4,10-5稀释液。分别吸取10-3,10-4,10-5悬液0.2 mL涂布于高氏Ⅰ号平板上,45℃培养3 d,观察并挑取单菌落,在平板上再划线纯化。将纯化后的菌株分别按“样品编号+G(代表放线菌)+数字”编号。
1.2.2 菌株的鉴定
1.2.2.1 形态观察 平板划线培养3 d后,观察菌落的大小、形态、正反面颜色、厚薄、质地、表面状况及是否产色素。插片培养2 d后,观察菌丝、孢子丝的形状和特点。美兰染色参照文献[19]。
1.2.2.2 16SrDNA的扩增 选取菌落形态有明显区别的放线菌菌株通过菌落PCR法[20]提取16SrDNA,扩增引物采用16SrDNA通用引物:27F,5′-AGAGTTTGATCCTGGCTCAG-3′;1541R,5′-ACGGTTACCTTGTTACGACTT-3′。PCR扩增体系:10×TransEasy Taq Buffer 2.5μL,dNTP1μL,引物(20μmol/L)各2.5μL,Trans Easy Taq DNA polymerase 0.5μL,加模板7.5μL,ddH2O补至25μL。各试剂加好后,轻轻混匀。PCR扩增条件:预变性94℃,5 min;变性94℃,30 s;退火55℃,30 s;延伸72℃,90 s;35个循环后,72℃保温10 min。PCR扩增产物和回收产物均经1%的琼脂糖凝胶电泳分离,UV凝胶成像系统检测。
1.2.2.3 16SrDNA片段PCR产物的连接、转化、测序 向无菌离心管中加入目的片段16SrDNA回收产物4μL和载体1μL,混匀后于25℃反应30 min,4℃反应16 h进行连接。将5μL连接产物全部加入感受态细胞(100μL)中,温和混匀,冰浴放置30 min;迅速转入42℃水浴,热激30 s,再迅速冰浴冷却2 min后,加入900μL的LB培养基,37℃下90 r/min振荡培养50 min;然后离心,弃掉大部分上清,剩下的用移液器吹匀,加入10μLIPTG(质量分数20%)和20μL X-gal(20 mg/mL),混匀后轻轻涂到LB平板(含有氨苄青霉素);37℃培养12~16 h后可见蓝色菌落。挑取白色菌落接种于LB液体培养基(含有氨苄青霉素),37℃下200 r/min过夜培养;将培养好的菌液分成2管,1管提取质粒DNA作酶切筛选用,1管留作测序用。Eco RI限制性内切酶酶切筛选:向无菌EP管中依次加入8μL ddH2O,0.5μL Eco R I Buffer,1μL质粒DNA,0.5μL Eco R I,于37℃反应2 h。反应结束后,产物经0.7%琼脂糖凝胶电泳检测,含有目的片段重组子的大小共约4 600 bp。选取含目的片段的重组子送华大基因研究中心进行测序。本试验样品来自于芦笋老茎堆肥,克隆编号方法采用“ASC+重组子号”。
1.2.2.4 菌株16SrDNA序列的同源性分析 将测序获得的阳性克隆的16SrDNA序列运用CHECK_CHIMERA程序在线进行嵌合体检验并去除明显的Chimera序列。在GenBank数据库中用VECTOR SCREEN程序剔除载体序列,经BLAST比对后对菌株进行同源性分析。将获得的序列与GenBank数据库中最相似的序列一起使用软件Clustal X 1.8和MEGA 3.1,采用邻近法(Neighor Joining),在Kimura双参数模型(Kimura 2-parameter)下,绘制相关菌株的系统发育树和Bootstrap检验系统树,自展值设为1 000。
1.2.2.5 16SrDNA序列在GenBank数据库中的登录号 本研究所获得16SrDNA序列已提交Gen-Bank数据库,登录号分别为:JQ358562,JQ358563,JQ358585,JQ358587,JQ358588,JQ358594~JQ35 8596,JQ358598,JQ358599,JQ358602,JQ358607,JQ358565,JQ358567,JQ358569,JQ358572~JQ35 8575,JQ358577,JQ358578,JQ358580,Q358583。
2 结果与分析
2.1 菌株分离
采用稀释涂布法从芦笋老茎堆肥的6个样品中分离到菌落形态有明显区别的23个放线菌菌株,其中,A样品3株,B样品8株,C样品1株,D样品2株,E样品3株,F样品6株。编号分别为:AG1~AG3;BG1~BG8;CG1;DG1,DG2;EG1~EG3;FG1~FG6。
2.2 菌株的鉴定
2.2.1 形态观察 23株放线菌经形态观察都属于链霉菌属(Streptomyces),都有螺旋或(和)波曲形状的孢子丝;基丝生长良好,多分支,不形成横隔,也不断裂。菌落形状以圆形为主,其中,BG7菌落是无规则形;菌落正面颜色以不同层次的灰色和黑色为主,其中,BG7和FG5菌落为灰绿色;一般菌落凸起,表面粉粒状,其中,AG2,BG2,CG1,DG1和EG1菌落表面较平坦湿润。23株链霉菌中,有4株产色素,分别是AG2产褐色色素、BG3产酱色色素、EG2和FG4产黄色色素。
2.2.2 16SrDNA的扩增 每株菌取10个单菌落,加1 mL ddH2O煮沸5 min,12 000 r/min离心2 min,取上清液作模板进行PCR扩增。其中,1株放线菌的PCR扩增产物如图1所示,可见获得条带约为1.6 kb的16SrDNA序列。
2.2.3 16SrDNA序列测定和比对 测序后,将23株链霉菌的16SrDNA序列与GenBank数据库中已发表的序列进行比对(表1)。
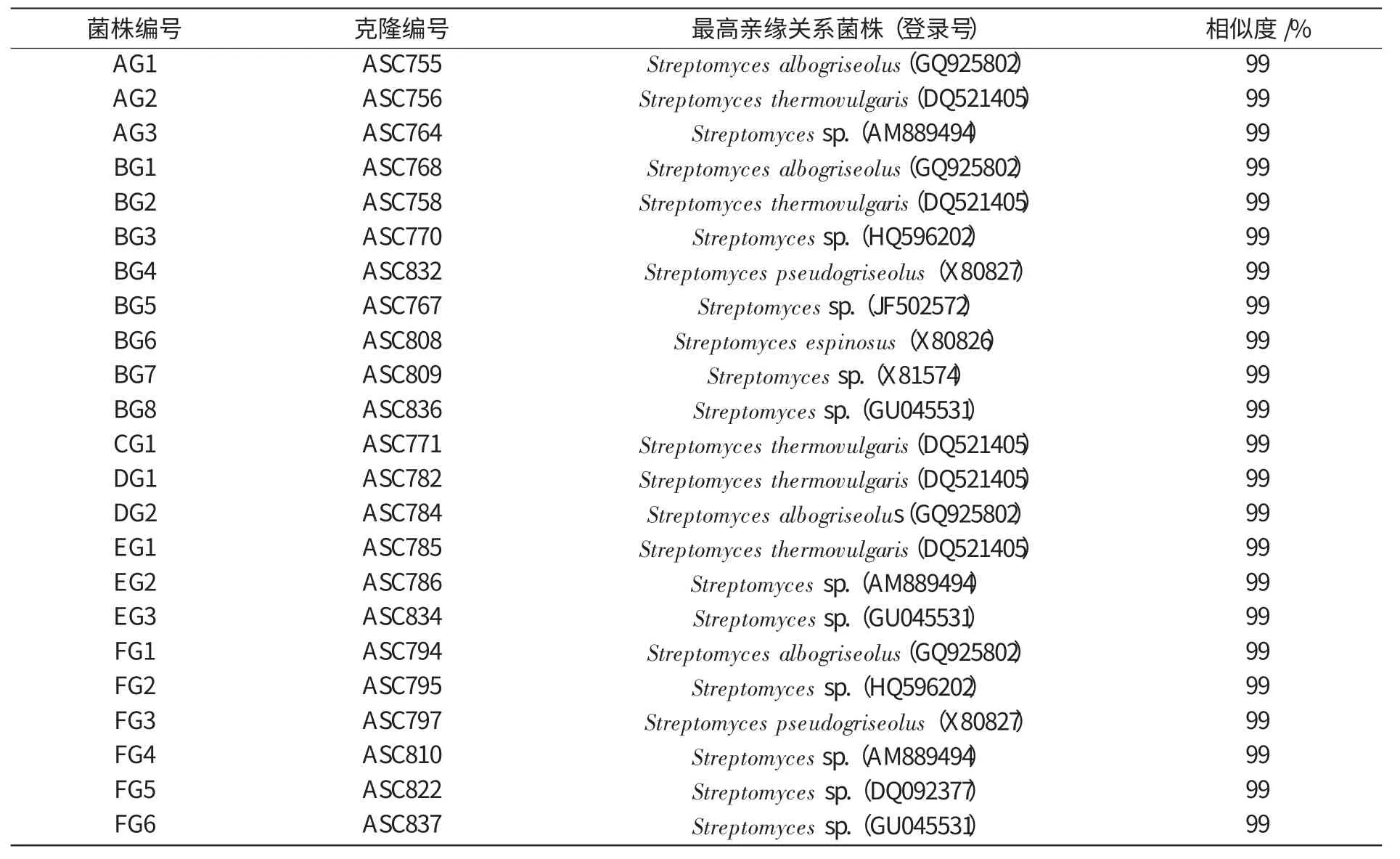
表1 样品中的放线菌菌株按照16S rDNA 序列的比对结果
由表1可知,A样品3个菌株分别与白浅灰链霉菌Streptomyces albogriseolus(GQ925802),热普通链霉菌 Streptomyces thermovulgaris(DQ521405),Streptomyces sp.(AM889494)的同源性最高;B样品8个菌株分别与白浅灰链霉菌Streptomyces albogriseolus(GQ925802),热普通链霉菌Streptomyces thermovulgaris(DQ521405),Streptomyces sp.(HQ596202),假浅灰链霉菌Streptomyces pseudogriseolus(X80827),Streptomyces sp.(JF502572),刺棘链霉菌Streptomyces espinosus(X80826),Streptomyces sp.(X81574),Streptomyces sp.(GU045531)的同源性最高;C样品1个菌株与热普通链霉菌Streptomyces thermovulgaris(DQ521405)的的同源性最高;D样品2个菌株分别与热普通链莓菌Streptomyces thermovulgaris(DQ521405),白浅灰链霉菌Streptomyces albogriseolus (GQ925802)的同源性最高;E样品3个菌株分别与热普通链霉菌Streptomyces thermovulgaris(DQ521405),Streptomyces sp.(AM889494),Streptomyces sp.(GU045531)的同源性最高;F样品6个菌株分别与白浅灰链霉菌Streptomyces albogriseolus(GQ925802),Streptomyces sp.(HQ596202),假浅灰链霉菌Streptomyces pseudogriseolus(X80827),Streptomyces sp.(AM889494),Streptomyces sp.(DQ092377),Streptomyces sp.(GU045531)的同源性最高。
由测序比对结果可知,23株链霉菌中,4株的序列与白浅灰链霉菌的同源性最高,5株的序列与热普通链霉菌的同源性最高,2株的序列与假浅灰链霉菌的同源性最高,1株的序列与刺棘链霉菌的同源性最高,有11株在GenBank数据库中未找到与其相似的已知链霉菌种的序列,分类地位待定。结果说明,芦笋老茎堆肥中优势的嗜热可培养放线菌主要是白浅灰链霉菌和热普通链霉菌。
根据6个样品中23株链霉菌的16SrDNA序列和同源性最高的已知菌株的序列构建的系统发育树如图2所示。

3 讨论
本研究在分离嗜热放线菌时,首先对样品进行了物理处理,即120℃烘1 h,然后在分离的高氏Ⅰ号培养基中添加抑菌剂,一方面可以促进放线菌孢子的萌发,另一方面可以除掉大部分的细菌、真菌。母连军[16]、司美茹等[21-22]发现,适当的热处理可以促进放线菌孢子活化,能有效地提高放线菌的种类和数量;在分离的培养基中添加化学抑菌剂或抗生素,也可提高放线菌的出菌量。司美茹等[21]利用9种培养基来分离特殊环境中的放线菌发现,高氏Ⅰ号分离得到的放线菌数量和种类最多。本试验在分离的过程中,完全没有细菌、霉菌的干扰,而且嗜热放线菌的出菌量较好。
本试验通过菌落PCR方法,直接煮沸菌体导致细胞破裂,以暴露出的DNA为模板进行PCR扩增。结果证明,该方法可以对芦笋老茎堆肥中分离到的放线菌进行大规模的鉴定。菌落PCR方法不仅大大减少了工作量,而且提高了试验效率。从电泳图上可以看出,菌落PCR还存在着一些非特异性的结合,可能是因为模板中存在其他蛋白质和多糖等杂质影响或抑制了扩增反应,导致扩增效果不太理想。但是如果用作定性检测,菌落PCR不仅能得到与常规PCR相同的结论,还可以节省大量的人力和物力,是一种切实可行的检测方法。
微生物群落及其结构在堆料物质转化过程中起着非常重要的作用,堆料中微生物数量及种群分布与堆肥中有机物质成分和含量、微生物间的相互作用等多种因素密切相关[9]。本研究结果表明,芦笋老茎堆肥中的嗜热放线菌主要是链霉菌,而且几乎分离到的放线菌都属于链霉菌。23株链霉菌中,有11个菌株在GenBank数据库中未找到与其相似的已知菌株的序列,分类地位待定,这为下一步从芦笋老茎堆肥中筛选分离有价值的新嗜热可培菌提供了可能。本研究只分离到属于链霉菌属的嗜热放线菌,没有分离到像高温放线菌(Thermoactinomyces) 和高温单孢菌(Thermomonospora)等属的嗜热放线菌,这可能是因为在芦笋老茎堆肥过程中起作用的嗜热放线菌主要是链霉菌,也可能是因为选用的高氏Ⅰ号分离培养基不适合其他嗜热放线菌的生长。在下一步的分离工作中,有必要选择更多的分离培养基来进行比较研究。
[1]孙春艳,赵伯涛,郁志芳,等.芦笋的化学成分及药理作用研究进展[J].中国野生植物资源,2004,23(5):1-5.
[2]毛兴平,朱志强,张平,等.绿芦笋采后品质变化概述[J].天津农业科学,2010,16(4):123-127.
[3]娜日苏.石刁柏栽培技术[J].内蒙古农业科技,2008(6):110.
[4]郜春花,卢朝东.山西省芦笋生产现状及发展对策探讨[J].山西农业科学,2007,35(1):15-18.
[5]申挺挺,郭珺,李欣欣,等.杏鲍菇、白灵菇对芦笋老茎中木质纤维素的分解利用 [J].山西农业科学,2011,39(11):1170-1173.
[6]Wang Q,Li B B,Li H.Yield,dry matter and polysaccharides content of themushroom Agaricus blazei produced on asparagus straw substrate[J].Scientia Horticulturae,2010,125:16-18.
[7]Mitali D,Todd V R,Laura G L.Diversity of fungi,bacteria and actinomyceteson leavesdecomposingin astream[J].Applied and Environmental Microbiology,2007,73(3):756-767.
[8]Xi BD,Liu H L,Zeng GM,et al.Composting MSW and sewage sludgewith effectivecomplex microorganisms[J].Journal of Environmental Sciences,2002,14(2):264-268.
[9]万水霞,郭熙盛,朱宏赋,等.自然堆肥过程中微生物群落的动态变化[J].安徽农业科学,2009,37(28):13710-13711.
[10]何丽鸿,于荣利,陈明杰,等.采用ARDRA研究双孢蘑菇培养料后发酵过程中的细菌群落结构(Ⅰ)[J].食用菌学报,2008,15(4):11-15.
[11]邓强,杨向科,王慧杰.微生态调节剂对猪粪堆肥过程中微生物群落的影响[J].河南农业科学,2007(8):75-77.
[12]王春铭,雷恒毅,王国惠,等.城市污泥摸拟堆肥过程中高温菌群的筛选、鉴定及降解效果 [J].环境科学学报,2007,27(6):979-986.
[13]党秋玲,刘驰,希北斗,等.生活垃圾堆肥过程中细菌群落演替规律[J].环境科学研究,2011,24(2):236-242.
[14]Wuest PJ.Compost and the composting-process[J].Mushroom News,1978,11:25-29.
[15]池致念,王泽生,廖剑华,等.蘑菇培养料集中二次发酵的初步研究[J].中国食用菌,2002,21(1):30-32.
[16]母连军,胡永松,王忠彦.放线菌分离方法的进展[J].四川食品工业科技,1996(3):4-6.
[17]李新,纪明山.土壤中拮抗放线菌的分离和筛选[J].河南农业科学,2008(1):58-60.
[18]冯轶男,杨润清.放线菌分离与筛选方法的研究进展[J].生物技术,2010,20(4):95-98.
[19]沈萍,陈向东.微生物学实验[M].北京:高等教育出版社,2007.
[20]唐晔盛,李英.菌落PCR在大规模基因组测序中的应用[J].生物化学与生物物理进展,2002,29(2):316-318.
[21]司美茹,薛泉宏,来航线.放线菌分离培养基筛选及杂菌抑制方法研究[J].微生物学通报,2004,31(2):61-65.
[22]宋影,王潮钟,郭世英.放线菌菌株Ys.03的分离筛选及其抗病毒作用初步研究[J].华北农学报,2010,25(6):164-166.
