西伯利亚鲟糖异生途径关键酶基因全长cDNA的克隆和序列分析
2013-12-20苏晓鸥吴秀峰郑银桦
宫 官 薛 敏 王 嘉* 苏晓鸥 吴秀峰 郑银桦 韩 芳
(1.中国农业科学院饲料研究所,国家水产饲料安全评价基地,北京 100081;2.中国农业科学院农业质量标准与检测技术研究所,北京 100081)
在饲料三大能源物质中,碳水化合物(糖)是最廉价的能源。但在鱼类,尤其是肉食性鱼类中,摄食高碳水化合物饲料后会表现出持久的餐后高血糖现象,最终导致生长性能的下降[1-2]。目前为止,肉食性鱼类对高碳水化合物不耐受的生物学机制仍然不完全清楚,而近期的观点认为是由于其肝脏中缺乏对维持机体葡萄糖平衡的糖异生的调节机能所致[3-4]。因此,系统地研究肉食性鱼类应对不同营养状况时糖异生途径的调节响应,对于阐明鱼类碳水化合物不耐受机制,提高鱼类对碳水化合物的利用能力,发挥其对蛋白质的节约效应,降低饲料成本,降低养殖过程中氮、磷排放等均具有重要的理论指导意义和实际应用价值。
糖异生作用是机体在饥饿状态下,通过各种非糖苷底物,如乳酸盐、甘油和α-酮酸等,经过一系列酶促反应合成内源性葡萄糖的过程,其在肝脏中与糖酵解作用共同维持机体的葡萄糖平衡[4]。糖异生代谢途径中的关键酶包括磷酸烯醇式丙酮酸羧基酶(phosphoenolpyruvate carboxykinase,PEPCK;EC 4.1.1.32)、果糖 -1,6 -二磷酸酶(fructose-1,6-bisphosphatase,FBPase;EC 3.1.3.11)和葡萄糖 -6-磷酸酶(glucose-6-phosphatase,G6Pase;EC 3.1.3.9)。这些酶类编码基因的cDNA全长或部分序列已经从多种鱼类中克隆出,包括虹鳟(Oncorhynchus mykiss)、大西洋鲑(Salmon salar)、真鲷(Sparus aurata)、鲤鱼(Cyprinus carpio)和翘嘴红鲌(Erythroculter ilishaeformis)等。研究证明,肉食性鱼类,如虹鳟[5]、大西洋鲑[6]、真 鲷[7-10]和 欧 鲈 (Dicentratchus labrax)[11-12]等肝脏糖异生途径关键酶基因 mRNA表达量和活性不受饲料碳水化合物含量的影响。而在杂食性鱼类,如鲤鱼的研究中,饲料中升高的碳水化合物含量显著抑制了肝脏FBPase的活性[13]。由此可见,肉食性鱼类缺乏外源碳水化合物对糖异生作用的抑制机制可能是其无法有效控制血糖水平,无法有效利用碳水化合物的原因[4]。
西伯利亚鲟(Acipenser baerii)属鲟形目,鲟科,是一类古老的软骨硬鳞鱼类,在鱼类进化史上占有重要地位,常常被作为鱼类生物学的模型动物。世界鱼类数据库(FishBase,www.fishbase.org)将西伯利亚鲟描述为肉食性鱼类,但盛洪建[14]报道,在理想蛋白质模型下,在40%和36%饲料蛋白质水平下由混合植物蛋白质100%替代鱼粉对西伯利亚鲟的生长和蛋白质储积没有显著影响;吴秀峰等[15]报道,在理想蛋白质模式下,脱酚棉籽粉可等蛋白质替代40%的鱼粉而不会对西伯利亚鲟幼鱼的生长产生显著影响。以上研究结果与已报道的肉食性硬骨鱼类对植物蛋白质利用效果的结果[16-17]差异较大。植物蛋白质中含有的大量碳水化合物是造成肉食性鱼类不能对其很好利用的重要原因[18-19]。然而,西伯利亚鲟能够很好地利用植物蛋白质源,说明其能很好的利用其中的碳水化合物,与典型的肉食性硬骨鱼类相比可能存在不同的糖代谢调控机理。
本研究拟通过简并引物反转录聚合酶链式反应(RT-PCR)和cDNA末端快速扩增(RACE)技术相结合的方法,克隆西伯利亚鲟肝脏中糖异生途径关键酶基因的cDNA全长序列,为进一步研究西伯利亚鲟在不同营养状况下糖异生途径的调控能力、比较其与肉食性硬骨鱼类的异同奠定分子生物学基础。
1 材料与方法
1.1 试验材料
试验所用西伯利亚鲟购自于北京市水产科学研究所房山基地。
1.2 引物设计与合成
根据NCBI GenBank中已知其他物种的C型PEPCK(PEPCK-C)、M 型 PEPCK(PEPCK-M)、FBPase和G6Pase氨基酸序列寻找相应的保守片段,采用CodeHop原理[20]设计简并引物克隆西伯利亚鲟相应基因的保守片段。根据扩增到的保守片段,使用软件 Primer 5.0设计用于 3'-和 5'-RACE扩增的特异性引物。所有引物均由生工生物工程(上海)有限公司合成。本试验中所用到的引物序列及相应的退火温度(Tm)见表1。
1.3 总RNA提取
解剖西伯利亚鲟,取新鲜肝脏,经液氮研磨后用RNAiso Plus(TaKaRa,大连)抽提总 RNA。总RNA用变性琼脂糖凝胶电泳显示28S和18S条带,验证所提取的总RNA的完整性。采用Power-WaveTMXS2全波长酶标仪(BioTek,美国)检测其在260 nm处的吸光值与在280 nm处的吸光值的比值(A260/A280),并对所提取的总RNA进行定量。
1.4 保守片段克隆
cDNA第1链的合成采用PrimeScriptⅡ 1stStrand cDNA Synthesis Kit(TaKaRa,大连),每个反转录反应使用约1.0μg肝脏总RNA,操作步骤参照说明书进行。分别采用各基因的简并引物进行保守片段聚合酶链式反应(PCR)扩增,PCR反应体系包含合成的 cDNA第1链1μL、10×PCR Buffer 2.5 μL、TaKaRa Ex Taq 0.625 U、dNTP 200μmol/L、上游引物 0.4 μmol/L、下游引物0.4μmol/L,加灭菌水至总体积 25 μL。PCR 扩增条件为:94 ℃,30 s;Tm,30 s;72 ℃,1 min;共35个循环。PCR产物经电泳后,在紫外灯下切取与目标序列大小一致的片段,使用Agarose Gel DNA Purification Kit Ver.2.0(TaKaRa,大连)对扩增片段进行纯化。纯化产物与pMD19-T Vctor(TaKa-Ra,大连)连接,转化入大肠杆菌(E.coli)JM109感受态细胞(TaKaRa,大连)中,经蓝白斑筛选后,对有插入片段的阳性克隆进行测序(北京华大基因服务公司)。

表1 本试验中所用到的引物序列及相应的退火温度Table 1 Primer sequence and Tm used in this experiment
1.5 3'-RACE 扩增
3'-RACE扩增采用3'-Full RACE Core Set Ver.2.0(TaKaRa,大连),操作步骤参照说明书进行。采用1.0μg肝脏总RNA和3'-RACE Adaptor合成cDNA第1链,经过2轮嵌套PCR反应扩增各基因的3'末端。首先以合成的cDNA第1链为模板,分别以试剂盒提供的3'-RACE Outer primer和所设计的基因特异性3'-RACE Outer primer为引物进行第1轮PCR扩增,反应条件为:94℃,30 s;Tm,30 s;72 ℃,1.5 min;共35 个循环。再以100倍稀释的Outer PCR产物为模板,分别以试剂盒提供的3'-RACE Inner primer和所设计的基因特异性3'-RACE Inner primer为引物进行第2轮PCR扩增,反应条件为:94℃,30 s;Tm,30 s;72℃,1.5 min;共30个循环。对Inner PCR产物进行电泳、切胶纯化、连接、转化和测序。
1.6 5'-RACE 扩增
5'-RACE扩增采用SMART TM RACE cDNA Amplification Kit(Clontech,美国),操作步骤参照说明书进行。采用1.0μg肝脏总 RNA、5'-CDS Primer A和SMARTⅡA Oligo合成cDNA第1链,以 Advantage 2 PCR Enzyme System(Clontech,美国)、UPM引物和所设计的基因特异性5'-RACE引物进行PCR扩增。扩增采用Touch-Down PCR程序:94 ℃,30 s;72℃,2.5 min;5个循环;94℃,30 s;70℃,30 s;72℃,2 min;5个循环;94℃,30 s;68℃,30 s;72℃,2 min;30个循环。对PCR产物进行电泳、切胶纯化、连接、转化和测序。
1.7 序列分析、同源性比对及系统进化树构建
将测序所获得的核心片段、3'和5'区域拼接即获得了糖异生关键酶各基因的全长序列。将拼接结果与NCBI GenBank中的已知序列作blastx同源性分析,用 DNAMAN(VERSION 5.2.2)软件分析所得cDNA序列及开放阅读框,预测编码氨基酸序列。根据GenBank中已报道的糖异生关键酶各基因的蛋白质序列用Lasergene 7.0程序中MegAlign进行多序列比对和一致性数值计算。采用MEGA 5.1软件,选择邻接法(neighbor-joining,NJ)构建系统进化树[21]。
2 结果
2.1 全长序列和结构分析
西伯利亚鲟PEPCK-C全长cDNA序列及推测编码的氨基酸序列见图1。cDNA全长2 598 bp,包括 174 bp的 5'非翻译区、1 869 bp的开放阅读框及555 bp的3'非翻译区;开放阅读框编码1个由622个氨基酸组成的蛋白质,预测其分子质量为 69.62 ku,等电点为5.94。经过 blastx比对发现其与其他物种PEPCK-C具有很高的相似性,具有与草酰乙酸结合的PEPCK特有结构域以及与三磷酸鸟苷(GTP)三磷酸链结合的激酶1和激酶2基序。将该序列提交至GenBank获得登录号为JQ995143。

图1 西伯利亚鲟PEPCK-C全长cDNA序列和预测的氨基酸序列(GenBank登录号JQ995143)Fig.1 PEPCK-C full length cDNA sequence and deduced amino acid sequence of Siberian sturgeon(Acipenser baerii Brandt)(GenBank accession No.JQ995143)
西伯利亚鲟PEPCK-M全长cDNA序列及推测编码的氨基酸序列见图2。cDNA全长3 277 bp,包括 279 bp的 5'非翻译区、1 935 bp的开放阅读框及1 063 bp的3'非翻译区;开放阅读框编码1个由644个氨基酸组成的蛋白质,预测其分子质量为71.11 ku,等电点为8.23。经过blastx比对发现其与其他物种PEPCK-M具有很高的相似性,具有与草酰乙酸结合的PEPCK特有结构域以及与GTP三磷酸链结合的激酶1和激酶2基序。将该序列提交至GenBank获得登录号为JQ995142。

图2 西伯利亚鲟PEPCK-M全长cDNA序列和预测的氨基酸序列(GenBank登录号JQ995142)Fig.2 PEPCK-M full length cDNA sequence and deduced amino acid sequence of Siberian sturgeon(Acipenser baerii Brandt)(GenBank accession No.JQ995142)
西伯利亚鲟FBPase全长cDNA序列及推测编码的氨基酸序列见图3。cDNA全长1 372 bp,包括71 bp的5'非翻译区、1 017 bp的开放阅读框及284 bp的3'非翻译区;开放阅读框编码1个由338个氨基酸组成的蛋白质,预测其分子质量为36.60 ku,等电点为7.13。经过 blastx比对发现其与其他物种FBPase具有很高的相似性。将该序列提交至GenBank获得登录号为JF834908。

图3 西伯利亚鲟FBPase全长cDNA序列和预测的氨基酸序列(GenBank登录号JF834908)Fig.3 FBPase full length cDNA sequence and deduced amino acid sequence of Siberian sturgeon(Acipenser baerii Brandt)(GenBank accession No.JF834908)
西伯利亚鲟G6Pase全长cDNA序列及推测编码的氨基酸序列见图4。cDNA全长2 625 bp,包括69 bp的5'非翻译区、1 080 bp的开放阅读框及1 476 bp的3'非翻译区;开放阅读框编码1个由359个氨基酸组成的蛋白质,预测其分子质量为40.62 ku,等电点为 8.69。经过 blastx 比对发现其与其他物种G6Pase具有很高的相似性,具有磷酸酶家族保守结构域PAP2。将该序列提交至GenBank获得登录号为JF834907。
2.2 多序列比对分析
将西伯利亚鲟PEPCK-C氨基酸序列与其他物种的PEPCK-C氨基酸序列进行同源性比对,结果见图5。分析表明,西伯利亚鲟PEPCK-C氨基酸序列与大西洋鲑同源性最高,为80.5%,与斑马鱼(Danio rerio)、军曹鱼(Rachycentron canadum)、人(Homo sapiens)、小鼠(Mus musculus)、鸡(Gallus gallus)和鸭嘴兽(Ornithorhynchus anatinus)的同源性分别为 78.3%、78.0%、77.5%、78.0%、78.1% 和 77.3%。
将西伯利亚鲟PEPCK-M氨基酸序列与其他物种的PEPCK-M氨基酸序列进行同源性比对,结果见图6。分析表明,西伯利亚鲟PEPCK-M氨基酸序列与斑马鱼同源性最高,为78.3%,与大西洋鲑、军曹鱼、人、小鼠、鸡和鸭嘴兽的同源性分别为74.2%、72.8%、72.7%、71.3%、64.6% 和72.8% 。

图4 西伯利亚鲟G6Pase全长cDNA序列和推测的氨基酸序列(GenBank登录号JF834907)Fig.4 G6Pase full length cDNA sequence and deduced amino acid sequence of Siberian sturgeon(Acipenser baerii Brandt)(GenBank accession No.JF834907)
将西伯利亚鲟FBPase氨基酸序列与其他物种的FBPase氨基酸序列进行同源性比对,结果见图7。分析表明,西伯利亚鲟FBPase氨基酸序列与裸盖鱼(Anoplopoma fimbria)同源性最高,为88.7% ,与人、小鼠、鸡、爪蟾(Xenopus tropicalis)、银鲫(Carassius gibelio)和胡瓜鱼(Osmerus mordax)的同源性分别为79.3% 、73.4%、74.6%、81.7% 、87.2%和 85.5%。
将西伯利亚鲟G6Pase氨基酸序列与其他物种的G6Pase氨基酸序列进行同源性比对,结果见图8。分析表明,西伯利亚鲟G6Pase氨基酸序列与翘嘴红鲌同源性最高,为72.7%,与人、小鼠、猫(Felis catus)、猪(Sus scrofa)、火鸡(Meleagris gallopavo)、绿变色蜥(Anolis carolinensis)、胡瓜鱼和花鲈(Lateolabrax japonicus)的同源性分别为67.8% 、67.2%、68.1%、68.6% 、70.4%、72.3%、71.1% 和 71.8%。
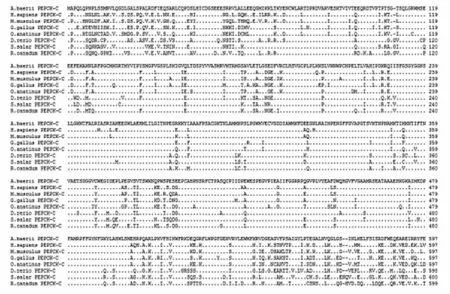
图5 西伯利亚鲟与其他物种PEPCK-C氨基酸序列比对结果Fig.5 Alignment results of PEPCK-C amino acid sequences between Acipenser baerii and other species
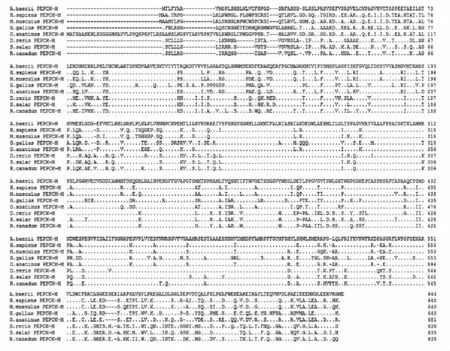
图6 西伯利亚鲟与其他物种PEPCK-M氨基酸序列比对结果Fig.6 Alignment results of PEPCK-M amino acid sequences between Acipenser baerii and other species

图7 西伯利亚鲟与其他物种FBPase氨基酸序列比对结果Fig.7 Alignment results of FBPase amino acid sequences between Acipenser baerii and other species
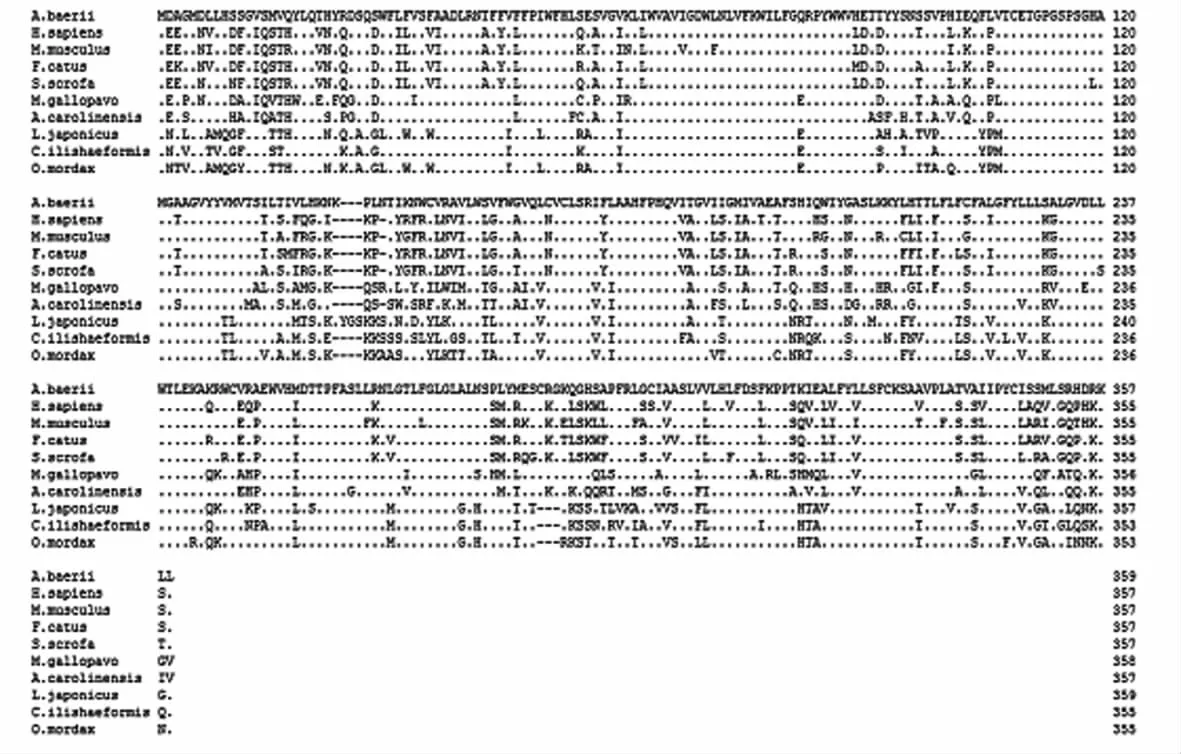
图8 西伯利亚鲟与其他物种G6Pase氨基酸序列比对结果Fig.8 Alignment results of G6Pase amino acid sequences between Acipenser baerii and other species
2.3 系统进化树分析
PEPCK氨基酸序列系统进化树结果见图9。如图所示,西伯利亚鲟PEPCK-C与其他鱼类的PEPCK-C以bootstrap值为73的支持度形成了一个分支,再以bootstrap值为100的支持度与其他陆生脊椎动物的PEPCK-C合并为一个PEPCK-C分支;西伯利亚鲟PEPCK-M与其他鱼类的PEPCK-M以bootstrap值为95的支持度形成了一个分支,再以bootstrap值为80的支持度与PEPCK-C分支合并;哺乳动物的PEPCK-M单独形成一个分支。
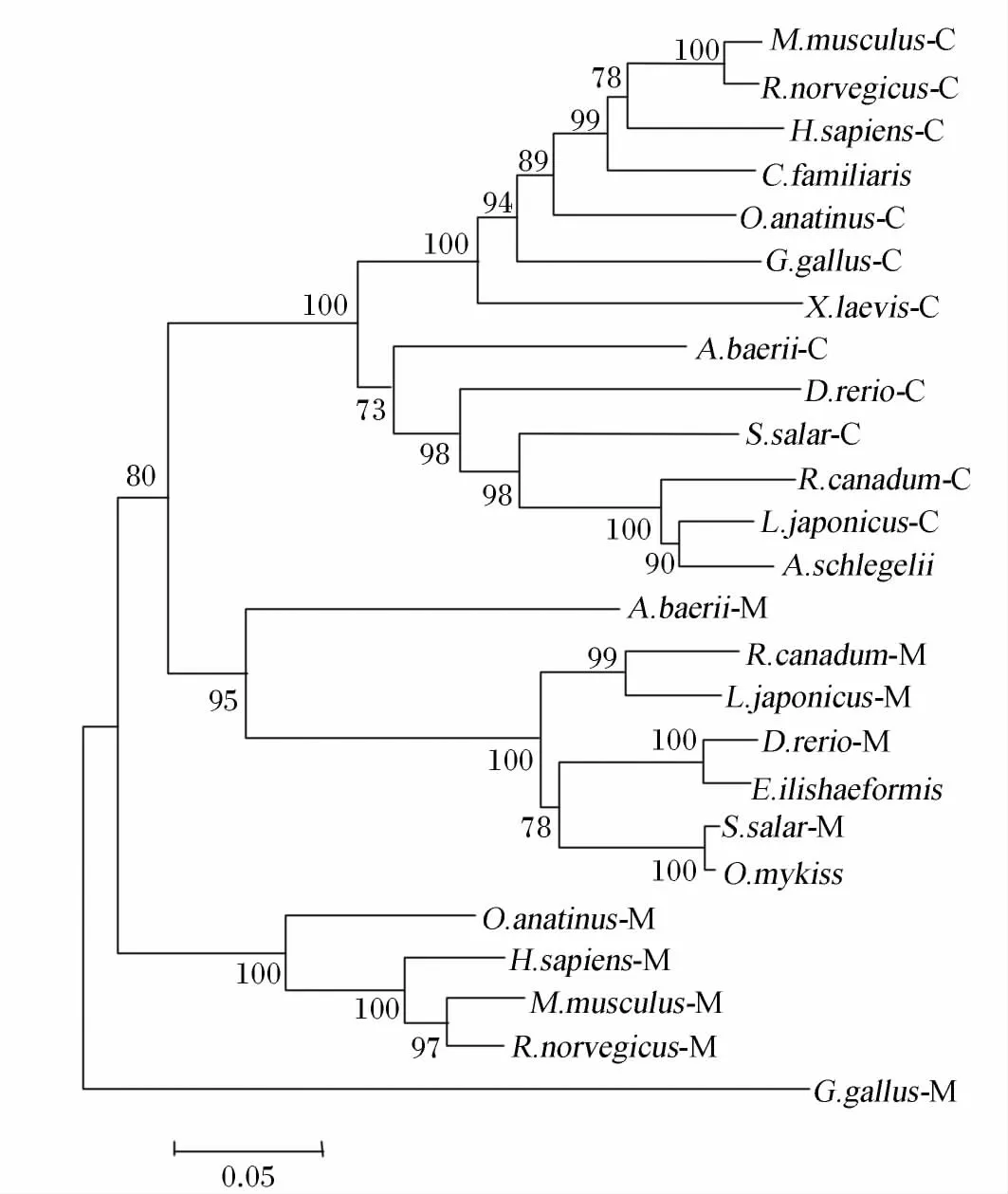
图9 西伯利亚鲟和其他物种PEPCK氨基酸序列系统进化树聚类分析Fig.9 Cluster analysis of phylogenetic tree of PEPCK amino acid sequence between Acipenser baerii and other species
FBPase氨基酸序列系统进化树结果见图10。如图所示,西伯利亚鲟FBPase以bootstrap值为62的支持度与裸盖鱼和胡瓜鱼合并为一支,然后与银鲫和斑点叉尾(Ictalurus punctatus)合并为鱼类分支,再与爪蟾合并为一支;乌梢蛇(Zaocys dhumnades)与鸡合并后与其他哺乳动物合并为另外一支。
G6Pase氨基酸序列系统进化树结果见图11。如图所示,西伯利亚鲟G6Pase以bootstrap值为99的支持度与其他鱼类合并成为一个分支;绿变色蜥和火鸡合并后,与哺乳动物共同形成另一个分支。
3 讨论
PEPCK是催化糖异生作用第1步的关键酶,依赖GTP将草酰乙酸盐转变成磷酸烯醇式丙酮酸盐。PEPCK在动物体内存在2种形式,即细胞溶质来源的PEPCK-C和线粒体来源的PEPCK-M,二者具有相似的动力学特性和分子质量大小,但编码的基因不同[22]。在本研究中,我们成功克隆到西伯利亚鲟2条编码PEPCK基因的全长cDNA序列,通过序列比对分析发现这2条序列间具有很高的相似性(71.2%),所编码的氨基酸序列都具有PEPCK特有的和草酰乙酸结合的结构域以及与GTP三磷酸链和Mg2+结合的激酶1和激酶2基序(图1和图2)。通过多序列比对发现,西伯利亚鲟PEPCK-C氨基酸序列与其他鱼类的相似性大于78.0%,与其他脊椎动物的相似性也均大于77%;PEPCK-M氨基酸序列与其他鱼类的相似性大于72%,与其他脊椎动物的相似性也均大于64%,说明其在进化过程中具有较高的保守性。系统进化树分析结果显示,西伯利亚鲟PEPCK-C和PEPCK-M分别与鱼类的PEPCK-C和PEPCK-M相聚合,在进化上具有较近的亲缘关系。有趣的是,鱼类的PEPCK-M聚合的分支并没有与哺乳动物的PEPCK-M分支聚合,而是首先与PEPCK-C分支聚合(图9),说明在进化上鱼类的PEPCK-M和PEPCK-C间的亲缘关系更近,相似度更高,而哺乳动物的则较远。这可能是由于随着物种由低等向高等进化,基因的分化和变异程度更高所致。
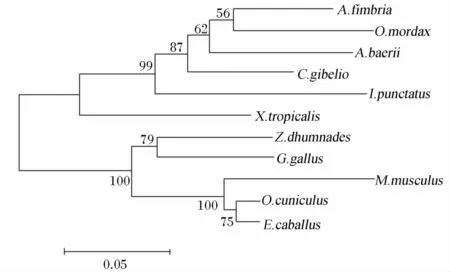
图10 西伯利亚鲟和其他物种FBPase氨基酸序列系统进化树聚类分析Fig.10 Cluster analysis of phylogenetic tree of FBPase amino acid sequence between Acipenser baerii and other species
2种来源的PEPCK活性在动物肝脏细胞中的比例是有物种特异性的,如在鼠中PEPCK-C来源的活性几乎为总活性的100%,而在鸟类中则是PEPCK-M来源的活性占100%。PEPCK的活性主要受基因表达转录调控,PEPCK-C基因受营养状况和激素的调控,被认为是糖异生作用的限速酶[22-23];而线粒体来源的 PEPCK-M 基因为构成性表达,不受营养状况和激素的调控。在鱼类的研究中,对虹鳟的研究证明其PEPCK活性主要来源为 PEPCK-M(>80%)[24],并且其基因一直高表达而与营养状况无关[25]。从鲤鱼和真鲷中克隆到的PEPCK cDNA部分序列由于5'末端的缺失而无法确定其来源,在营养状况对其表达量影响的研究中发现,鲤鱼摄食后24 h的PEPCE基因的表达显著高于摄食后6 h,但真鲷PEPCK基因的表达却不受摄食时间及碳水化合物种类的影响,一直维持在相似的水平[8]。该研究结果可能与肉食性和杂食性鱼类本身的差异有关,但也可能是由于未能区分其PEPCK基因的来源所致。因此,在研究营养状况对鱼类PEPCK活性及其基因表达调控时,首先必须确定其在研究物种中的分配比例,确定编码PEPCK-C的基因,如不加以区分的话则可能由于PEPCK-M比例较高而掩盖结果[4]。本研究通过全长序列的克隆、同源性分析和系统进化树构建,成功的区分出西伯利亚鲟2种来源的PEPCK基因,为今后进一步研究营养状况对其表达的影响奠定了基础。
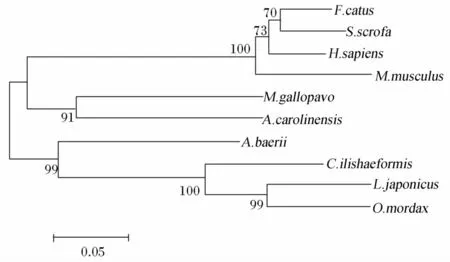
图11 西伯利亚鲟和其他物种G6Pase氨基酸序列系统进化树聚类分析Fig.11 Cluster analysis of phylogenetic tree of G6Pase amino acid sequence between Acipenser baerii and other species
FBPase依赖Mg2+催化果糖-1,6-二磷酸盐水解成为果糖-6-磷酸盐。在哺乳动物中,FBPase存在2种形式,主要在肝脏中表达的FBPase1和主要在肌肉中表达的FBPase2,二者具有相似的功能,但编码的基因不同[26]。在鱼类的研究主要集中在肝脏来源的FBPase上,多个鱼类肝脏中FBPase基因全长或部分cDNA序列被克隆,如虹鳟[27]、鲤鱼和真鲷[8]等,目前尚未见肌肉来源的FBPase的研究报道。本研究成功克隆出西伯利亚鲟肝脏中的FBPase编码基因全长cDNA序列,通过多序列比对发现西伯利亚鲟FBPase氨基酸序列与其他鱼类的相似性大于85%,与其他脊椎动物的相似性也均大于73%,说明其在进化中具有较高的保守性。系统进化树分析结果也显示其与鱼类的亲缘关系更近。在鱼类中,多个研究证实包 括 大 西 洋 鲑[6]、鲤 鱼[8]、真 鲷[9-10]和 欧 洲鲈[11-12]饲料中的碳水化合物对肝脏中 FBPase基因的表达及其酶的活性均没有明显的抑制作用,以上研究结果与在哺乳动物中的研究结果相反。
G6Pase催化糖异生作用的最后一步,由葡萄糖-6-磷酸盐水解脱磷酸形成葡萄糖。本研究克隆到的西伯利亚鲟G6Pase编码基因全长cDNA序列所编码的氨基酸序列具有G6Pase家族特有的PAP2蛋白质结构域,其中有3个保守基序(KXXXXXXRP、PSGH、SRXXXXXHXXXQ/D)(图4)。通过多序列比对发现西伯利亚鲟G6Pase氨基酸序列与其他鱼类的相似性很高(>71.1%),系统进化树分析也显示其与鱼类的亲缘关系更近。但在比对中也发现,其与其他某些鱼类,如斑马鱼、真鲷和欧鲈的相似性仅为59.2%、55.3%和56.9%,甚至不如与哺乳动物的相似性(>67.2%),这证明在鱼类中可能存在不同的G6Pase类型,唐永凯等[28]在对克隆到的翘嘴红鲌的G6Pase编码全长cDNA序列进行比对过程中也发现了类似的现象。但目前在鱼类上并没有同一物种同时存在不同类型G6Pase的报道,其结构和功能上的差异以及存在不同类型的G6Pase对鱼类有何特殊的意义需要进一步的研究加以确认。在鱼类的研究中显示,其肝脏中G6Pase的表达调控与食性存在密切联系,杂食性的鲤鱼摄食含淀粉、葡萄糖、果糖的饲料后G6Pase活性受到明显抑制[13];而肉食性的虹鳟[5]、真鲷[7-8]和欧鲈[11]肝脏中G6Pase表达(mRNA和活性)则不受饲料碳水化合物水平以及餐后时间的影响。
在哺乳动物中,糖异生途径关键酶的活性和基因表达会被饥饿所诱导并被摄食富含碳水化合物的饲料所抑制,其表达主要在转录水平受到相应激素如胰岛素、胰高血糖素和糖皮质激素等的调控。在PEPCK和G6Pase基因的启动子区域存在多个响应元件,如胰岛素和糖皮质激素受体以及cAMP和甲状腺激素响应元件等[29-30]。在不同的营养状况下,激素水平的变化影响多种转录因子的表达,转录因子又与启动子上的响应元件结合,刺激或抑制糖异生途径关键酶的转录表达[31]。而鱼类,尤其是肉食性鱼类,却缺乏外源碳水化合物对糖异生途径关键酶表达的调控,其糖异生作用一直保持在较高水平,这可能是导致肉食性鱼类餐后持续高血糖,无法较好地利用饲料中碳水化合物的重要原因[4]。但目前并没有有关鱼类糖异生途径转录调控的研究报道,肉食性鱼类和杂食性及草食性鱼类的糖异生关键酶基因在cDNA结构上没有明显的差异,可能在转录调控水平对营养状况存在不同的响应机理,需要进一步的研究加以确定。在FishBase中西伯利亚鲟虽然被归为肉食性鱼类,但相比典型的肉食性硬骨鱼类却能较好的利用饲料中的碳水化合物[14-15],因此,其可能存在与典型肉食性硬骨鱼类不同的对糖异生途径的调控机制,本试验成功克隆到西伯利亚鲟糖异生途径上全部关键酶的编码基因全长cDNA序列,为进一步研究其调控机理奠定了基础。
4 结论
①本研究成功克隆到西伯利亚鲟肝脏中糖异生途径关键酶 PEPCK-C、PEPCK-M、FBPase和G6Pase的编码基因全长cDNA序列,经多序列比对发现其与其他已知物种的相应基因具有很高的相似性。
② 系统进化树分析显示,西伯利亚鲟PEPCK-C、PEPCK-M、FBPase和 G6Pase均与鱼类的亲缘关系较近,但G6Pase在鱼类中可能存在不同的类型,其结构和功能上的差异以及存在不同类型的G6Pase对鱼类有何特殊的意义需要进一步的研究加以确认。
[1] WILSON R P.Utilization of dietary carbohydrate by fish[J].Aquaculture,1994,124:67 -80.
[2] MOON T W.Glucose intolerance in teleost fish:face or fiction?[J]Comparative Biochemistry and Physiology,2001,129B:243 -249.
[3] PANSERAT S,CAPILLA E,GUTIERREZ J,et al.Glucokinase is highly induced and glucose-6-phosphatase poorly repressed in liver of rainbow trout(Oncorhynchus mykiss)by a single meal with glucose[J].Comparative Biochemistry and Physiology,2001,128B:275 -283.
[4] ENES P,PANSERAT S,KAUSHIK S,et al.Nutritional regulation of hepatic glucose metabolism in fish[J].Fish Physiology and Biochemistry,2009,35:519-539.
[5] PANSERAT S,MEDALE F,BREQUE J,et al.Lack of significant long-term effect of dietary carbohydrates on hepatic glucose-6-phosphatase expression in rainbow trout(Oncorhynchus mykiss)[J].The Journal of Nutrition Biochemistry,2000,11:22 -29.
[6] TRANULIS M A,DREGNI O,CHRISTOPHERSEN B,et al.A glucokinase-like enzyme in the liver of Atlantic salmon(Salmo salar)[J].Comparative Biochemistry and Physiology,1996,114B:35 -39.
[7] CASERAS A,METON I,VIVES C,et al.Nutritional regulation of glucose-6-phosphatase gene expression in liver of the gilthead sea bream(Sparus aurata)[J].British Journal of Nutrition,2002,88:607 -614.
[8] PANSERAT S,PLAGNES-JUAN E,KAUSHIK S.Gluconeogenic enzyme gene expression is decreased by dietary carbohydrates in common carp(Cyprinus carpio)and gilthead seabream(Sparus aurata)[J].Biochimica et Biophysica Acta,2002,1579:35 -42.
[9] FERNANDEZ F,MIQUEL A G,CORDOBA M,et al.Effects of diets with distinct protein-to-carbohydrate ratios on nutrient digestibility,growth performance,body composition and liver intermediary enzyme ac-tivities in gilthead sea bream(Sparus aurata,L.)fingerlings[J].Journal of Experimental Marine Biology and Ecology,2007,343:1 -10.
[10] ENES P,PANSERAT S,KAUSHIK S,et al.Growth performance and metabolic utilization of diets with native and waxy maize starch by gilthead sea bream(Sparus aurata)juveniles[J].Aquaculture,2008,274:101-108.
[11] ENES P,PANSERAT S,KAUSHIK S,et al.Effect of normal and waxy maize starch on growth,food utilization and hepatic glucose metabolism in European sea bass(Dicentrarchus labrax)juveniles[J].Comparative Biochemistry and Physiology,2006,143A:89 -96.
[12] MOREIRA I S,PERESH,COUTO A,et al.Temperature and dietary carbohydrate levels effects on performance and metabolic utilisation of diets in European sea bass(Dicentrarchus labrax)juveniles[J].Aquaculture,2008,274:153 -160.
[13] SHIKATA T,IWANAGA S,SHIMENO S.Effects of dietary glucose,fructose,and galactose on hepatopancreatic enzyme activities and body composition in carp[J].Fisheries Science,1994,60:613 -617.
[14] 盛洪建.理想蛋白模式下西伯利亚鲟日粮中混合蛋白替代鱼粉的研究[D].硕士学位论文.北京:中国农业科学院,2009.
[15] 吴秀峰,薛敏,郭利亚,等.脱酚棉籽粉替代部分鱼粉对西伯利亚鲟幼鱼生长、体成分及血清生化指标的影响[J].动物营养学报,2010,22(1):117 -124.
[16] 罗琳,薛敏,吴秀峰,等.脱酚棉籽蛋白对日本鲈的生长、体成分及营养成分表观消化率的影响[J].水产学报,2005,29(6):866 -870.
[17] LUO L,XUE M,WU X F,et al.Partially or totally replacement of fishmeal by solvent-extracted cottonseed meal in diets for juvenile rainbow trout(Oncorhynchus mykiss)[J].Aquaculture Nutrition,2006,12:418-424.
[18] REIGH R C,ELLIS S C.Effects of dietary soybean and fish protein ratios on growth and body composition of red drum(Sciaenops ocellatus)fed isonitrogenous diets[J].Aquaculture,1992,104:279 - 292.
[19] SULLIVAN J A,REIGH R C.Apparent digestibility of selected feedstuffs in diets for hybrid striped bass(Morone sanatilin × Morone chrysops)[J].Aquaculture,1995,138:313 -322.
[20] ROSE T M,SCHULTZ E R,HENIKOFF J G,et al.Consensus-degenerate hybrid oligonucleotide primers for amplification of distantlyrelated sequences[J].Nucleic Acids Research,1998,26(7):1628 -1635.
[21] KUMAR S,TAMURA K,JAKOBSEN I B,et al.MEGA2:molecular evolutionary genetics analysis software[J].Bioinformatics,2001,17:1244 - 1245.
[22] HANSON R W,RESHEF L.Regulation of phosphoenolpyruvate carboxykinase(GTP)gene expression[J].Annual Review of Biochemistry,1997,66:581-611.
[23] CHAKRAVARTY K,CASSUTO H,RESHEF L,et al.Factors that control the tissue-specific transcription of the gene for phosphoenolpyruvate carboxykinase-C[J].Critical Reviews in Biochemistry and Molecular Biology,2005,40:129 -154.
[24] WALTON M J,COWEY C B.Gluconeogenesis from serine in rainbow trout liver[J].Comparative Biochemistry and Physiology,1979,62B:497 -499.
[25] PANSERAT S,PLAGNES-JUAN E,BREQUE J,et al.Hepatic phosphoenolpyruvate carboxykinase gene expression is not repressed by dietary carbohydrates in rainbow trout(Oncorhynchus mykiss)[J].Journal of Experimental Biology,2001,204:359 -365.
[26] STEIN S,LIEHR T,ESCHRICH K.Characterization of the mouse liver fructose-1,6-bisphosphatase gene[J].Gene,2001,264:215 -224.
[27] PANSERAT S,PLAGNES-JUAN E,KAUSHIK S.Nutritional regulation and tissue specificity of gene expression for proteins involved in hepatic glucose metabolism in rainbow trout(Oncorhynchus mykiss)[J].Journal of Experimental,2001,204:2351 -2360.
[28] 唐永凯,俞菊华,刘波,等.翘嘴红鲌肝脏G6Pase催化亚基的克隆以及摄食和饲料中碳水化合物对其表达的影响[J].水产学报,2007(3):45-53.
[29] YANG J,KALHAN SC,HANSON R W.What is the metabolic role of phosphoenolpyruvate carboxykinase?[J]Journal of Biological Chemistry,2009,284:27025-27029.
[30] YANG J,RESHEF L,CASSUTO H A,et al.Aspects of the control of phosphoenolpyruvate carboxykinase gene transcription[J].Journal of Biological Chemistry,2009,284:27031 -27035.
[31] JITRAPAKDEE S.Transcription factors and coactivators controlling nutrient and hormonal regulation of hepatic gluconeogenesis[J].The International Journal of Biochemistry and Cell Biology,2012,44:33 -45.
