猪流行性腹泻病毒P55野生毒株主要结构蛋白遗传分析
2013-12-18高杏丹陈曦杨金先俞伏松李英英林天龙宋铁英
高杏丹陈 曦杨金先俞伏松李英英林天龙宋铁英*
(1.福建师范大学生命科学学院 福州 350108;2.福建省农业科学院生物技术研究所 福州 350003)
猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV),是一种引发猪消化道黏膜发炎和糜烂等流行性腹泻病症的病毒。其临床特征表现为发热、厌食、呕吐、腹泻以及脱水等,且各种年龄和不同品种的猪群都有易感性,尤其对哺乳仔猪危害最严重,死亡率高。PED于1971年在英国首次被报道[1],此后日本、韩国及泰国等多个国家都报道了PEDV的发生[2-4]。PEDV基因组是单股正链具有感染性的RNA,全长27 000~33 000个核苷酸,编码非结构蛋白(复制酶多聚蛋白1ab,ORF3蛋白)和4种主要结构蛋白。结构蛋白从5’→3’端依次为:纤突蛋白(Spike,S)、小膜蛋白(sma11Membrane,sM)、膜蛋白(Membrane,M)和核衣壳蛋白(Nuc1eocapsid,N)。
近年来,PED在我国呈现了不断扩散和暴发的态势[5-7]。特别是2010年以来,在全国包括福建地区均出现了大面积的暴发,给养猪业发展造成沉重的打击[8]。2010-2012年,本研究室对福建省内20多个猪场进行了分子流行病学调查,筛选到一株标号为P55的野生毒株,其在与病毒毒力相关的ORF3区域的412到465位置出现了与CH/GSJIII/07(GeneBank:GU372743)相似的大片段缺失[9]。为研究P55片段中是否存在有其他影响病毒毒力的变异,需要对该株的其余结构基因展开研究。本文以野生变异毒株P55株为研究对象,通过对M蛋白和N蛋白的核苷酸和氨基酸序列与参考毒株的比较分析,同时参考先期研究中的S1和ORF3蛋白的结果[10],充分评估该毒株的变异情况,为进一步的功能性研究奠定基础。
1 材料与方法
1.1 病料收集与处理 病料样品采自福建省某猪场内表现急性肠炎和水样腹泻仔猪的部分肠组织。样品采集后,置于研钵研磨,加入pH7.4的 PBS制成1:9的组织匀浆,在涡旋仪上振荡混匀,1 700×g离心10 min,吸取上清液,-80℃冻存备用。
1.2 引物的设计与合成 参照GenBank中己发表的PEDV CV777 M基因和N基因序列,设计特异性扩增引物,该引物由宝生物大连(TaKaRa)有限公司合成。见表1。
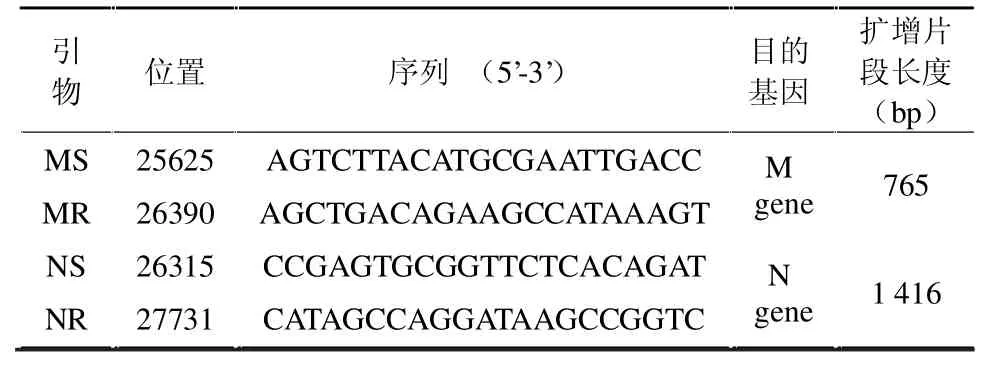
表1 M与N基因对应的特异性引物
1.3 总RNA的提取 取200 uL样品上清液,加入800 uL RNAiso P1us Reagen(t宝生物工程有限公司)振荡混匀,室温静置5 min后,使用氯仿和异丙醇进行抽提、离心,所得沉淀用乙醇洗涤,自然干燥后加入RNase-free水溶解沉淀,即为所提取RNA模板,-40℃冻存备用。
1.4 PEDV M、N基因的扩增 使用PrimescriptOne Step RT-PCR Kit Ver.2(宝生物工程有限公司)进行基因扩增。反应总体系为25 uL:包含PrimescriptOne Step Enzyme Mix 1 uL、2×1 Step Buffer 12.5 uL、上游引物和下游引物(10 pmo1/uL)各 1 uL、RNA模板0.5 uL、双蒸水9 uL。反应条件为:50℃反转录30 min,94℃预变性2 min,94℃变性30 s、50℃退火30 s、72℃延伸1 min,30个循环。PCR产物进行1.5%琼脂糖凝胶电泳分析,确认结果后产物送交Takara公司测序。
1.5 序列比对及进化树分析 将所得P55毒株的M与N基因序列与选择的GeneBank中的参考序列,置入MegA1ign4.0(DNA Star)程序中,采用C1usta1W方法加以比较,得到各毒株间的遗传距离,以此为基础绘制其遗传系统进化树,结合前期研究结果中S1和ORF3区的进化树,进行P55与各毒株间的遗传距离计算和分析。
2 结果与分析
2.1 PEDV目的片段的RT-PCR鉴定 采用两组特异性引物(见表1)扩增所得的RT-PCR结果见图1,对应的目的片段M、N的长度分别为746 bp和1 464 bp。
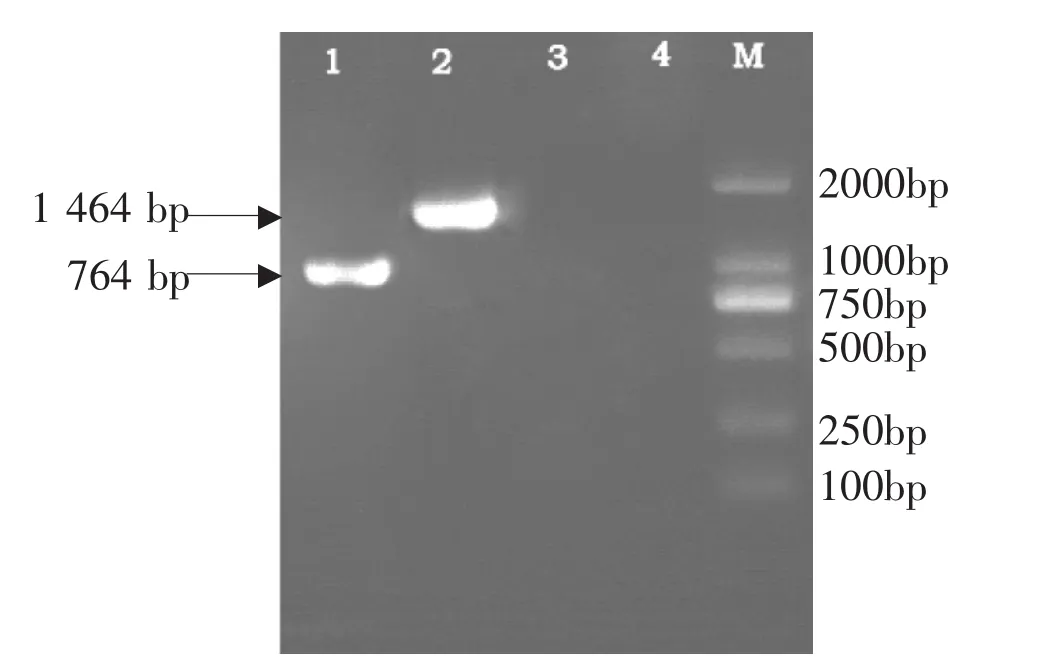
图1 PEDV目的片段的RT-PCR扩增
2.2 M基因的分析 试验株P55获得的M基因由746个核苷酸组成,(G+C)含量为45.08%,编码226个氨基酸,蛋白分子量约为25.38 kD,GenBank登录号为JQ723736。选取与P55同源性较高(Max score>1 210)的20株毒株为参考进行类比。比较结果显示,P55毒株M基因序列无特有的片段缺失和插入,也未出现特有的碱基点突变。此外,在P55M基因序列的启动子上游11个核苷酸的位置,存在有高度保守的转录调控因子(TRSs),其核心区由6个核苷酸组成(5’-ATAAAC-3’)[11]。
采用MegA1ign4.0(DNA Star)软件得到的氨基酸进化分析结果显示:M蛋白总变异度为2.4。氨基酸序列分析结果未发现P55存在特有突变,其具有的潜在的天冬酰胺酸糖基化结合位点的数量(3个),与经典株CV777一致。在氨基酸进化树分组中P55与从中国南方的广东、湖北、上海分离的5株病毒(CHGD-01、GD-A、AJ1102、ZJCZ4、LC) 遗传距离较近。见图2。
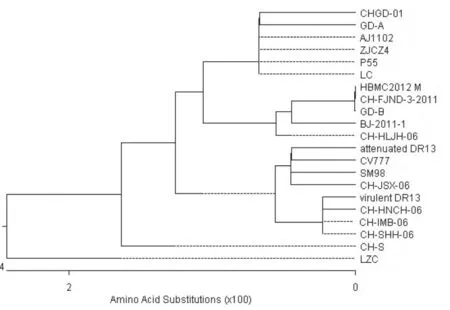
图2 P55株M基因遗传进化树
2.3 N基因的分析 试验株P55获得的N基因由1 464个核苷酸组成,(G+C)含量为47.51%,编码有441个氨基酸,蛋白分子量约为48.95 kD,GenBank登录号为JQ723731。从GeneBank中选取选取与P55同源性较高(Max score>2 300)的 22株毒株为参考进行类比。结果显示,P55毒株N基因序列无特有的片段缺失和插入,但出现了4个该株特有的点突变,影响到3个氨基酸的翻译,位点见表2。这些变异并未改变其在启动子上游9个核苷酸处存在的转录调控因子,该因子的核心区为 5’-AGAAAC-3’[11]。
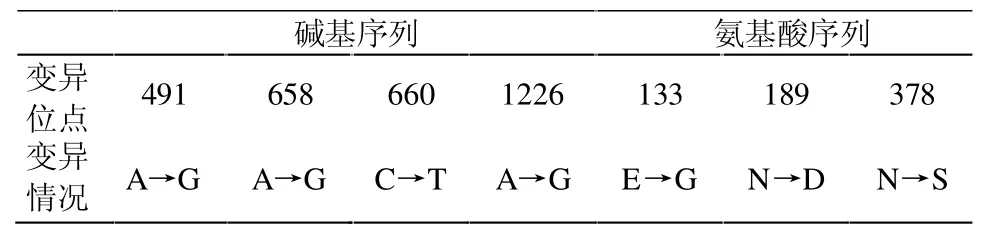
表2 P55株N蛋白变异情况
同样采用MegA1ign4.0(DNA Star)软件得到的氨基酸进化分析结果显示,N蛋白总变异度为3.7。此外,P55毒株N蛋白的氨基酸序列存在6个潜在的天冬酰胺酸糖基化结合位点,与CV777毒株的N蛋白一致。在氨基酸进化树分组中P55与CHGDS-09、CH-IMT-06、CH-S、attenuated DR13 遗传距离接近,其中attenuated DR13来源于韩国,其余毒株均从中国分离(见表3、图3)。
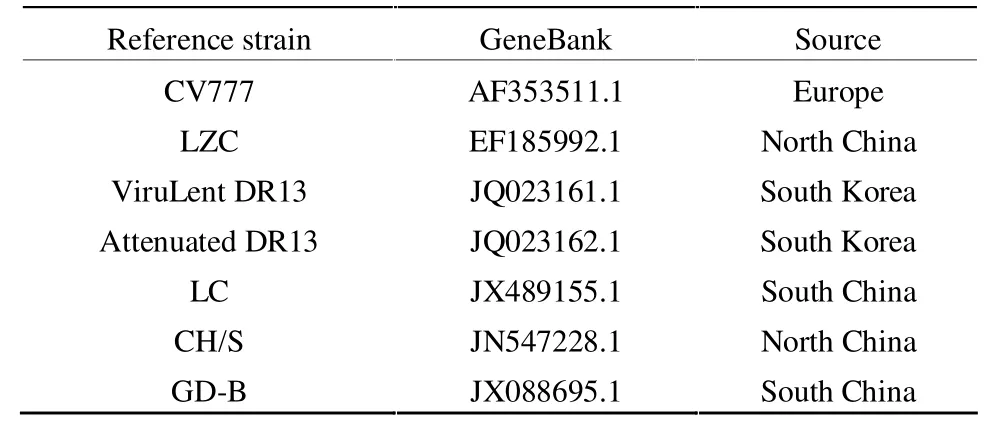
表3 具有全基因组序列的PEDV参考株
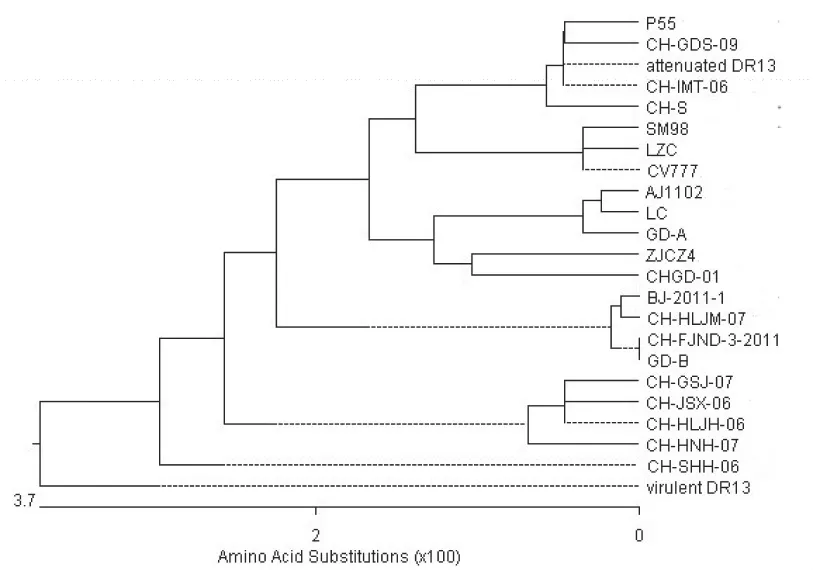
图3 P55株N基因遗传进化树
2.4 M、N、S1、ORF3蛋白的遗传距离分析 抽选在P55毒株的4组蛋白比较中共有的参考毒株进行分析,并对各组值进行方差分析(见表4)。结果显示,遗传距离:S1蛋白>ORF3蛋白>N蛋白>M蛋白。
3 讨论
1)20世纪90年代以来,虽然各种灭活疫苗和弱毒疫苗已经被应用于PED的防控,但其在中国的流行性暴发仍时有发生,特别是2010年以来PED大规模暴发对我省养猪业造成的重大的经济损失[8,12],警示我们有必要对PEDV毒株的变异情况进行调查。
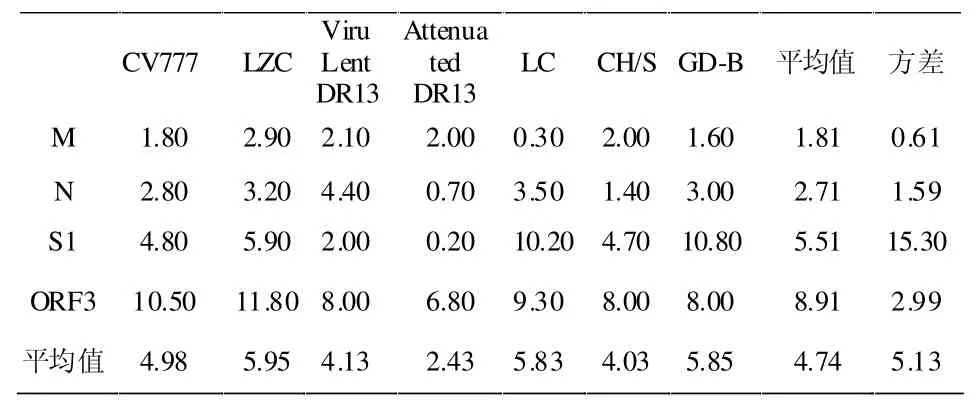
表4 P55株与参考株M、N、S1、ORF3蛋白的遗传距离
2)本研究中,P55的M蛋白未发现特殊变异,进化树变异度和遗传距离的方差结果显示,M蛋白在主要结构蛋白中是最为保守的。由于M蛋白在病毒粒子的组装和出芽过程中具有重要作用[13],且能介导机体产生干扰素[14],因此,研究结果再次证明可以将M基因作为PEDV基因工程疫苗的候选基因。另外,在进化树中,P55所在分支的所有毒株均来自于中国,推测在M蛋白区域,P55表现出一定的地域保守性。
3)相较M蛋白,P55的N蛋白变异度增加,其N蛋白基因的ORF均由1326 nt组成,无核苷酸缺失和插入现象,但是碱基序列出现了4个点突变,从而影响了3个氨基酸的变异,由于N蛋白参与病毒的复制和转录[15],该变异是否会影响其功能尚待研究。
4)将前期结果中P55毒株的S1和ORF3蛋白与本研究的M、N蛋白遗传距离的方差分析结果进行比较,由于方差表现的是各毒株与P55之间遗传距离的变化程度,该结果揭示了各蛋白保守程度的差异,M和N蛋白的变异度较S1和ORF3小[10]。同时,P55与各毒株之间的平均遗传距离结果表明,P55毒株和来源于韩国的attenuated DR13亲缘关系较近。
5)Song等证明,ORF3区域的缺失或变异会导致病毒毒力的改变[16]。但近期的相关研究中陆续报道了4株与P55的ORF3区域存在类似突变的野生毒株,其中3株分离自中国,说明类似的缺失变异毒株在我国出现并已开始流行扩散[9,17](CH/BJ/2011 GenBank:JQ027019;Zhejiang-08 GenBank:JX0027 03)。因此,研究病毒毒力的分子机制对预防和控制该类变异毒株至关重要。对P55的M和N蛋白区域基因序列与参考序列进行比较,未发现特殊的缺失和变异。综上所述,P55毒株中是否存在有除了M、N、S1之外的其他影响病毒毒力因素,尚待进一步对病毒的如S2蛋白区和PPo1区等进行分析研究。
[1] OLDHAMJ.Pig Farming(October Supp1)[M].1972:72-73.
[2] TAKAHASHI K,OKADA K,OHSHIMA K.An outbreak of swine diarrhea of a new-type associated with coronavirus-1ike partic1es in Japan [J].Nihon Juigaku Zasshi,1983,45(6):829-832.
[3] PURANAVEJA S,POOLPERM P,LERTWATCHARASARAKUL P,et a1.Chinese-1ike strain of porcine epidemic diarrhea virus,Thai1and [J].Emerg Infect Dis,2009,15(7):1112-1115.
[4] PARK SJ,MOON H J,YANG J S,et a1.Sequence ana1ysis of the partia1 spike g1ycoprotein gene of porcine epidemic diarrhea viruses iso1ated in Korea [J].Virus Genes,2007,35(2):321-332.
[5] 刘畅.目前猪传染性腹泻疾病的流行情况与防控方案[J].今日畜牧兽医,2012(8):15-16.
[6] 李思银,杨亮宇,杨玉艾.猪流行性腹泻的实验室诊断方法[J].猪业科学,2010(12):54-57.
[7] 林忠武.猪流行性腹泻的流行特点及其防治措施[J].福建畜牧兽医,2011,33(4):14-15.
[8] 张世忠,江斌.2011年福建省猪流行性腹泻的流行特点及其防治措施[J].福建畜牧兽医,2012,34(2):23-25.
[9] CHEN J,WANG C,SHI H,et a1.Mo1ecuLar epidemio1ogy of porcine epidemic diarrhea virus in China.[J].Arch Viro1,2010,155(9):1471-1476.
[10] Chen X,Yang J,Yu F,et a1.Mo1ecuLar characterization and phy1ogenetic ana1ysis of porcine epidemic diarrhea virus(PEDV)samp1es from fie1d cases in Fujian,China[J].Virus Genes,2012,45(3):499-507.
[11] PENZES Z,GONZALEZ J M,CALVO E,et a1.Comp1ete genome sequence of transmissib1e gastroenteritis coronavirus PUR46-MAD c1one and evo1ution of the purdue virus c1uster[J].Virus Genes,2001,23(1):105-118.
[12] 徐国栋,李峰,张广峰.国内猪流行性腹泻防治概况[J].畜牧与兽医,2011,43(12):88-93.
[13] DE HAAN C A,KUO L,MASTERS P S,et a1.Coronavirus partic1e assemb1y:primary structure requirements of the membrane protein [J].J Viro1,1998,72(8):6838-6850.
[14] LAUDE H,GELFI J,LAVENANT L,et a1.Sing1e amino acid changes in the vira1g1ycoprotein M affect induction of a1pha interferon by the coronavirus transmissib1e gastroenteritis virus[J].JViro1,1992,66(2):743-749.
[15] Jian-Qiang Li,Ji-Xing Liu,Xi Lan,et a1.C1oning the Structure Genes and Expression the N Gene of Porcine Epidemic Diarrhea Virus DX[J].中国病毒学:英文版,2009,24(3):179-186.
[16] SONG D S,YANG J S,OH J S,et a1.Differentiation of a Vero ce11 adapted porcine epidemic diarrhea virus from Korean fie1d strains by restriction fragment 1ength po1ymorphism ana1ysis of ORF3 [J].Vaccine,2003,21(17/18):1833-1842.
[17] PARK SJ,KIM H K,SONG D S,et a1.Mo1ecuLar characterization and phy1ogenetic ana1ysis of porcine epidemic diarrhea virus(PEDV)fie1d iso1ates in Korea[J].Arch Viro1,2011,156(4):577-585.
