利用肠杆菌基因间重复序列PCR(ERIC-PCR)方法对阪崎克罗诺杆菌进行分子分型
2013-12-10陈万义艾连中穆海波
陈万义,艾连中,任 婧,穆海波
(光明乳业股份有限公司 光明乳业研究院,乳业生物技术国家重点实验室,上海 200436)
0 前言
阪崎克罗诺杆菌是人和动物肠道内寄生的一种革兰阴性无芽孢杆菌.直至1980年,其被鉴定命名为阪崎肠杆菌(Enterobacter sakazakii),归属于肠杆菌科肠杆菌属,并包括15个生物型[1].2008年,Iversen等[2-3]根据16S rRNA基因序列、核糖体分型、荧光扩增片段长度多态性(fluorescent-amplified fragment length polymorphism,f-AFLP) 和 DNADNA杂交试验对阪崎肠杆菌重新进行系统学分类,建议将该菌定义为一个新的属,即克罗诺杆菌属(Cronobacter spp).属内包括 5个新种(其中Cronobacter sakazakii称为新组合)和1个克罗诺杆菌基因种,即阪崎克罗诺杆菌、丙二酸盐阳性克罗诺杆菌、穆汀斯克罗诺杆菌、都柏林克罗诺杆菌、苏黎世克罗诺杆菌、克罗诺杆菌基因种1.
作为一种重要的食源性条件致病菌,阪崎克罗诺杆菌能够引起婴儿致命的感染,致死率高达10%~80%.它通常能导致婴儿严重的临床症状,例如脑脓疡、脑膜炎、坏死性小肠结肠炎和全身性败血症.出生两个月之内的新生婴儿或早产儿都存在通过食用污染有克罗诺杆菌属的婴儿配方粉而感染阪崎克罗诺杆菌的风险[4].2004年我国安徽阜阳劣质婴儿配方粉事件引起了我国政府的高度重视.这也是国内首次从婴儿配方粉中分离到阪崎克罗诺杆菌菌株[5].
虽然在许多临床感染病例中尚不能确定阪崎克罗诺杆菌真正的宿主,同时阪崎克罗诺杆菌也曾在其他食品中检出,而且冲调环境和器皿的污染也是重要的传染源,但越来越多的报道证明婴儿配方粉是主要的传染源和传播媒介,并且只有婴儿配方奶粉(powdered infant formula,PIF)与疾病的暴发相关[6].
目前,研究发现克罗诺杆菌属菌株在自然界中分布广泛,已发现的生存地点为纯净水、土壤、污水、香草和蔬菜,同时动物和人的粪便也是该菌的生存地之一[7].克罗诺杆菌属内6个种对化学物质的敏感性以及温度和抗生素方面存在较大的差异[8],而且各个菌的致病因子在特定情况下的表达也各不相同[9-10],从而导致的致病力大小也不尽相同.因此,克罗诺杆菌属菌株水平上的快速准确的鉴定和分型方法对于控制由该属引起的食源性疾病的监控、预防和控制具有十分重要的意义.同时,对于分子流行病学的研究也是至关重要的.传统的分型方法主要包括:生物型[1]、血清型、质粒分型,它们已经难以满足当今快速溯源技术的要求.越来越多的基因分型方法已经被用于克罗诺杆菌属内菌株的分子分型,包括核糖体分型[11]、随机扩增DNA片段多态性分析(RAPD)[12]、肠杆菌基因间回文重复序列PCR(REP-PCR)[13]、随机片段长度多态性分析(AFLP)[2]和脉冲场凝胶电泳(PFGE)[14].分子分型技术产生的DNA指纹图谱比传统的生物型具有更高的分辨率和可靠性.本研究主要采用肠杆菌间保守重复序列PCR(ERIC-PCR)对43株克罗诺杆菌属菌株和5株肠杆菌属菌株进行分子分型,期望建立一种适合用于我国阪崎克罗诺诺杆菌快速溯源的的分子分型方法.

表1 试验菌株
1 材料与方法
1.1 材料
本次试验共使用了43株克罗诺杆菌属菌株,其中8株为模式菌株(由上海出入境检验检疫局惠赠),剩余35株为食品分离菌株,另外5株为肠杆菌菌株,其中3株为模式菌株(购买于中国微生物菌种保藏中心),2株为分离菌株(见表1).
1.2 试剂及仪器
1.2.1 试剂
溶菌酶、蛋白酶K和RNAseA均为Sigma进口分装;CTAB、SDS、NaCl、苯酚、乙醇、氯仿和异戊醇等均为国产分析纯;琼脂糖为西班牙分装;DNA Marker为北京天根生物科技有限公司.
1.2.2 仪器
PCR 仪:ABI Veriti 96 well thermal cycler,美国生命科技有限公司;核酸测定仪:Thermo Scientific,NANODROP 2000C,基因仪器有限公司;凝胶成像仪:BIO-RAD公司,美国.
1.3 方法
1.3.1 基因组DNA提取及浓度测定
采用CTAB法[15]提取43株克罗诺杆菌属菌株和5株肠杆菌属菌株.将提取好的DNA原液用核酸测定仪进行浓度的测定,并将所有的DNA原液稀释到40 ng/μL用于ERIC-PCR试验.
1.3.2 ERIC-PCR
该试验引用文献[16]报道的一对22 bp的上下游引物,其中上游引物为ERIC 1R(5’-ATG TAA GCT CCT GGG GAT TCA C3’),下游引物为ERIC 2 (5’-AAG TAA GTG ACT GGG GTGAGC G-3’).25μL PCR反应体系中含有2.5μL10×PCR反应缓冲 液 ,2 μL 2.5 mmol/L Mg2+,1 μL 2.5 mmol/L DNTP,1μL含有正向和反向引物(10μmol/L)的混合液,以及2μL的模板DNA.PCR反应条件如下:95℃ 5 min以完全激活 Taq DNA聚合酶,变性:94℃ 45 s;退火:52℃ 1 min;延伸:72℃5min;此过程共进行35个 循环;此后在72℃下继续延伸 10 min.PCR扩增产物在1.5%的琼脂糖凝胶上以恒流80 A、电泳50 min后,溴化乙啶(EB)染色15 min后在凝胶成像仪观察并保存结果(图像格式保存成TIFF格式).
1.3.3 数据数理
ERIC-PCR电泳图谱结果采用数学软件BioN-umericus version 5.0(Applied Maths,Belgium)进行分析,根据非加权算术平均数聚类法(unweightedpair group method using average linkages,UPGMA)进行聚类分析,产生一个相似度矩阵,基于相似度矩阵构建一个最小生成树(分子进化树状图).该基因分型方法的分辨能力的大小计算是根据辛普森多样性方程[17]进行分析的,即根据D值大小判断分辨率的大小,一般情况下D值大于0.9表明分辨率较高,反之,说明分辨率较低.
2 结果与分析
2.1 基因组DNA浓度均一化
使用CTAB法提取的基因组DNA浓度和纯度都较高,而且都稀释到相同的浓度(40 ng/μL)进行3次相同的重复试验,高质量和高纯度的DNA保证了ERIC-PCR结果的准确性和可重复性.
2.2 ERIC-PCR电泳结果分析
对分子流行病学上不相关的43株克罗诺杆菌属菌株和5株肠杆菌属菌株进行ERIC-PCR分型后,都能够扩增出大小及数目不同的条带.从琼脂糖凝胶电泳图谱(图1)上可以明显看出:条带数目为5~10,扩增的片段长度大都在150~4 000 bp之间,而且分子量为1 500 bp,2 000 bp和3 100 bp的扩增片段存在于大多数菌株中.
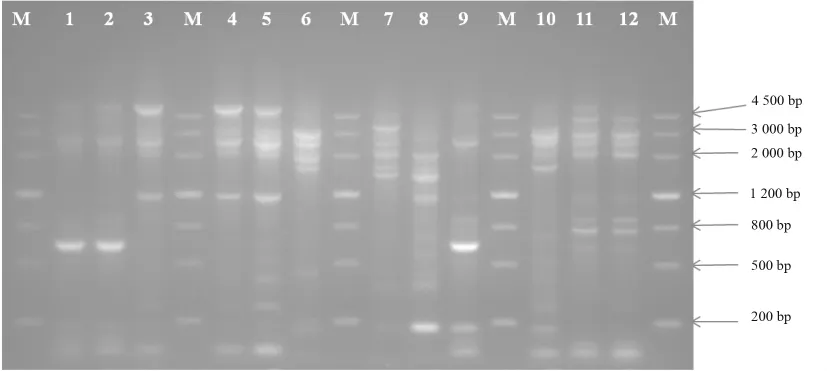
图1 43株克罗诺杆菌属菌株和5株肠杆菌菌株中部位代表菌株的ERIC-PCR电泳图谱
2.3 聚类分析
43株克罗诺杆菌属菌株和5株肠杆菌属菌株的ERIC-PCR电泳图谱经Bionumericus version 5.0软件分析后,根据相似性值大于80%可以将48株菌株分成36个不同的类群(Cluster),详见图 2.根据非加权算术平均数聚类法 (UPGMA)进行聚类分析,构建了所有菌株之间的分子进化树状图谱(图2),从图2可以明显看出,克罗诺杆菌属菌株和肠杆菌属菌株分别聚成一类,表明它们之间的遗传相似性较远.
2.4 分辨率
43株克罗诺杆菌属菌株和5株肠杆菌属菌株的ERIC-PCR电泳图谱经数学软件BioNumerics version 5.0处理后,如果根据相似性值大于0.80,经辛普森多样性方程计算得到ERIC-PCR分型的分辨系数是0.984(D=0.984),这表明ERIC-PCR方法的分辨率相对较高,能够用于我国克罗诺杆菌属菌株分离菌株的亚种分型.
3 结论与讨论
分子分型在食物中毒事件调查中,可鉴别和排除那些与暴发不相关的菌株,精炼暴发数据流行病学分析.目前分子分型的方法有多种,具有不同的适用范围,ERIC-PCR作为其中的一种技术,由于操作简便,分辨率高,在细菌分子流行病学溯源领域已经得到广泛应用[18].

图2 43株克罗诺杆菌属菌株和5株肠杆菌菌株ERIC-PCR电泳图谱经BioNUmericus软件根据UPGMA方法构建的分子进化树状图谱
目前,细菌分子分型中比较常用的分子分型方法是核糖体分型(Ribotyping)和脉冲场凝胶电泳(PFGE)分型.尽管核糖体分型技术已经成功地被引入到多种细菌种间鉴别和分型之中[19].然而,核糖体分型通常产生的条带数目较少,因此分辨率相对较低,而且结果解释起来相对简单一些,并且对某些属的细菌难以将种水平上的差异区别开来.PFGE具有高的分辨率和较好的重复性,一直以来被认为是对病原微生物进行流行病学研究和调查的“金标准”[20],广泛用于对多种细菌进行分型和鉴定.然而PFGE也有自身的缺陷,总有一小部分菌株无法进行PFGE分型,而且有时候这部分菌株所占的比例相对较大(23%)[18],这可能是由于在酶切过程或其他处理步骤中导致DNA的降解,从而无法得到令人满意的结果.ERIC-PCR最早应用于肠杆菌科肠杆菌属细菌的分子分型中,后来发现许多细菌染色体上也存在这种重复序列,因此陆续被引入到其他多种细菌的分型鉴定中[18].由于ERIC-PCR相对PFGE是一种更加快速和操作简便的方法,因此对于大规模菌株的流行病学调查和关键点监控是非常适用的.然而,有时候一些扩增条带较弱,因此会给结果的判读带来一定的困难,并且有可能造成结果难以重复.因此,如果需要更加准确快速和高分辨率的分型方法时,联合其他的方法可能更加有效.对于亲缘关系更加密切的菌株之间的进化关系的分析,则需要结合目前分辨率较高的MLST来进行菌株之间的分型鉴定[21].
通过本次研究证明ERIC-PCR是一种适合用于我国阪崎克罗诺诺杆菌快速溯源的分子分型方法.根据非加权算术平均数聚类法(UPGMA)利用BioNumericus软件进行聚类分析,构建了流行病学无相关性的不同来源的食品分离株和临床致病菌株之间的分子进化树状图谱,根据分子进化树状图谱可以明显看出阪崎克罗诺杆菌不同来源分离菌株之间存在较大的基因多样性.该方法不但可以快速比较阪崎克罗诺杆菌分离菌株之间分子流行病学上的相关性,还可以进一步了解克罗诺杆菌属内不同来源分离菌株之间的分子进化关系.
细菌的分子分型已经广泛用于食源性病原菌流行病学研究和追溯其传播途径的研究之中.由于克罗诺杆菌属内菌株之间在表型上具有高度的一致性,因此传统的分型方法,例如生物学分型、血清学分型或质粒分型[11],已经不适用于其种间多样性差异的鉴别,这就迫切需要发展分辨率更高的基因分型方法用于阪崎克罗诺杆菌种间多样性的区别,及时发现其传染源,切断其传播途径,从而可以防患于未然.
[1] Nazarowec-White M,Farber J M.Enterobacter sakazakii:a review[J].International Journal of Food Microbiology,1997,34:103-113.
[2] Iversen C,Lehner A,Mullane N,et al.The taxonomy of Enterobacter sakazakii:proposal of a new genus Cronobacter gen.nov.and descriptions of Cronobacter sakazakii comb.nov.Cronobacter sakazakii subsp.sakazakii,comb.nov.,Cronobacter sakazakii subsp.malonaticus subsp.nov.,Cronobacter turicensis sp.nov.,Cronobacter muytjensii sp.nov.,Cronobacter dublinensis sp.nov.and Cronobacter genomospecies 1[J].BMC Evolutionary Biology,2007,7:64-74.
[3] Iversen C,Lehner A,MullaneN,etal.Identification of Cronobacter spp. (Enterobacter sakazakii)[J].Journal of Clinical Microbiology,2007,45:3814-3816.
[4] Hunter C J,Petrosyan M,Ford H R,et al.E nterobacter sakazakii:an emerging pathogen in infants and neonates[J].Surgery Infection,2008,9:533-539.
[5] 刘秀梅,裴晓燕,郭云昌.中国安徽阜阳劣质婴儿配方粉中阪崎肠杆菌的污染[J].中国食品卫生杂志,2005,17(1):10-12.
[6] Arseni A,Malamou-Ladas E,Koutsia C,et al.Outbreak of colonization of neonates with Enterobacter sakazakii[J].The Journal of Hospital Infection,1987,9:143-150.
[7] Iversen C,Forsythe S.Risk profile of Enterobacter sakazakii,an emergent pathogen associated with infant milk formula[J].Trends in Food Science&Technology,2003,14:443-454.
[8] Arroyo C,Condon S,Pagan R.Thermobacteriological characterization of Enterobacter sakazakii[J].International Journal of Food Microbiology,2009,136:110-118.
[9] Healy B,Huynh S,Mullane N,et al.Microarraybased comparative genomic indexing of the Cronobacter genus (Enterobacter sakazakii)[J].International Journal of Food Microbiology,2009,136:159-164.
[10] MacLean L L,Pagotto F,Farber J M,et al.The structure of the O-antigen in the endotoxin of the emerging food pathogen Cronobacter(Enterobacter)muytjensii strain 3270[J].Carbohydrate Research,2009,344:667-671.
[11] Iversen C,Mullane N,McCardell B,et al.Cronobacter gen.nov.,a new genus to accom-modate the biogroups of Enterobacter sakazakii,and proposal of Cronobacter sakazakii gen.nov.,comb.nov.,Cronobacter malonaticus sp.nov.,Cronobacter turicensis sp. nov.,Cronobacter muytjensii sp.nov.,Cronobacter dublinensis sp.nov.,Cronobacter genomospecies 1,and of three subspecies,Cronobacter dublinensis subsp.dublinensis subsp.nov.,Cronobacter dublinensis subsp.lausannensis subsp.nov.and Cronobacter dublinensis subsp.lactaridi subsp.nov[J].International Journal of Systematic and Evolutionary Microbiology,2008,58:1442-1447.
[12] Iversen C,Lehner A,Fricker-Feer C,et al.Gen-otyping of Cronobacter(Enterobacter sakazakii)strains isolated from a infant formula processing plant[J].Arch Lebensmittelhyg,2009,60:66-72.
[13] Mullane N,Healy B,Meade J,et al.Dissemination of Cronobacter spp.(Enterobacter sakazakii)in a powdered milk protein manufacturing facility[J].Applied and Environmental Microbiology,2008,74:5913-5917.
[14] Mullane N R,Whyte P,Wall P G,et al.Application of pulsed-field gel electrophoresis to characterise and trace the prevalence of Enterobacter sakazakii in an infant formula processing facility[J].International Journal of Food Microbiology,2007,116:73-81.
[15] Kalia A,Rattan A,Chopra P.A method for extraction of high-quality and high-quantity genomic DNA generally applicable to pathogenic bacteria[J].Analytical Biochemistry,1999,275:1-5.
[16] Versalovic J,Koeuth T,Lupski J R.Distribution of repetitive DNA sequences in eubac-teria and application to fingerprinting of bacterial genomes[J].Nucleic Acids Research,1991,19:6823-6831.
[17] Hunter P R,Gaston M A.Numerical index of the discriminatory ability of typing systems:an application of Simpson's index of diversity[J].Journal of Clinical Microbiology,1988, 26:2465-2466.
[18] Colombo M M,Mastrandrea S,Leite F,et al.Tracking of clinical and environmental Vibrio cholerae 01 strains by combined analysis of the presence of toxin cassette,plasmid content and ERIC PCR[J].FEMS Immunology and Medical Microbiology,1997,19:33-45.
[19] Austin B,Austin D A,Blanch A R,et al.A comparison of methods for the typing of fishpathogenic Vibrio spp.[J].Systematic and Applied Microbiology 1997,20(1):89-101.
[20] Swaminathan B,Barrett T J,Hunter S B,et al.PulseNet:the molecular subtyping network for foodborne bacterial disease surveillance,United States[J].Emerging Infectious Diseases,2001,7(3):382-389.
[21] Baldwin A,Loughlin M,Caubilla-Barron J,et al.Multilocus sequence typing of Crono-bacter sakazakii and Cronobacter malonaticus reveals stable clonal structures with clinical significance which do not correlate with biotypes[J].BMC Microbiology,2009,9:223-231.
