乳酸菌蛋白水解体系及相关基因表达的研究进展
2013-12-08杜越欧侯俊财
杜越欧,侯俊财
(东北农业大学,乳品科学教育部重点实验室,黑龙江哈尔滨150030)
乳酸菌是一类革兰氏阳性菌,能够在厌氧或兼性厌氧条件下发酵己糖产生乳酸,形成与人类生活密切相关的发酵产品。选育优良的乳酸菌发酵剂菌株对于生产高品质的发酵产品至关重要。乳酸菌自身不能利用外源蛋白质为菌体提供营养,必须利用自身的蛋白水解体系对外源蛋白质进行水解,最终满足对游离氨基酸的需要。乳酸菌的蛋白水解体系主要由胞外酶、转运系统和胞内酶组成。其中胞外酶将外源蛋白质(主要是酪蛋白)水解为多肽,这些多肽再由转运系统运送至胞内由胞内酶水解成游离的氨基酸以供菌体的生长需要。其中胞内酶的种类较多,主要有肽链内切酶和端肽酶两种。由此可见,乳酸菌的蛋白水解体系对于其自身的生长非常重要。因此,研究乳酸菌蛋白水解体系内的关键酶的变化规律对于提高乳酸菌的品质具有重要的意义。目前国内外的学者都开始从分子生物学的角度对乳酸菌的蛋白代谢进行深入研究,通过运用实时荧光定量PCR等方法检测蛋白酶基因的表达情况。此外,研究人员还通过对乳酸菌的蛋白水解酶基因进行基因修饰得到水解能力更强的发酵剂优势菌株[1]。
1 乳酸菌的蛋白水解体系
由于乳酸菌不能直接利用外源蛋白质和无机氮源,必须通过降解外源蛋白质或肽,以保证菌体正常生长代谢对氨基酸的需要。现乳酸菌已进化到拥有一套完整的蛋白水解体系,对大分子酪蛋白进行逐步降解,以满足对氨基酸的需要。该蛋白水解体系主要包括三个部分:第一部分为将大分子酪蛋白水解成多肽的胞外蛋白酶;第二部分是将多肽转运至胞内的多个转运系统;第三部分为将转运至胞内的多肽进一步水解成自由氨基酸的多种肽酶(图1),这些氨基酸最终进行代谢或合成蛋白质。

表 1 乳酸菌的肽酶[3,12]Table 1 The peptidases of the lactic acid bacteria[3,12]
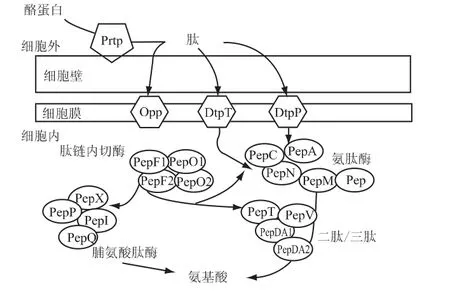
图1 乳酸菌的蛋白水解体系图解[2]Fig.1 Schematic representation of the lactic acid bacteria proteolytic system[2]
目前,乳酸菌的蛋白水解体系在乳球菌中已经得到了很好的研究[2-4],并发现乳酸杆菌的蛋白水解体系的结构特征和乳球菌相似。研究发现,菌体对于肽的吸收要优先于氨基酸的吸收。A Picon等人[5]对24株乳球菌的蛋白水解体系中相关酶的酶活进行测定,发现其中18株菌中含有Opp和Dpp两个转运系统,并且其活力较高。此外这些菌株还具有活力较高的肽酶。
由图1可见,乳酸菌水解酪蛋白的蛋白水解体系的三个部分分别包括多种重要的酶。下面分别对这三个部分的组成及作用进行综述。
1.1 胞外酶
乳酸菌水解酪蛋白的第一步是通过胞外酶来实现的。胞外酶是一类将酪蛋白分解成多肽的蛋白酶。研究发现,胞外酶主要有PrtP、PrtB和 PrtS三种,其中PrtP主要存在于乳酸乳球菌,PrtB主要存在于保加利亚乳杆菌,PrtS主要来自嗜热链球菌[6]。此外,瑞士乳杆菌中主要存在胞外酶基因为 PrtH,L Sadat-Mekmene等人[7]对瑞士乳杆菌中两种胞外蛋白酶PrtH和PrtH2的活性进行了研究,探讨含有一种或两种胞外蛋白酶的不同菌株对于外源蛋白质的水解活性的差异。
1.2 转运系统
由胞外酶水解下来的多肽需要通过细胞膜上的转运系统将其运送至胞内,这也是乳酸菌水解酪蛋白的第二步。经过研究证实,乳酸菌的多肽转运系统有三种,分别是转运二肽和三肽的Dtp P和Dtp T转运系统和转运寡肽的Opp转运系统。其中DtpT和Dpp系统是单个蛋白质,转运亲水性的二肽和三肽,但是由于二肽和三肽都不是由酪蛋白释放的,因此转运二肽和三肽的Dtp P和DtpT转运系统对乳酸菌的生长都是不必需的[8]。实验证明不能表达编码Opp系统的基因突变菌株不能转运多肽,也不能在乳中生长。由此得出,第三种Opp寡肽转运系统对于乳酸菌的蛋白水解体系是最重要的。Opp转运系统是由Opp复合体构成,其属于转运系统中的ABC(ATP结合盒)家族,它含有5个亚基:包括两个转运膜蛋白Opp B和Opp C,两个ATP结合蛋白OppD和Opp F和一个膜连接底物结合蛋白OppA[9-10]。
1.3 肽酶
乳酸菌蛋白水解体系的第三步,是将转运至胞内的多肽、二肽和三肽分解成游离的氨基酸,最终供菌体生长需要,这一步将由肽酶来实现。研究表明,肽酶大体分为内肽酶和端肽酶两种。内肽酶也称肽链内切酶或蛋白酶,主要是水解蛋白质和多肽链内部的肽键,形成多种多肽和寡肽。端肽酶又称肽链端解酶或外肽酶,主要是从肽链的一端开始水解,将氨基酸一个一个地从多肽链上分解下来。端肽酶根据其作用位置不同可分为氨肽酶、羧肽酶、二肽酶和三肽酶,氨肽酶是从肽链的氨基末端开始水解肽并释放N端氨基酸的酶,而羧肽酶是从链的羧基末端开始水解肽链。此外,二肽酶和三肽酶(PepT)在乳酸菌蛋白水解体系中同样具有非常重要的作用,研究发现,一些二肽酶属于脯氨酰氨基酸二肽酶或氨酰基脯氨酸二肽酶,主要水解N端或C端具有脯氨酸的肽[3,11]。表1列出一些关键的肽酶的名称和作用位置。
由表1可以看出,不同的胞内酶作用于多肽的位置大不相同,而相关研究还发现即使同一种肽酶也可能有不同的作用位置,如乳酸乳球菌中PepX除了能水解寡肽之外,还能水解二肽和一些不含脯氨酸的多肽[12]。
2 蛋白水解体系中相关基因的表达
2.1 检测基因表达的方法
目前检测基因表达的方法主要有Northern blot、RT-PCR、mRNA差异显示、cDNA代表性差异分析和实时荧光定量PCR,这些都是从转录水平上对基因的表达水平进行分析。其中实时荧光定量PCR是检测基因表达量差异的最有效的方法之一,其可以对某一基因在不同样本中的表达水平或在同一样本经过不同处理前后的表达水平进行定量分析,此技术已被广泛的用于各种基因在不同环境中的差异表达的研究[13]。
实时荧光定量PCR是在PCR反应体系中加入荧光基团,利用荧光信号累积实时监测整个PCR进程,最后通过特定数学原理对未知模板进行定量分析的一种快速有效的方法。而普通PCR只能通过终点法对扩增产物进行定性分析,而不能进行定量分析,并且存在误差[14]。定量PCR仪是在普通PCR仪的基础上加装了荧光激发装置和荧光检测装置,PCR扩增和检测同时进行,最终结果不需进行电泳检测,所以有效地避免了样品间的交叉污染。实时荧光定量PCR分为绝对定量和相对定量。在定量的过程中涉及到一个非常重要的概念-CT值。CT(Cycle threshold)值是指荧光定量PCR每个反应管内的荧光信号到达设定的域值时所经历的循环数,它与起始模板的对数存在线性关系因此可利用标准曲线对未知样品进行定量。不同的实验目的采用不同的定量方法,绝对定量需要制作标准曲线确定样本中某一基因的具体拷贝数或浓度。而相对定量一般应用于比较不同样本之间某一基因的表达量高低或是经过不同处理的几个样本之间基因表达水平的高低变化。在相对定量中,必需选择一个表达量恒定的内源性管家基因(house keeping gene)作为内参照物(简称内参),以管家基因的量来作为某种标准比较来源不同的样本中目的基因表达量的差异,最终运用 2-ΔΔCT法进行数据处理分析[15-16]。实时荧光定量PCR技术已被广泛的用于乳酸菌在不同胁迫或是生长条件下基因差异表达的研究[17]。
2.2 乳酸菌蛋白水解体系中相关基因的表达情况
乳酸菌蛋白水解体系中部分相关基因已经被检出,其中乳酸球菌的三个不同的肽类转运系统为:DtpT、DtpP和寡肽转运系统Opp。乳酸杆菌关于肽类转运系统的研究要明显少于乳酸球菌,只有Lactobacillus helveticus的一个转运蛋白的基因DtpT被克隆并测序,并证实 Lactobacillus helveticus的DtpT转运系统与Lactococcus lactis的DtpT转运系统高度相似[18]。此外,Kirsi Peltoniemi等人[18]已经克隆出Lactobacillus delbrueckii subsp.bulgaricus B14的Opp寡肽转运系统的基因。据研究发现,Opp的操纵子由OppD,OppF,OppB,Opp C和Opp A1五个基因组成。与此同时,通过基因比对发现保加利亚乳杆菌的Opp转运系统与其他细菌的转运系统和乳酸乳球菌的寡肽转运系统存在高度的相似性。此外,HENRIKSEN等人[19]将部分肽酶基因克隆并转入L.Lactis MG 1363中使其表达,制作成单一菌株发酵剂,结果表明,部分转基因的发酵剂对于改善奶酪品质有显著影响。Leila Sadat-Mekmene等人[20]对Lactobacillus helveticus中蛋白水解体系做了简要的介绍,并详细论述了蛋白水解体系中的胞外酶编码基因的最新信息以及胞外酶对于水解酪蛋白时的活性。M A Azcarate-Peril等人[21]研究了在脱脂乳中不同蛋白水解酶基因的差异表达,得出一些肽酶、转运系统的基因和PrtM、PrtP基因的表达量都是随着菌体的生长而上升。Opp1基因的表达量在4h时达到最大,Opp2基因的表达量则随着时间的延长不断增长至12h时才达到最大,由此得出两个转运系统拥有不同的特征。Ji-Cheng Wang等人[22]利用实时荧光定量PCR技术研究了Lactobacillus casei Zhang在豆乳中生长时,其蛋白水解体系中相关基因的表达量变化。结果发现,与菌体生长到稳定期时相比,在对数期时,胞外酶基因、转运系统基因以及多种胞内酶基因的表达量大都是显著上调的。得出结论,蛋白水解体系在菌体的生长过程中起到了非常重要的作用。Rina Wu等人[23]同样利用实时荧光定量PCR技术初步探讨了在mRNA水平上一些肽酶基因在低酸环境下的表达变化情况,发现肽酶Pep P基因的表达量随着pH的降低而下降。
Nicoline Vermeulen 等人[24]检出了 Lactobacillus sanfranciscensis DSM 20451中编码肽类转运系统Opp和DtpT的基因和编码胞内酶 PepT、Pep R、PepC、PepN、Pep X的基因,并且发现除了 Pep X基因,L.Sanfranciscensis的肽类转运系统和一些胞内肽酶的基因都是在发酵过程中得以表达。此外,Nicoline Vermeulen等人利用荧光定量PCR技术检测了L.Sanfranciscensis的 DtpT,Opp F,Pep C,PepR 和 Pep T基因在菌体不同生长阶段表达的动态变化规律(见表2)。由表2可以看出,不同生长时期和肽供应的不同对于pep T基因转录水平的影响要远远小于对于dtp T和opp F基因转录水平的影响。在未添加肽的发酵过程中,对数期OppF、DtpT和PepT的表达水平要明显高于稳定期,而在添加肽的发酵过程中,与对数期相比,稳定期Opp F和Dtp T的表达水平都是降低的,但是PepT的表达水平却是升高的。在发酵过程中随着肽含量的增多,Opp和Dtp T基因的表达量减少17倍,而PepT基因的表达量则在较小的范围内依赖于肽的含量。因此,肽含量的高低是乳酸菌蛋白水解体系中相关基因表达水平的一个限制性因素。

表2 L.Sanfranciscensis DSM 20451在发酵生长过程中肽类转运系统和水解肽类的相关基因的表达情况Table 2 Expression of genes related to peptide transport and hydrolysis by L.sanfranciscensis DSM 20451 during growth in sourdough
从上述研究中可以看出,乳酸菌蛋白水解体系中转运系统和相关酶的基因已经被检出,并且国内外学者正在利用分子技术研究这些基因在转录水平上表达的变化规律。初步得出,菌体在不同生长阶段时,蛋白水解体系中相关基因的转录水平有显著的差异,并且随着菌体的不断生长表达量呈下降趋势。此外,乳酸菌蛋白水解体系中相关基因的表达水平还受肽含量的影响。至于培养过程中的其他因素对乳酸菌蛋白水解体系中相关基因的表达水平是否有影响还有待进一步的研究。如能找到更多蛋白水解体系相关基因表达水平的限制性因素,通过优化培养条件,使关键基因得到最大的表达量,就能从根本上提高菌体的品质,生产出优质的发酵剂菌株。在以后的研究中,应该着重研究乳酸菌蛋白水解体系中相关基因的表达变化规律,从基因层面上改变发酵剂菌株的品质。
3 结语
乳酸菌自身不能利用外源无机氮和蛋白质,必需通过蛋白水解体系中的胞外酶将酪蛋白分解成多肽,再由Opp转运系统运送至胞内,通过胞内的肽链内切酶和端肽酶将各种多肽分解成游离的氨基酸以满足菌体的生长需要,因此蛋白水解能力的强弱影响着乳酸菌的正常生长。
目前,很多对乳酸菌蛋白代谢的研究都已经从分子的层面入手[25-29],通过深入研究可从基因水平掌握乳酸菌的蛋白代谢机制,从而利用代谢调控手段提高乳酸菌的生长数量和代谢水平,推动乳酸菌研究的迅速发展,获得高水平的发酵产品。
[1]Henriksen M E,Nilssond.Industrial application of genetically medified microorganisms:gene technology at Chr.Hansen A/S[J].International Dairy Journal,1999,9:17-23.
[2]Eric Guedon,Pierre Renault,Ehrlich S D,et al.Transcriptional pattern of genes coding for the proteolytic system of lactococcus lactis and evidence for coordinated regulation of key enzymes by peptide supply[J].Journal of Bacteriology,2001,183(12):3614-3622.
[3]Christensen J E,Dudley E G,Pedersin J A,et al.Peptidases and amino acid catabolism in lactic acid bacteria[J].Antonie van Leeuwenhoek,1999,76:217-246.
[4]Roland J Siezen.Multi-Domain,cell-envelope proteinases of lactic acid bacteria[J].Antonie van Leeuwenhoek,1999,76:139-155.
[5]Picon A,García-Casado M A,Nuñez M.Proteolytic activities,peptide utilization and oligopeptide transport systems of wild Lactococcus lactis strains[J].International Dairy Journal,2010,20(3):156-162.
[6]Edmund R S Kunji,Igor Mierau,Anja Hagfing,et al.The proteolytic systems of lactic acid bacteria[J].Antonie van Leeuwenhoek,1996,70:187-221.
[7]Sadat-Mekmene L,Jardin J,Corre C,et al.Simultaneous Presence of PrtH and PrtH2 Proteinases in Lactobacillus helveticus Strains Improves Breakdown of the Pureαs1-Casein[J].Applied and Environmental Microbiology,2011,77(1):179-186.
[8]Foucaud C,Kunji E R S,Hagting A,et al.Specificity of peptide transport systems in Lactococcus lactis:evidence for a third system which transports hydrophobic di-and tripeptides[J].JBacteriol,1995,177(80):4652-4657.
[9]Kunji E R S,Hagting A,de Vried C J,et al.Transport of β-casein-derived peptides by the oligopeptide transport system is a crucial step in the proteolytic pathway of Lactococcus lactis[J].J Biol Chem,1995,270(4):1569-1574.
[10]Tynkkynen S,Buist G,Kunji E,et al.Genetic and biochemical characterization of the oligopeptide transport system of Lactococcus lactis[J].JBacteriol,1993,175(12):7523-7532.
[11]Savijoki K, Palva A.Purification and molecular characterization of a tripeptidase(PepT)from Lactobacillus helveticus[J].Applied and Environmental Microbiology,2000,66(2):794-800.
[12]白凤翎,张柏林,赵宏飞.乳酸菌蛋白代谢研究进展[J].食品科学,2010,31(19):381-384.
[13]Mayo B,Kok J,Venema K,et al.Molecular cloning and sequence analysis of the X-propyl dipeptidyl aminopeptidase gene from Lactococcus lactis subsp.cremoris[J].Applied and Environmental Microbiology,1991,57(1):38-44.
[14]朱捷,杨成君,王军.荧光定量PCR技术及其在科研中的应用[J].生物技术通报,2009(2):73-76.
[15]Kenneth J,Livak,Thomas D Schmittgen.Analysis of Relative Gene Expression Data Using Real-Time Quantitative PCRand the 2-ΔΔCTMethod[J].Methods,2001,25:402-408.
[16]Hubert G M Niesters.Quantitation of viral load using realtime amplification[J].Methods,2001,25:419-429.
[17]黄桂东.Laetobacillus brevis NCL912的耐酸特性及其酸胁迫下差异表达蛋白的研究[D].南昌:南昌大学,2011.
[18]Kirsi Peltoniemi,Erkki Vesanto,Airi Palva.Genetic characterization of an oligopeptide transport system from Lactobacillus delbrueckii subsp.Bulgaricus[J].Arch Microbiol,2002,177:457-467.
[19]Henriksen M E,Nilssond.Industrial application of genetically medified microorganisms:gene technology at Chr.Hansen A/S[J].International Dairy Journal,1999,9:17-23.
[20]Leila Sadat-Mekmene,Magali Genay,Danièle Atlan,et al.Original features of cell-envelope proteinases of Lactobacillus helveticus.A review[J].International Journal of Food Microbiology,2011,146:1-13.
[21]Azcarate-Peril M A,Tallon R,Klaenhammer T R.Temporal gene expression and probiotic attributes of Lactobacillus acidophilus during growth in milk[J].Journal of Dairy Science,2009,92(3):870-886.
[22]Ji-Cheng Wang,Wen-Yi Zhang,Zhi Zhong,et al.Transcriptome analysis of probiotic Lactobacillus casei Zhang during fermentation in soymilk[J].J Ind Microbiol Biotechnol,2012,39:191-206.
[23]Rina Wu,Wenyi Zhang,Tiansong Sun,et al.Proteomic analysis of responses of a new probiotic bacterium Lactobacillus casei Zhang to low acid stress[J].International Journal of Food Microbiology,2011,147:181-187.
[24]Nicoline Vermeulen,Melanie Pavlovic,Matthias A,et al.Functional characterization of the proteolytic system of lactobacillus sanfranciscensis dsm 20451tduring growth in sourdough[J].Applied and Environmental Microbiology,2005,71(10):6260-6266.
[25]Magnani D,Barre O,Gerber S D,et al.Characterization of the CopR regulon of Lactococcus lactis IL1403[J].J Bacteriol,2008,190(2):536-545.
[26]Larsen R,Van Hijum S,Martinussen J,et al.Transcriptome analysis of the Lactococcus lactis ArgR and AhrC regulons[J].Applied and Environmental Microbiology,2008,74(15):4768-4771.
[27]Ramiah K,Van Reenen CA,Dicks L M T.Expression of the mucus adhesion genes Mub and MapA,adhesion-like factor EF-Tu and bacteriocin gene plaA of Lactobacillus plantarum 423,monitored with real-time PCR[J].International Journal of Food Microbiology,2007,116:405-409.
[28]Marco M L,Kleerebezem M.Assessment of real-time RTPCR for quantification of Lactobacillus plantarum gene expression during stationary phase and nutrient starvation[J].Journal of Applied Microbiology,2008,104:587-594.
[29]孙洁.乳酸菌发酵剂菌株的自溶特性及机理研究[D].中国农业科学院,2010.
