玉树马和德保矮马SLC36A1基因多态性研究*
2013-11-30黄洁萍王风巧党瑞华雷初朝
徐 苹,黄洁萍,王风巧,党瑞华,雷初朝
(西北农林科技大学 动物科技学院,陕西 杨凌712100)
毛色是鉴定马品种及性能的一项重要指标。随着养马业向赛马与娱乐方向发展,饲养者对马毛色的重视程度日益增加。马香槟毛色是饲养者非常钟爱的一种毛色,它表现为红毛色和黑毛色强度的同时减弱,造成马的被毛颜色从栗色变为金黄香槟色,枣色变为琥珀香槟色,黑色变为经典香槟色[1]。马香槟毛色由常染色体上CH(Champagne gene)基因家族控制,该基因家族位于14号染色体上6 cM区域内,含有4个候选基因SPARC、SLC36A1、SLC36A2、SLC36A3[1-4]。马SLC36A1(Solute Carrier 36 family A1)基因编码溶酶体氨基酸转运蛋白(LYAAT1/PAT1),在氨基酸及其衍生物跨膜运动中起到重要作用。研究表明SLC36A1基因在人类和大鼠的小肠上皮细胞及大脑部分神经细胞中表达,该基因表达的蛋白(PAT1)能够将溶酶体水解产生小分子蛋白转运到细胞外[5-7]。
迄今为止,SLC36A1基因的碱基突变对蛋白质功能的影响还不清楚,但是有关研究表明溶质载体36家族的同源基因与色素沉积有关,如SLC45A2(MATP)基因与马奶油毛色相关。Cook等[1]通过研究马SLC36A1基因,发现外显子2上C→G的突变是产生马香槟毛色的关键位点,该单碱基突变造成编码的苏氨酸变为精氨酸,从而改变皮肤色素的沉着,呈现香槟毛色,但是具体的分子作用机制还有待进一步研究。目前国内外对马香槟毛色的研究不多,尚没有关于中国马香槟毛色的研究,因此本试验以玉树马和德保矮马为对象,分析中国马SLC36A1基因的多态性,旨在从分子水平揭示中国家马毛色的遗传资源多样性,为中国家马毛色遗传多态性鉴定提供科学依据。
1 材料与方法
1.1 试验材料
采用随机抽样方法,从青海省玉树县和广西壮族自治区德保县分别采集了52匹玉树马(YSH)和22匹德保矮马(DBP)血样,均为颈静脉采血,ACD抗凝,-80 ℃超低温保存备用。
1.2 试验方法
1.2.1 引物设计与PCR扩增 利用DNA提取试剂盒(上海生工生物工程技术服务有限公司)提取马血液中的基因组DNA,4℃保存。马香槟毛色SLC36A1基因的10个外显子及部分内含子引物(表1)参照已经发表的文献[1]。以家马的混合DNA池(每10个不同样本混合成一个DNA池)做模板进行基因的PCR扩增。PCR扩增体系为:2 μL混合DNA池模板(10 ng/μL)、正反向引物各2 μL(10 μmol/L)、25 μL Mix、19 μL ddH2O,反应总体积为50 μL。PCR扩增程序:预变性94 ℃ 5 min,94 ℃ 35 s,退火(退火温度见表1)40 s,72 ℃延伸40 s,35个循环,最后72 ℃充分延伸10 min,4 ℃保存。PCR产物用1%的琼脂糖凝胶电泳检测。检测质量好的PCR产物送至上海生工生物工程技术服务有限公司进行双向测序。

表1 马SLC36A1基因扩增引物及条件Table1 Primers and PCR conditions of Equine SLC36A1 gene
注:每对外显子引物扩增片段包含部分内含子。
Note:The PCR product of every exon primer contains partial intron.
1.2.2 马SLC36A1基因PCR-RFLP分型 利用PCR-RFLP技术对确定的SNPs进行基因分型。酶切扩增体系为:10×Buffer 1 μL、限制性内切酶1 μL、PCR产物5 μL、灭菌水9 μL共16 μL体系混匀,按照限制性内切酶说明书中给定的反应温度水浴8 h(见表2)。反应产物用3%的琼脂糖进行电泳检测,使用凝胶成像仪照相,统计基因型。对于没有合适的酶切位点的SNPs,采用直接测序分型。
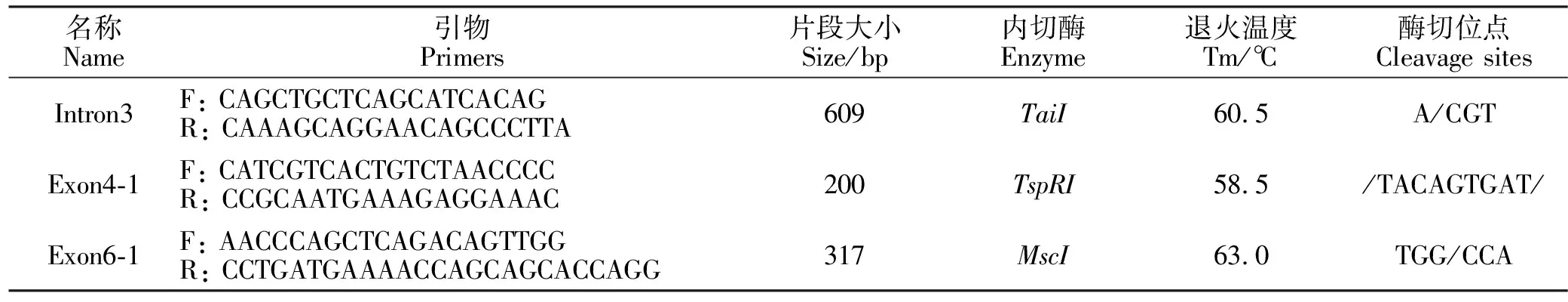
表2 PCR-RFLP引物及条件Table 2 Primers and conditions of PCR-RFLP
注:外显子4和外显子6的酶切引物为另外设计。
Note: The cleavage primers for exon4 and exon6 are designed additionally.
1.2.3 数据分析 利用DNASTAR Lasergene软件校对测序产物的原始序列,用ClustalX软件进行序列比对。利用SHEsis在线软件计算基因型频率,哈代-温伯格平衡检测,单倍型频率分析;利用PopGene软件计算单倍型多样度(H)及Ewens-Watterson中性检验。
2 结果与分析
2.1 马SLC36A1基因多态性
利用家马DNA池为模板,通过PCR扩增和DNA测序分析玉树马52个样本和德保矮马22个样本中SLC36A1基因(NW_001867377.1)的10个外显子及部分内含子序列,共发现了5个SNPs,分别位于内含子3(g.26699953 A>G, g.26699851 G>C, g.26699850 G>C),外显子4(g.26699562 G>A)及外显子6(g.26697018 C>T)。其中,外显子4(g.26699562 G>A)的SNP为同义突变,外显子6(g.26697018 C>T)为错义突变(p. Ala172Val)。
依次利用TaiI, TspRI, MscI三种限制性内切酶对内含子3(g.26699850 G>C)、外显子4及外显子6上的3个SNPs进行酶切分型。其中内含子3存在3个酶切位点,73 bp处为发现的SNP位点;外显子4有3个酶切位点,117 bp处为SNP存在的位点,外显子6有1个酶切位点(见表3)。结果发现内含子3的SNP(g.26699850 G>C)有GG、GC基因型;外显子4的SNP有GG、GA基因型;外显子6的SNP有CC、CT基因型(见图1)。

表3 马SLC36A1基因PCR-RFLP多态性Table 3 The PCR-RFLP polymorphism of horse SLC36A1 gene

图1 马SLC36A1基因3个SNP位点的PCR-RFLP多态性Fig.1 The PCR-RFLP polymorphism of the three SNPs in horse SLC36A1 gene
内含子3上发现的另外2个SNPs(g.26699851 G>C;g.26699953 A>G)找不到合适的限制性内切酶的切点,因此对这2个SNPs进行直接测序分型,结果发现这2个SNPs都有3种基因型(见图2)。根据PCR-RFLP和直接测序分型结果统计基因型频率(见表4),结果显示5个SNPs的纯合突变较少,野生型为优势基因型。哈代-温伯格平衡检验表明玉树马和德保矮马在5个突变位点均处于平衡状态(P>0.01)。
2.2 SLC36A1基因单倍型与遗传多样度
通过分析玉树马和德保矮马SLC36A1基因的5个SNPs,共鉴定了9种单倍型(H1-H9),其中单倍型H5所占比例最高(73.0%),其余各单倍型所占比例均很少,以H1和H7单倍型出现的概率最低。单倍型H1,H4,H6仅存在于玉树马中,H3和H8只在德保矮马中(见表5)。两个家马品种的平均遗传多样度为0.3160,德保矮马的单倍型多样度为0.4190,高于玉树马(0.2228)。Ewens-Watterson中性检验结果显示,5个SNPs位点的观测频率处于显著水平上下限值之间,表明这5个位点均没有偏离明显的中性区,没有受到明显的自然选择作用(见表6)。

图2 马SLC36A1基因内含子3的2个SNPs直接测序鉴定的基因型Fig.2 The genotype of 2 SNPs of intron 3 in horse SLC36A1 gene

品种Breed个数SamplesSLC36A1基因型及频率 Genotype and genotype frequencies of SLC36A1 geneg.26697018C>Tg.26699562G>Ag.26699850G>Cg.26699851G>Cg.26699953A>GCCCTGGGAGGGCGGGCCCAAAGGGYSH521.0000.0000.9230.0770.7500.2500.8080.1920.000 0.808 0.1920.000 DBP220.6820.3181.0000.0000.4560.5440.545 0.3180.1360.545 0.3180.136哈代-温伯格平衡Hardy-WenbergequilibriumP=0.699P=0.811P=0.080P=0.283P=0.283
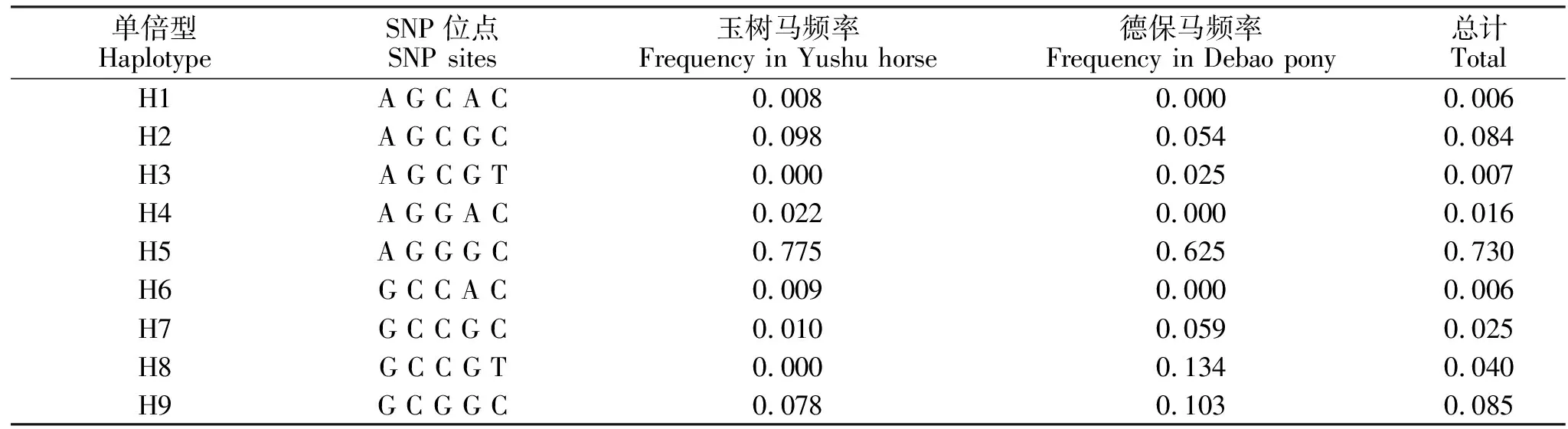
表5 玉树马和德保矮马SLC36A1基因单倍型及频率Table 5 Haplotype frequency of SLC36A1 gene in Yushu horse and Debao pony
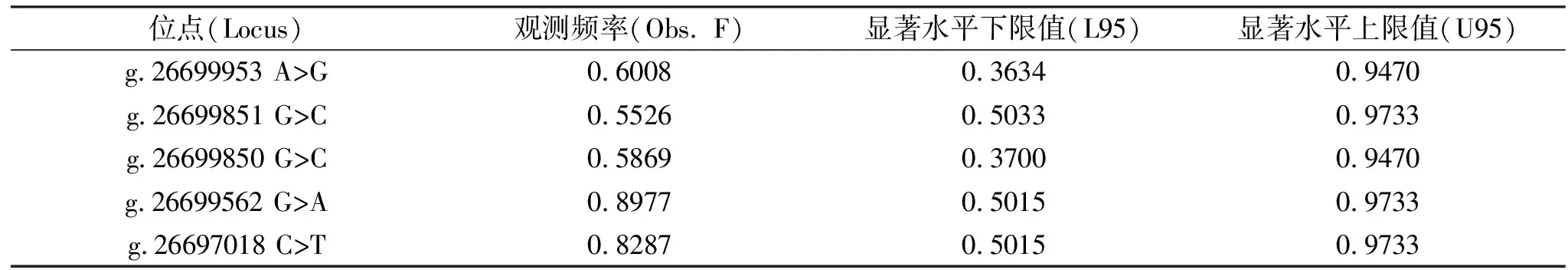
表6 马5个SNPs位点的Ewens-Watterson 中性检验Table 6 The Ewens-Watterson test for neutrality of horse five SNPs
3 讨 论
毛色是马品种鉴定的重要因素之一,近年来研究者对哺乳动物的毛色进行了大量研究,已发现了超过300个毛色基因位点和150个与毛色相关的基因[8]。Stachurska[9]对1130匹纯血马和796 匹阿拉伯马不同毛色(棕色、栗色、深棕色等亮色系毛色)进行了QTL分析,认为MC1R 位点(E位点) 的活性与马的亮色系毛色形成有关;Sulem等[10]研究表明,在欧洲人群中,ASIP 基因的多态性与皮肤光敏感性、雀斑以及红发有直接的关系;Haase 等[11]对不同马种中KIT基因的全部21个外显子进行研究,证明KIT基因多态性与马白色系毛色形成有关。尽管如此,目前有关于马香槟毛色的研究还相对有限,Cook等[1]对马SLC36A1基因的研究是目前唯一1篇有关SLC36A1基因与香槟毛色关系的报道,该研究发现SLC36A1基因外显子2上的一个碱基突变造成了马香槟毛色,推测可能是该突变改变了黑色素体合成过程中的pH值,从而影响酪氨酸的合成,但是其确切的作用机制尚不清楚。溶质载体(SLC)家族的其他同源基因已经被证实与相应的毛色相关,马SLC45A2(MATP)基因与奶油毛色密切相关[12];而在人类中,SLC45A2的突变(p.Ala111Thr)与皮肤颜色有关[13];小鼠SLC45A5基因则与金黄毛色消退相关[14]。
本研究对中国马SLC36A1基因的遗传多样性进行研究,共发现5个碱基突变,其中前两处及最后一处为颠换,第三处和第四处为转换,外显子4为同义突变,外显子6为错义突变(p. Ala172Val)。5个SNPs共构建了9种单倍型,其中H5所占比例最大为最主要单倍型。其中,玉树马有7个单倍型,德保矮马有6个单倍型,H2、H5、H7、H9为共享单倍型。Cook等[1]在SLC36A1基因中发现的唯一突变C→G位于外显子2上,并证实该突变造成香槟毛色表型。本研究没有在外显子2上发现造成香槟毛色的C→G碱基突变,但是确定了SLC36A1基因外显子4和外显子6上的碱基突变(外显子4 g.26699562 G>A,外显子6 g.26697018 C>T),并且发现外显子6的碱基突变造成该位点编码的氨基酸由Ala变为Val。由于本试验采集的样本没有相关香槟毛色表型的记录,因此不能简单的根据外显子2中的突变来确定玉树马和德保马中香槟毛色个体的存在。这一问题的解决还亟待于对外显子6中SNP与香槟毛色表型性状关系的进一步研究。单倍型多样度分析表明两个马品种的平均遗传多样度为0.3160,表明玉树马和德保矮马SLC36A1基因的遗传多样性均不丰富,中国马毛色表型遗传多样性低。Ludwig等[15]研究认为马最早出现的毛色为红棕色(12000 BC),在进化过程中,马的红棕色表型逐渐向黑色推进,到5000 BC时开始出现毛色消褪表型和斑点表型,这表明马毛色消退基因出现较晚。因此本研究认为中国家马香槟毛色表型出现较晚,表型扩张范围有限,推测中国马毛色表型可能比较古老,这与毛春春等[16]在马KIT基因多态性上的研究结果一致。本研究采集的玉树马样本数大于德保矮马,但是结果发现德保马的单倍型多样度高于玉树马,这可能是由于玉树马采自青海省偏远地带,自然环境恶劣,马匹之间遗传关系较近。另外,随着马役用价值的减弱,很多地方马品种的数量减少,加之饲养者对其毛色的重视和选育程度不够,最终造成了香槟毛色遗传多样性低的现状。哈代-温伯格平衡研究表明,所发现的5个碱基突变均处于平衡状态。Ewens-Watterson中性检验结果也表明,该5个SNPs没有偏离明显的中性区,因此说明中国家马SLC36A1基因没有受到明显的自然选择。本研究首次对中国家马SLC36A1基因遗传多样性进行研究,发现其单倍型多样度相对较低,可以对今后研究家马香槟毛色多样性、选育研究提供理论依据。此外,本试验没有找到外显子2上与香槟毛色相关的突变,但是确定了外显子6上的碱基突变造成编码的丙氨酸转变为缬氨酸,因此需要通过更多的研究来确定该位点突变与香槟毛色的关系以及中国香槟毛色家马分布情况。
参考文献:
[1] Barsh G S, Cook D, Brooks S, et al. Missense Mutation in Exon 2 of SLC36A1 Responsible for Champagne Dilution in Horses[J]. PLoS Genetics, 2008, 4(9): e1000195.
[2] Boll M, Daniel H, Gasnier B. The SLC36 family: proton-coupled transporters for the absorption of selected amino acids from extracellular and intracellular proteolysis [J]. Pflügers Archiv European Journal of Physiology, 2004, 447(5): 776-779.
[3] Boll M, Foltz M, Rubio-Aliaga I, et al. A cluster of proton amino acid transporter genes in the human and mouse genomes [J]. Genomics, 2003, 82(1): 47-56.
[4] Thwaites D T, Mcewan G T A, Cook M J, et al. H+-coupled (Na+-independent) proline transport in human intestinal (Caco-2) epithelial cell monolayers [J]. FEBS letters, 1993, 333(1): 78-82.
[5] Chen Z, Fei Y J, Anderson C M H, et al. Structure, function and immunolocalization of a proton-coupled amino acid transporter (hPAT1) in the human intestinal cell line Caco‐2 [J]. The Journal of Physiology, 2003, 546(2): 349-61.
[6] Bermingham J R, Pennington J. Organization and expression of the SLC36 cluster of amino acid transporter genes [J]. Mammalian Genome, 2004, 15(2): 114-125.
[7] Anderson C M, Grenade D S, Boll M, et al. H+amino acid transporter 1 (PAT1) is the imino acid carrier: An intestinal nutrient drug transporter in human and rat [J]. Gastroenterology, 2004, 127(5): 1 410-1 422.
[8] Cieslak M, Reissmann M, Hofreiter M, et al. Colours of domestication [J]. Biological Reviews, 2011, 86(4): 885-899.
[9] Stachurska A, Pieta M, Lojek J, et al. Performance in racehorses of various colours [J]. Livestock Science, 2007, 106(2): 282-286.
[10] Sulem P, Gudbjartsson D F, Stacey S N, et al. Two newly identified genetic determinants of pigmentation in Europeans [J]. Nature Genetics, 2008, 40(7): 835-837.
[11] Haase B, Brooks S A, Schlumbaum A, et al. Allelic heterogeneity at the equine KIT locus in dominant white (W) horses [J]. PLoS Genetics, 2007, 3(11): e195.
[12] Mariat D, Taourit S, Guérin G. A mutation in the MATP gene causes the cream coat colour in the horse [J]. Genetics Selection Evolution, 2003, 35(1): 119.
[13] Graf J, Hodgson R, Van Daal A. Single nucleotide polymorphisms in the MATP gene are associated with normal human pigmentation variation [J]. Human mutation, 2005, 25(3): 278-284.
[14] Hoekstra H. Genetics, development and evolution of adaptive pigmentation in vertebrates [J]. Heredity, 2006, 97(3): 222-234.
[15] Ludwig A, Pruvost M, Reissmann M, et al. Coat color variation at the beginning of horse domestication [J]. Science, 2009, 324(5926): 485.
[16] 毛春春,孙伟,刘东花,等. 德保矮马与哈萨克马KIT基因多态性分析[J]. 家畜生态学报, 2012,33(3):19-22.
