斑马鱼3种胰蛋白酶原基因的克隆、序列分析及组织表达*
2013-11-24陈文波李卫国焦春磊
陈文波,李卫国,张 珍,焦春磊,吴 帆
(河南理工大学资源环境学院,河南焦作 454000)
胰蛋白酶Trypsin(EC3.4.21.4)是丝氨酸蛋白酶家族的重要成员,是动物消化蛋白质的重要酶类[1]。胰蛋白酶以非活性酶原的形式被合成和分泌,被肠激酶或者激活的胰蛋白酶切割除去氨基端的酸性激活肽,继而转变成有活性的胰蛋白酶[2]。胰蛋白酶是一种肽链内切酶,主要裂解碱性氨基酸Arg或Lys羧基侧链肽。激活后的胰蛋白酶不仅可以激活胰蛋白酶原,而且还可以激活胰凝乳蛋白酶原、弹性蛋白酶原以及羧肽酶原等[3-5]。
目前研究认为,胰蛋白酶主要来源于鱼类的肝胰脏、肠道和幽门盲囊,在鱼类消化、生长速率和仔鱼的成活率方面都有着重要的作用。截至到目前,已在一些鱼类中也克隆得到了胰蛋白酶原的cDNA序列,如大西洋鳕鱼Gadus morhua[6]、牙鲆Paralichthys olivaceus[7]、蓝鳕鱼 Paranotothenia magellanica[8]、日本鳗 鲡 Anguilla japonica[9]、冬鲽Pseudopleuronectes americanus[10]、塞内加尔鳎 Solea senegalensis[11]、金头鲷Sparus aurata[12]、 草 鱼Ctenopharyngodon idellus[1]、翘嘴鲌 Culter alburnus[1]等。根据等电点的不同,目前把硬骨鱼类胰蛋白酶原主要分成了两种类型,分别为阴离子型胰蛋白酶原和阳离子型胰蛋白酶原,另外还有一种特殊的类型,主要是与极端环境相关[14]。同时,在一些海水鱼类中已发现多个编码胰蛋白酶原的基因,且分属于不同的胰蛋白酶类型,并展示了不同的组织和发育表达模式,提示这些胰蛋白酶的功能差异[6-7,10-11]。
目前在淡水鱼类中对胰蛋白酶的研究相对较少,仅在草鱼和翘嘴鲌中获得了其cDNA序列,且只报道了一种基因[1]。是否在淡水鱼类中也含有多个编码胰蛋白酶原的基因还需要进一步研究。本文以模式生物斑马鱼Danio rerio为研究对象,利用RT-PCR和RACE技术获得了3种斑马鱼胰蛋白酶原cDNA序列及氨基酸序列,并对其蛋白结构特征、同源性和进化关系进行了分析。同时,利用RT-PCR检测了三者的组织表达模式。这些结果将为深入研究斑马鱼以及脊椎动物胰蛋白酶原基因的进化和生理功能提供理论基础。
1 材料与方法
1.1 实验鱼和样品
实验用健康成年野生型斑马鱼购自花鸟鱼市场。用曝气除氯的自来水饲养于实验室的鱼缸中,用加热棒控制水温在24℃,控制昼夜时间比为14 h:10 h。取样时,先冰上麻醉斑马鱼,然后小心取出适量用于基因克隆和组织表达所需样品。样品立即置入预冷的含有1.0 mL Trizol的离心管中进行总RNA提取。所用各种试剂、消耗品和容器均用DEPC处理,解剖器具和玻璃器皿经180℃烘烤3 h,以灭活RNA酶。
1.2 试剂
Trizol Reagent购自 Invitrogen(USA)公司;E.Z.N.A胶回收试剂盒为Omega公司产品;Taq DNA Polymerase、DNase I购自 Fermentas(USA)公司;ReverTra Ace-α First-strand cDNA Synthesis Kit为Toyobo(Japan)公司产品。PCR所用引物和DNA测序均由上海英俊生物技术有限公司完成;其余均为国产分析纯试剂。
1.3 斑马鱼胰蛋白酶原基因的克隆
用Trizol法提取斑马鱼肝脏总RNA,260 nm下的吸光值衡量RNA的浓度,然后用w=0.8%琼脂糖凝胶,通过28S和18S条带的亮度来判断总RNA完整性。按照ReverTra Ace-α First-strand cDNA Synthesis Kit试剂盒说明书合成cDNA第一链。
对NCBI GenBank中已登录的鱼类胰蛋白酶原cDNA序列进行比对,在保守区域设计一对引物(TryF1和TryR1)进行斑马鱼胰蛋白酶原基因的克隆。PCR反应程序为94℃预变性3 min,然后94℃变性30 s,56℃退火30 s,72℃延伸30 s,40个循环后72℃延伸10 min。根据所获得中间片度序列设计特异引物,结合RACE试剂盒接头引物AAP和 AUAP,进行3'-RACE和5'-RACE扩增。将扩增得到的PCR产物在w=1.5%琼脂糖凝胶上电泳分离DNA片段,并在紫外光下回收目的条带,用E.Z.N.A胶回收试剂盒纯化目的产物并进行测序。

表1 用于斑马鱼胰蛋白酶原基因cDNA克隆和RT-PCR所需引物Table 1 Primers used for zebrafish trypsinogens gene cloning and expression analysis
1.4 序列分析
用DNAtools 6.0对开放阅读框进行分析并翻译成蛋白质;利用 SignalP 3.0 Server(http://www.cbs.dtu.dk/service/SignalP)进行信号肽的预测;采用Clustalx 1.83软件对多个物种胰蛋白酶原氨基酸序列进行比对,并用DNAstar软件对各序列的同源性进行计算;运用Mega 3.1软件,采用Neighbor-Joining法构建脊椎动物胰蛋白酶原蛋白的系统进化树。
1.5 斑马鱼3种胰蛋白酶原基因的组织表达分析
提取脑、肝脏、心脏、肌肉、卵巢、脾脏和肠组织的总RNA,经DNase I处理后,按照ReverTra Ace-α First-strand cDNA Synthesis Kit试剂盒说明书,用1 μg RNA进行反转录。PCR扩增前,分别对退火温度和循环数进行优化。PCR反应体系为20 μL,PCR程序为94℃预变性3 min,94℃变性15 s,56℃退火15 s,72℃延伸30 s,35个循环后72℃延伸2 min。同时,以18S rRNA为内参,PCR程序同上,循环数改为30。PCR反应结束后,分别取5 μL扩增产物进行w=1.5%琼脂糖凝胶电泳,用AlphaImager EP(Alpha Innotech,USA)凝胶成像系统对电泳图像进行拍照和分析。
2 结果
2.1 斑马鱼胰蛋白酶原cDNA克隆
以斑马鱼肝脏总RNA为模版,首先进行斑马鱼胰蛋白酶原基因保守区域的克隆,得到一条428 bp的DNA片段,通过测序我们得到两条相似的序列。然后针对这两个序列的差异设计特异性引物分别进行3'-RACE和5'-RACE扩增。把各得到3个序列进行拼接得到两条斑马鱼胰蛋白酶原cDNA全长序列,分别命名为zftry1a和zftry1b。zftry1a cDNA全长891 bp,包含一段编码242个氨基酸残基的729 bp开放读码框 (ORF),一段53 bp的5'非编码区 (5'-UTR)和一段109 bp的3'-UTR(Gen-Bank登录号:JQ999996)。zftry1b cDNA全长858 bp,其中包含一段编码242个氨基酸残基的729 bp ORF,一段36 bp的5'-UTR和一段93 bp的3'-UTR(GenBank登录号:JQ999997)。
以人胰蛋白酶原基因和zftry1a或zftry1b蛋白序列作为种子序列,选择tblastn比对工具在斑马鱼基因组中进行同源比对,结果搜索到一条同源性较高的基因 (ENSDART00000077661)。根据设计的特异性引物在斑马鱼肝脏中克隆得到了胰蛋白酶原2(zftry2)的ORF全长。zftry2 cDNA ORF全长744 bp,编码247个氨基酸残基 (GenBank登录号:JQ999998)。
2.2 斑马鱼3种胰蛋白酶原的基本特征分析
预测的 zftry1a和 zftry1b相对分子质量均26400,等电点PI分别为4.94和4.93,预测的信号肽由15个氨基酸组成,为 MRSLVFLVLL-GAAFA,激活肽含有5个氨基酸残基,为LDDDK。zftry1a和zftry1b在PH 7.0下带电荷量分别为-8.608和-6.944。预测的zftryp2相对分子质量26500,PI为7.46,15个氨基酸组成的信号肽MKAFILLALFAVAYA,激活肽由9个氨基酸残基组成,为APLGDDDDK。在PH 7.0下带电荷量+1.209。
2.3 氨基酸序列比对和同源性分析
氨基酸序列比对分析发现 (图1),斑马鱼3种胰蛋白酶原均具有胰蛋白酶原的保守结构特征:胰蛋白酶催化活性必需的高度保守的催化三联体氨基酸 (His-57,Asp-102和Ser-195);位于底物结合口袋底部的Asp-189和口袋开口处的Gly-216、Gly-226;具有构成6个二硫键所需的12个半胱氨酸残基;决定结合底物特异性的一个关键氨基酸Tyr-172;丝氨酸蛋白酶的典型特征序列即位于催化活性部位丝氨酸附近的氨基酸 (Gly-Asp-Ser-Gly-Gly-Pro)等。
利用MegAlign进行同源性分析显示,zftry1a和zftry1b的同源性高达96.7%,zftry2与zftry1a和zftry1b的同源性为64.9%。zftry1a和zftry1b与草鱼、青鳉、牙鲆、日本鳗鲡和金头鲷的同源性(77.3%~88.8%)高于 zftry2与它们的同源性(60.7%~67.4%),而zftry1a和zftry1b与翘嘴鲌、石斑鱼和胡瓜鱼的同源性 (64.9%~66.9%)低于zftry2与它们的同源性 (78.8%~89.9%),zftry1a和zftry1b与大西洋鳕鱼和红鳍东方鲀的同源性与zftry2与它们的同源性接近,均低于与上述鱼类的同源性 (表2)。

图1 斑马鱼与其它部分物种的胰蛋白酶原的氨基酸序列比对Fig.1 Alignment of the deduced amino acid sequences of zebrafish trypsinogens and some other vertebrate trypsinogens deposited in Gen Bank
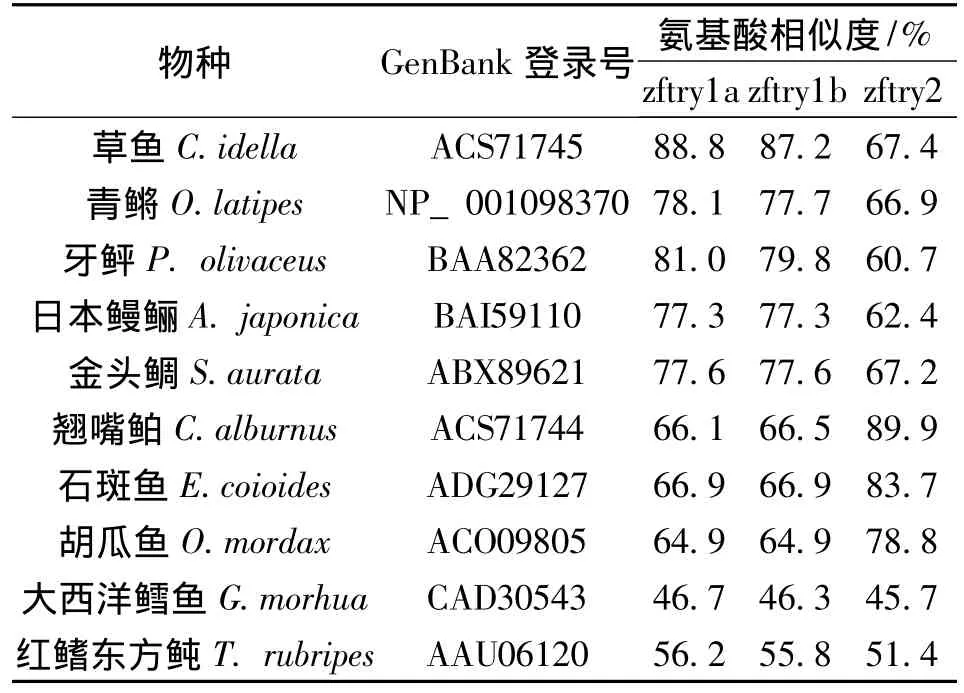
表2 斑马鱼3种胰蛋白酶原与其它部分鱼类的氨基酸相似度Table 2 Amino acid sequence percent identity of zebrafish trypsinogens campared to several fish
2.4 系统进化分析
从GenBank上获得已知的脊椎动物胰蛋白酶原的氨基酸序列经ClustalW方法比对后,利用Mega3.1以邻位相接 (N-J)构建系统进化树 (图2)。由图可以得出鱼类胰蛋白酶原分为三支(Group I、II、III),按照Roach et al(2002)有关脊椎动物胰蛋白酶原的聚类方法[13],斑马鱼zftry1a和zftry1b位于硬骨鱼类group I,属于阴离子型胰蛋白酶原。斑马鱼zftry2位于group II,属于阳离子型胰蛋白酶原。
2.5 斑马鱼3种胰蛋白酶原基因的组织表达
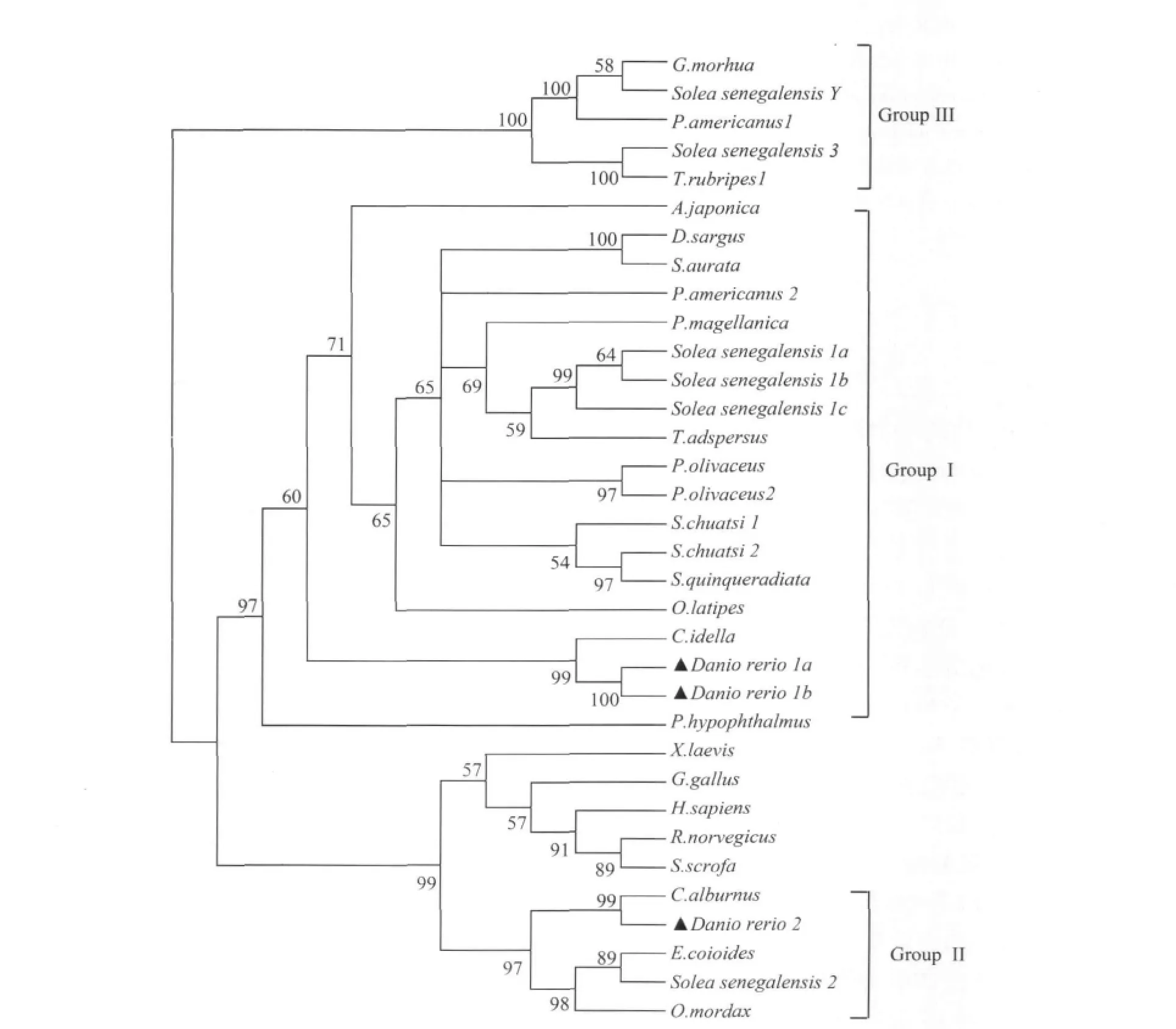
图2 胰蛋白酶原的进化树分析Fig.2 Phylogenetic tree based upon the alignment of amino acid sequences from a wide range of vertebrates
采用RT-PCR方法,以18S作为内参,对3种胰蛋白酶原基因在成年斑马鱼脑和6个外周组织中的表达进行了检测。结果如图所示 (图3),3种胰蛋白酶原基因表达分布基本类似,在肠中的表达量均显著高于其他组织,特别是zftry1a和zftry1b。同时,在脾脏中没有检测到三者基因的表达。但是,zftry1a和zftry1b不同的是,除在脾脏没有检测到表达之外,在肌肉中也没有检测到其mRNA分布。
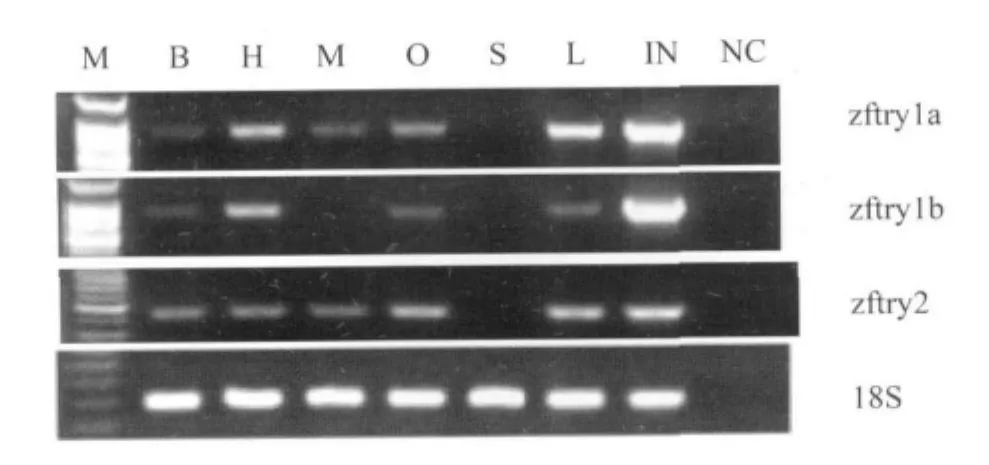
图3 RT-PCR方法检测胰蛋白酶原基因在成年斑马鱼各组织中的表达Fig.3 RT-PCR analysis of the expression of trypsinogens mRNA in the adult zebrafish tissues.
3 讨论
本文利用RT-PCR和RACE技术,结合基因组数据库,成功克隆得到了编码斑马鱼3种胰蛋白酶原的cDNA序列,并对其蛋白结构、同源性和进化关系以及组织表达进行了研究。根据蛋白质序列以及同源性分析主要归为两类:zftry1(zftry1a和zftry1b)和zftry2。分析发现,斑马鱼3种胰蛋白酶原均具有:催化活性必需的高度保守的催化三联体氨基酸 (His-57,Asp-102和 Ser-195);位于底物结合口袋底部的Asp-189和口袋开口处的Gly-216、Gly-226;具有构成6个二硫键所需的12个半胱氨酸残基;决定结合底物特异性的一个关键氨基酸Tyr-172;丝氨酸蛋白酶的典型特征序列即位于催化活性部位丝氨酸附近的氨基酸 (Gly-Asp-Ser-Gly-Gly-Pro)。功能氨基酸以及基序的一致性表明斑马鱼3种胰蛋白酶原功能的保守性。Hedstrom等[14]研究发现丝氨酸蛋白酶所形成的两个口袋(S1和S2)对底物的结合至关重要。比对发现,斑马鱼3种胰蛋白酶原形成S1口袋的氨基酸组成完全相同,但是组成S2口袋的氨基酸略有差异。这些结果提示,斑马鱼3种胰蛋白酶可能有不同的底物选择特异性。
胰蛋白酶原被合成和分泌后,在小肠中存在Ca2+情况下被肠激酶切去激活肽转变成有活性的胰蛋白酶[2]。肠激酶能够识别包含一簇阴离子氨基酸残基的激活肽[15]。在大部分鱼类中激活肽包含3个酸性氨基酸 (D或E),而在哺乳动物中则是连续4个天冬氨酸[1]。我们的氨基酸序列比对发现,斑马鱼zftry1和zftry2的激活肽长度和组成氨基酸不同。zftry1a和zftry1b激活肽含有3个酸性氨基酸残基簇 (DDD),而zftry2与哺乳动物类似,含有4个天冬氨酸组成的激活肽。这与我们进化分析的结果相一致,zftry1a和zftry1b归为group I,属于阴离子型胰蛋白酶;斑马鱼zftry2位于group II,属于阳离子型胰蛋白酶。这些不同物种的激活肽序列及其组成的差别可能是由于在进化过程中选择性压力造成的结果[16]。
目前,在海水鱼类冬鲽[10]和塞内加尔鳎[11]中分别已克隆得到2和6种编码胰蛋白酶原的cDNA序列。目前在淡水鱼类中还没有发现多种胰蛋白酶原基因。本文通过RT-PCR结合基因组数据库成功克隆到3种斑马鱼胰蛋白酶原基因。组织表达分析发现,3种胰蛋白酶原基因在肠中表达量最高,提示其主要具有消化功能。另外,尽管同属阴离子型,zftry1a和zftry1b的组织表达分布也略有差异,zftry1a在肌肉有表达,而zftry1b则无,这表明二者在组织中可能存在功能差异。一个有意思的结果是,在脑中均检测到了三者的表达,且zftry2的表达量要高于zftry1a和zftry1b的表达量。这与在塞内加尔鳎中得到的结果相一致[11]。尽管6种塞内加尔鳎胰蛋白酶原基因 (ssetryp1,ssetryp1a,ssetryp1b, ssetryp1c, ssetryp2, ssetryp3, ssetrypY)在脑中都有表达,但是以ssetryp2的表达量为最高,分别是ssetryp1和ssetrypY表达量的8.7和69.3倍。因为zftry2和ssetryp2同属阳离子性胰蛋白酶,是否阳离子型胰蛋白酶在脑中有着特殊的功能还需要进一步研究。总之,胰蛋白酶原基因组织表达的广泛性以及在同一组织中各基因表达量的差异表明胰蛋白酶除了扮演主要的消化功能外,在神经系统[17]、生殖系统[18]等都具有一定的功能。
一般而言,鱼类蛋白质消化酶和代谢酶活性越高,其对营养物质的消化吸收和利用的能力越强,从而提高其生长发育的速度。目前对胰蛋白酶的研究还主要集中在基本特性分析和基因克隆上,对相关营养物质对胰蛋白酶的表达调控研究相对较少。王桂芹等最近研究认为饲料能量和维生素B6对肝脏蛋白酶活性有影响,但是否对胰蛋白酶的表达调控有无影响还未知[19]。要想进一步了解斑马鱼3种胰蛋白酶原基因的表达调控机制,启动子序列的克隆则是必须的。
总之,本文克隆得到了3种斑马鱼胰蛋白酶原基因。氨基酸序列比对和进化树分析表明,三者在功能上是保守的,且zftry1a和zftry1b属于阴离子型胰蛋白酶原,zftry2属于阳离子型胰蛋白酶原。RT-PCR结果显示,三者的组织表达模式略有不同,提示它们的功能既有共性又有特异性。
[1]RUAN Guoliang,LI Yang,GAO Zexia,et al.Molecular characterization of trypsinogens and development of trypsinogen gene expression and tryptic activities in grass carp(Ctenopharyngodon idellus)and topmouth culter(Culter alburnus)[J].Comp Biochem Physiol B Biochem Mol Biol,2010,155(1):77 -85.
[2]王镜岩,朱圣庚,徐长发.生物化学[M].3版.北京:高等教育出版社,2002:405.
[3]CHEN J M,KUKOR Z,LEMARECHAL C,et al.Evolution of trypsinogen activation peptides[J].Mol Biol Evol,2003,20(11):1767 -1777.
[4]RUST M B.Nutritional physiology[M]//HALVER J E,HARDY R W.Fish nutrition.3rd ed.Amsterdam:Elsevier,2002:367 -452.
[5]RYPNIEWSKI W,PERRAKIS A,VORGIAS C E,et al.Evolutionary divergence and conservation of trypsin[J].Protein Eng,1994,7(1):57-64.
[6]GUDMUNDSDOTTIR A,GUDMUNDSDOTTIR E,OSKARSSON S,et al.Isolation and characterization of cDNAs from Atlantic cod encoding two different forms of trypsinogen[J].Eur J Biochem,1993,217(3):1091-1097.
[7]SUZUKI T,SRIVASTAVA A S,KUROKAWA T.cDNA cloning and phylogenetic analysis of pancreatic serine proteases from Japanese flounder,Paralichthys olivaceus[J].Comp Biochem Physiol B Biochem Mol Biol,2002,131(1):63-70.
[8]GENICOT S,RENTIER-DELRUE F,EDWARDS D,et al.Trypsin and trypsinogen from an Antarctic fish:molecular basis of cold adaptation[J].Biochim Biophys Acta,1996,1298(1):45-57.
[9]KUROKAWA T,SUZUKI T,OHTA H,et al.Expression of pancreatic enzyme genes during the early larval stage of Japanese eel Anguilla japonica[J].Fish Sci,2002,68(4):736-744.
[10]DOUGLAS S E,GALLANT J W.Isolation of cDNAs for trypsinogen from the winter flounder,Pleuronectes americanus[J].J Mar Biotechnol,1998,6(4):214 -219.
[11]MANCHADO M,INFANTE C,ASENSIO E,et al.Molecular characterization and gene expression of six trypsinogens in the flatfish Senegalese sole(Solea senegalensis Kaup)during larval development and in tissues[J].Comp Biochem Physiol B Biochem Mol Biol,2008,149(2):334-344.
[12]PSOCHIOU E,SARROPOULOU E,MAMURIS Z,et al.Sequence analysis and tissue expression pattern of Sparus aurata chymotrypsinogens and trypsinogen[J].Comp Biochem Physiol B Biochem Mol Biol,2007,147(3):367-377.
[13]ROACH J C.A clade of trypsins found in cold-adapted fish[J].Proteins,2002,47(1):31 -44.
[14]HEDSTROM L,SZILAGYI L,RUTTER W J.Converting trypsin to chymotrypsin:the role of surface loops[J].Science,1992,255(5049):1249-1253.
[15]LOUVARD M N,PUIGSERVER A.On bovine and porcine anionic trypsinogens[J].Biochim Biophys Acta,1974,371(1):177-185.
[16]ROACH J C,WANG K,GAN L,et al.The molecular evolution of the vertebrate trypsinogens[J].J Mol Evol,1997,45(6):640-652.
[17]KOSHIKAWA N,HASEGAWA S,NAGASHIMA Y,et al.Expression of trypsin by epithelial cells of various tissues,leukocytes,and neurons in human and mouse[J].Am J Pathol,1998,153(3):937 -944.
[18]MIURA C,OHTA T,OZAKI Y,et al.Trypsin is a multifunctional factor in spermatogenesis[J].Proc Natl Acad Sci U S A,2009,106(49):20972-20977.
[19]王桂芹,李子平,牛小天,等.饲料能量和维生素B6对乌鳢生长和蛋白质代谢酶活性的影响[J].中山大学学报:自然科学版,2012,50(4):96-99.
