半套式RT-PCR检测动物狂犬病病毒的研究
2013-11-22袁颖烁赵敬慧宋菲菲张守峰张乐萃扈荣良
袁颖烁,赵敬慧,宋菲菲,刘 晔,张守峰,张乐萃,扈荣良
(1.青岛农业大学动物科技学院,山东 青岛 266109;2.军事医学科学院军事兽医研究所 吉林省人兽共患病预防与控制重点实验室,吉林 长春 130122)
狂犬病(Rabies)是由狂犬病病毒(RV)感染引起的高致死性人兽共患传染病,人和所有温血动物对狂犬病病毒都易感,一旦发病,死亡率几乎为100%,严重威胁人畜的健康。我国是受狂犬病危害最为严重的国家之一。不同毒株的狂犬病病毒N蛋白基因序列具有较高的保守性、同源性和高拷贝复制的特点,因此可用于狂犬病的分型、诊断、流行病学分析调查研究等。
目前可以通过检测组织中的病毒、病毒蛋白(抗原)或基因组RNA(核酸),比较肯定地对狂犬病作出诊断。具体方法包括:电镜直接观察病毒颗粒状态、病毒的体内或体外分离培养、通过标记的荧光抗体或间接染色的方法检测病毒蛋白、反转录-聚合酶反应检测病毒的RNA等。另外,狂犬病病毒感染的间接证据,可通过检测未免疫血清中的病毒中和性抗体或脑脊髓液中存在的抗体确定[1-2]。其中,RT-PCR方法不仅可以检测细胞培养的RV,还可以直接用于临床样品的检测,已广泛应用于狂犬病的实验室诊断和流行病学调查,具有很好的应用前景。本研究针对我国狂犬病病毒均属基因1型,且易感动物种类多、流行毒株可能存在多样性分布的特点,以N基因保守区为靶序列设计了两对引物,建立了一种特异性好、灵敏度高、简便快速的半套式RT-PCR方法。
1 材料与方法
1.1 主要试剂 Trizol Reagent RNA提取试剂盒(Invitrogen);One Step RNA PCR Kit(AMV)(TaKaRa),EX-Taq酶等试剂,均购自宝生物工程(大连)有限公司;FITC标记的狂犬病病毒核蛋白单克隆抗体,由军事兽医研究所流行病学研究室制备。
1.2 主要耗材和仪器 TGRANDIENT PCR仪,购自 HYBAID公司;PCR管,购自AXYGEN公司;VI Firereader XS凝胶成像系统,购自华粤行公司;DYFⅢ2型电泳仪,购自Bio-Rad公司。
1.3 毒株、乳鼠及病料 CVS-11、SRV9、ERA、Flury-LEP细胞毒、CVS-24脑毒以及23株RABV野外分离株(*)均为本实验室保存,其他临床病料信息见表2;新生ICR乳鼠(清洁级),由吉林大学医学部实验动物中心提供。
1.4 半套式RT-PCR检测方法的建立
1.4.1 引物设计与合成 参考GenBank公布的RV N基因全序列进行同源性比较分析,选取序列中保守的区域,根据RT-PCR引物设计原则,利用Primer5.0软件设计了2对半套式RT-PCR引物。(引物信息见表1)外套引物(NF/NR1)扩增片段长874bp,内套引物(NF/NR2)扩增片段长508bp。引物由宝生物工程(大连)有限公司合成,使用时用DEPC水稀释至10μmol/L。
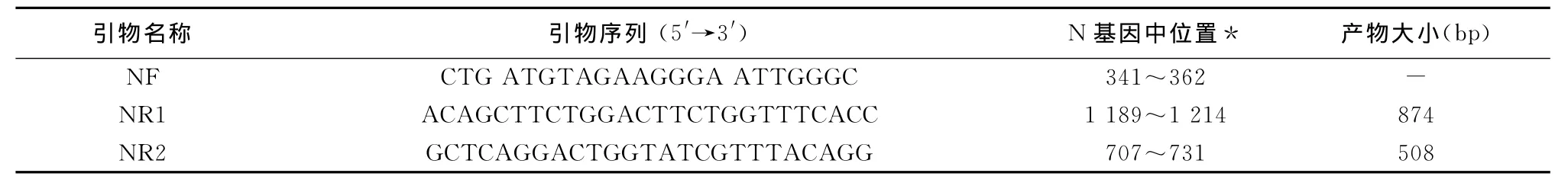
表1 引物序列
1.4.2 细胞毒和临床病料总RNA的提取 参考Trizol Reagent RNA提取试剂盒说明书操作。
1.4.3 一步法RT反应及外套RT-PCR反应 参照One Step RNA PCR Kit(AMV)的说明书进行,将提取好的总RNA沉淀中加入23μL的Rnase Free H2O使之溶解后,直接加入如下体系的PCR管中:10×One Step RNA PCR Buffer 5.0μL,MgCl2(25mmol/L)10μL,dNTP Mixture(各 10 mmol/L)5.0μL,RNase Inhibitor(40U/μL)1μL,AMV RTase XL(5U/μL)1μL,AMV-OptimizedTaq(5U/μL)1μL,上游引物 NF(10μmol/L)2 μL,下游引物 NR1(10μμmol/L)2μL,总体积50 μL。经50℃水浴30min后,直接进行外套PCR反应,也可立即放置-70℃保存备用。外套PCR循环步骤为:94℃,3min→(94℃,30s→59℃,30s→72℃,40s)30个循环→72℃,8min延伸。
1.4.4 半套式 RT-PCR 取2μL PCR外套扩增产物为模板,建立50μL内套PCR反应体系如下:PremixTaq25μL,上游引物NF(10μmol/L)2μL,下游引物 NR2(10μmol/L)2μL,2ddH2O 19μL。内套PCR循环步骤为:94℃,3min→(94℃,30s→64℃,30s→72℃,30s),30个循环→72℃,7min延伸。
1.5 灵敏度试验 将SRV9弱毒株细胞培养物(约10-4.25TCID50/mL)用0.01mol/L 1×PBS(pH 值7.4)进行1×10-1~1×10-1010倍系列稀释,分别取200μL按照上述方法提取总RNA后进行PCR检测。
1.6 特异性试验
1.6.1 非狂犬病病毒的检测 采用上述方法对犬瘟热病毒(CDV)、犬细小病毒(CPV)、犬腺病病毒(CAV)、伪狂犬病病毒(PRV)等几种非狂犬病病毒进行RNA或DNA提取,首先用病毒提供者建议的RT-PCR或PCR方法对各病毒进行鉴定,,并设阴性对照,鉴定成功后再进行半套式RT-PCR检测,以验证该方法的特异性。其中,CPV、CAV、PRV为DNA病毒,可直接进行半套式RT-PCR,CDV为RNA病毒,检测步骤与RV完全相同。
1.6.2 不同动物脑组织的检测 用该方法对犬、鼬獾(江西)、黄鼠狼等3种不同易感的脑组织样品进行检测,并与FAT试验相比较,以确定所建立方法对狂犬病病毒易感动物检测的特异性(样品信息见表2)。

表2 检测用不同动物脑组织的背景信息及试验
1.7 敏感性试验 分别对 CVS-11、SRV9、ERA、Flury-LEP、CVS-24和本实验室保存的23株野外分离株脑毒进行半套式RT-PCR和FAT同步检测,验证引物的敏感性及两者符合情况。
2 结果
2.1 半套式RT-PCR扩增结果 在优化了PCR条件的基础上,本研究建立了一种以N基因为靶序列的半套式RT-PCR方法。结果显示,阳性样品可扩增出特异性目的片段,且背景清晰,无拖带现象,而阴性对照无特异条带。
2.2 灵敏度试验 经外套扩增可见到大小为874 bp的特异性目的片段;经内套扩增后,1×10-3倍稀释的细胞毒也可见到508bp的特异扩增产物,即对SRV9细胞毒的检测灵敏度可达到0.01个TCID50/mL(图1)。
2.3 特异性试验
2.3.1 非狂犬病病毒的检测 针对犬瘟热病毒(CDV)、犬细小病毒(CPV)、犬腺病病毒(CAV)、伪狂犬病病毒(PRV)等非狂犬病病毒的RT-PCR或者PCR方法检测均扩增出阳性目的片段,阴性对照成立,而用上述方法病毒样品均未表现阳性结果。
2.3.2 不同动物脑组织的检测 对犬、鼬獾、黄鼠狼等样品进行检测,结果均有特异性目的条带出现。24例免疫荧光抗体试验(FAT)确定为阳性的样品,在半套式RT-PCR中均为阳性;FAT为阴性的样品,在半套式RT-PCR中2例为扩增阳性,将样品进行小鼠脑内接种试验(MIT),取死亡小鼠脑做FAT试验,结果呈阴性;将脑组织样品在37℃放置72h后,用半套式RT-PCR法仍可检测到阳性结果。新鲜样品检测为阴性的样品,以同样条件放置后仍为扩增阴性(见图2、表2)。

图1 半套式RT-PCR检测SRV9细胞培养物的电泳图

图2 半套式RT-PCR检测RV易感动物脑组织的电泳
2.4 敏感性试验 分别对 CVS-11、SRV9、ERA、Flury-LEP细胞毒和CVS-24脑毒及本实验室保存的23株野外分离株脑毒进行半套式RT-PCR检测及脑毒FAT同步检测,均获得阳性结果,敏感性较高。
3 讨论
本试验针对基因Ⅰ型狂犬病毒(RV)并偏顾于我国固定毒株和流行性毒株,设计2对引物,建立了一种特异检测RV的半套式RT-PCR方法,该方法不仅可以检测细胞培养的RV,还可以直接用于临床样品的检测。世界卫生组织推荐FAT为诊断人和动物狂犬病快速而敏感的金标方法[3],但该方法也有其局限性。例如,它需要荧光显微镜的支持且耗时较长[4],并且结果受抗体质量的影响很大,病毒细胞培养则存在病变是否产生的问题[5],另外,它对样本的新鲜度有一定要求,在腐败样品的检测中敏感度下降,易出现假阴性,且在暗环境中进行大量样品检测时,工作人员易产生视觉疲劳,从而造成误判、漏判。本试验所建立的半套式RT-PCR方法特异性和敏感性均较高,检测结果直观,更适合大量样品的检测工作,且基本克服了FAT对腐败样品检测的问题。值得一提的是,考虑到多样品检测时半套式RT-PCR可能存在的漏检和交叉污染现象,本研究采用一步法RT-PCR,反转录和PCR均在同一管中进行热循环,无需打开管盖,操作简便快速,避免了批量样品检测时存在的的交叉污染问题[6]。本研究设计的引物扩增片段含盖了不同毒株间N基因变异最大区域[7],有助于狂犬病的分子生物学诊断和流行病学调查分析。本研究对36份脑组织样品分别进行了检测,共检出23份阳性脑组织,与FAT检测结果完全一致。该方法能从腐败样品中检出病毒RNA,更适用于腐败样品的检测,条件优化后,RNA提取到获得结果仅用5h即可完成狂犬病的检测,可用于我国狂犬病临床样品的定性检测和大规模流行病学调查研究,为控制狂犬病工作提供有力的技术保障。
[1] Meslin F X,Kaplan M M,Koprowski H,eds.Laboratory techniquesinrabies4thed Geneva World Health Organization,1996.
[2] 扈荣良.狂犬病理论、技术与防治[M].北京:科学出版社,2007:92.
[3] WHO Expert Committee on Rabies Ninth report Geneva World Health Organization[J].2004(WHO Technical Report Series,No.931).
[4] Woldehiwet Z.Clinicallaboratoryadvancesinthedetectionof rabiesvirus[J].Clin Chim Acta,2005,351(1-2):49-63.
[5] Poschl B.Waneesorn J,Thekisoe O,etal.Comparative diagnosis of malaria infections by microscopy,nested PCR,and LAMP in northern Thailand[J].Am J Trop Med Hyg,2010,83(1):56-60.
[6] Gupta P K,Singh R K,Sharma R N,etal.Preliminary report on a single-tube non-interrupted reverse transcription-polymerase chain reaction for the detection of rabies virus in brain tissue[J].Veterinary ResearchCommunications,2001,25(3):239-247.
[7] Kissi B,Tordot N,Bourhy.Genetic polymorphism in the Rabies virus nucleoprotein gene [J].Virology,1995,209:526-537.
