假基因鉴定及其功能分析
2013-11-12刘慧邹枨林凤
刘慧,邹枨,林凤
1 沈阳农业大学生物科学技术学院,辽宁 沈阳 110866
2 中国农业科学院作物科学研究所,北京 100081
1977年,Jacq等[1]通过单基因克隆的方法获得了与功能基因相似性非常高但却不能行使功能的基因,这种序列首次被发现并被定义为假基因。随着测序技术的飞速发展,许多物种全基因组序列陆续被发表,这为基因组范围内假基因的鉴定奠定了基础。近年来,许多科研小组根据物种基因组数据对假基因进行了鉴定,以人类为例:Yao等[2]依据人类全基因组转录及蛋白序列鉴定了2 011个假基因并进行了分类,同时找到加工及未加工假基因,并在此基础上鉴定了转录的假基因。Molineris等[3]在人类基因组中鉴定获得2 288个加工假基因。Zhang等[4]对人类及其近缘物种的基因库进行分析,鉴定了 76个单一假基因 (Unitary pseudogenes)。
另外,假基因的功能研究也取得了重大的进展[5],例如与功能基因 pou5f1相似性达到 97%的假基因 pou5f1p1可以在细胞系和结肠癌组织中表达,其功能与肿瘤形成有关[6]。除此之外,假基因还能够通过生物体内小RNA产生关键性的调节作用:如通过产生siRNA进行干扰以调控基因的表达;又如假基因作为miRNA的靶位点,通过与功能基因靶位点竞争miRNA的结合调控功能基因的表达。因此,对假基因功能的探索成为研究基因表达调控的一个重要内容。本文围绕近年来假基因鉴定和假基因功能两个研究热点进行较全面的介绍。
1 概述
1.1 假基因定义及产生方式
假基因的概念最初由 Jacq等[1]克隆 1个5S rRNA相关基因时提出:由于基因序列的5′端缺失或错配使这个截短的5S rRNA丧失功能,并将其描述为假基因。后来研究发现,假基因大多是由于存在提前终止子或移码突变而丧失了正常编码蛋白的功能[7]。所以,目前,公认的假基因定义表述为与已知功能基因组DNA有很高的序列相似性,但由于某些遗传缺陷造成不能正常表达的基因序列。
对于假基因的产生,目前认为主要存在以下两种方式:一种是DNA复制过程中,由于碱基突变产生移码突变或提前终止子进而形成假基因,被称为未加工方式;另一种通过反转录转座作用获得,即DNA转录为mRNA后,再由mRNA反转录成cDNA,然后cDNA随机插入到基因组位点后形成加工后假基因,又被称为逆转座型假基因。另外,由于启动子丧失功能使一些低表达的基因逐渐假基因化 (Pseudogenization),也可以形成假基因[8]。
1.2 假基因在染色体上的数量及分布
长期以来,人们认为假基因是存在于生物体中无功能的“死亡基因”,是基因组进化过程当中的“化石”[9]。人们对生物体内这一“垃圾基因”进行了研究和分析,到目前已经在拟南芥、果蝇、斑马鱼、小鼠、人类等物种中获得了假基因序列信息 (表1),下面列出几种模式物种体内假基因含量。
从图中我们可以发现:人类和小鼠基因组包含大约 22 000个已注释的蛋白编码基因和分别大约 17 000个和 19 000个假基因。果蝇D. melanogaster中有大约14 000个蛋白编码基因,假基因只有2 200个。可能由于果蝇中具有高基因组消亡率[10],而造成了其功能基因与假基因比例与其他物种相差较大。植物中已获得假基因数量的主要为水稻和拟南芥,分别含有5 600个和 2 700个假基因[11-12],将其与水稻基因组487 Mb——大约45 000基因 (International Rice Genome Sequencing Project,2005)以及拟南芥基因组135 Mb——大约27 411基因 (http://www.arabidopsis.org,TAIR10,2010)比较可知,基因组大小不同可能是造成两者假基因差距较大的原因。Podlaha等[13]对假基因数量进行了深入研究后发现,决定假基因数量差异的因素主要是不同物种内基因的产生率和消亡率。假基因产生率决定于DNA和RNA发生变化的概率;而消亡率主要决定于进化当中中性突变及清除速率。除了决定性因素外,影响假基因尤其是加工假基因产生的还有表达组织和表达量两个因素。新产生的假基因需要隶属于生殖细胞系或胚胎干细胞(其可产生生殖细胞系),而仅仅在体细胞中表达的基因不能产生加工假基因。在表达量方面,管家基因等高表达的基因由于能够被逆转录插入的 mRNA分子很多,因而有更大产生逆转录转座子的可能性。研究发现能够产生最多假基因的基因类型有:核糖体蛋白、DNA及RNA结合蛋白、特定结构蛋白和代谢酶等[13],例如:人类核糖体蛋白被大约 80个基因编码,有大约 2 000个假基因[14]。除上面因素外,还有两种情况也会产生假基因,一种为上游调节区域的突变使开放阅读框表达衰退进而形成假基因,例如:Yang等[8]对拟南芥中已注释的蛋白编码基因进行分析,鉴定了1 939个几乎没有表达证据但已被注释为编码蛋白的基因,这些基因普遍比表达基因短,非同义突变概率高出正常基因2倍,上游序列趋异的概率也比表达基因高,说明转座元件的插入等因素使得这些低表达的基因通过启动子退化作用走上假基因化道路;另一种为突变作用,其产生也会影响假基因的产生率,尤其是一个物种在生态上突然变化造成很多有功能的基因变得无功能。

表1 不同物种假基因数量分布Table 1 Pseudogene distribution in different species
对于假基因在染色体上的分布,不同物种有较大区别:酵母Saccharomyces cerevisiae H. 基因组中98个假基因有44%分布于近端粒区域,着丝粒附近较少[15];果蝇 D. melanogaster大部分假基因分布在着丝粒附近。而对人类的研究结果却表明,近端粒和着丝粒处的加工假基因数目都比较少[16],这可能分别与端粒附近易发生基因重组和 DNA置换,而着丝粒附近拥有较低 GC含量有关[17]。刘国庆等[18]以加工假基因为例,研究假基因在染色体上分布的影响因素,发现基因分布、重组率和GC含量3个因素较为关键且影响程度依次减弱。加工假基因分布与基因分布为正相关,即较多地分布在基因密区;重组率对加工假基因分布有负相关作用。而对于GC含量,它与重组率、基因密度等变量之间互相关联。去掉重组率和基因密度影响时,加工假基因密度和 GC含量间的关系会由原先的正相关变为负相关,这种负相关性体现在较长加工假基因上(>400 bp),而短加工假基因的密度与 GC含量之间却没有显著相关性。
2 假基因鉴定流程
全基因组范围内进行假基因鉴定的操作流程主要包括 PseudoPipe、PseudoFinder、RetroFinder、REGEXP 等[3,19]。PseudoPipe 是一种基于同源性搜索全面鉴定假基因的方法,过程主要通过本地 Blast[20]找能够匹配到蛋白序列的基因组序列,然后去掉已注释的编码基因序列和重复冗余序列 (http://www.girinst.org/repbase/index.html),将同一方向得分最高的蛋白序列根据内含子大小特点进行合并[12],再通过同源性、内含子-外显子结构、提前终止子或移码突变等假基因特征判断序列中造成编码缺陷的突变类型和位置,进而鉴定得到假基因。Zheng等[21]以PseudoPipe作为基础进行了延伸:在获得候选假基因片段后,通过寻找外显子-内含子剪接处,依据剪接处对假基因进行分类 (PseudoPipe鉴定流程如图 1所示)。Zou等[12]在鉴定拟南芥和水稻的过程中也利用上述方法,通过对比假基因与蛋白序列寻找外显子-内含子剪接位置,计算剪接位置的个数,通过个数不同划分其为加工、未加工假基因以及假基因片段区。
除了PseudoPipe,其他科研小组也根据假基因特点提出了自己的鉴定流程:由加利福尼亚大学发表的 PseudoFinder方法[22],通过同源匹配(Homologous mapping) 鉴定了人类当中的假基因。此方法利用已知的人类基因组序列作为参考序列[23],通过HomoMap找到参考序列的同源序列片段,将这些片段进行连接后再与已知参考序列进行比对,得到一致性分数、提前终止子的数量等一系列结果。利用Support Vector Machines(SVMs)从所有结果中挑出阳性样本 (与已知的假基因匹配的基因)和阴性样本 (与参考基因有重叠的基因)并做标记。最后将没有显著功能、具有多项假基因特征的片段保留,将假基因特征不明显、证据不充足的部分去掉获得最终假基因。PseudoFinder准确度较高,通过10X交叉验证测试结果表明此方法比其他方法更有效。
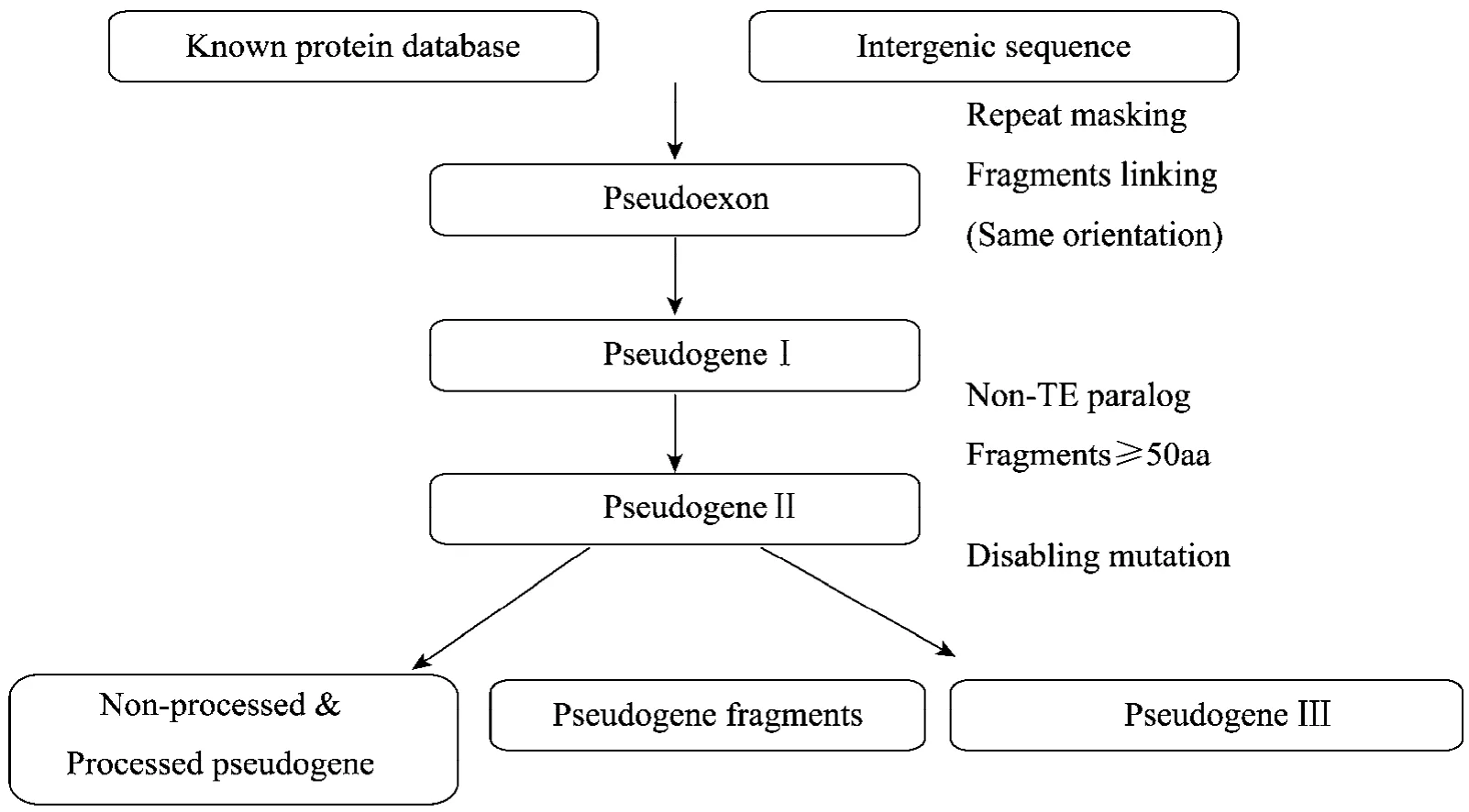
图1 假基因鉴定流程图[12]Fig. 1 Pseudogene Identification pipeline[12].
同样由加利福尼亚大学发表的 RetroFinder方法专注于加工假基因的注释[22]。其首先从GenBank中检索人类所有 mRNA序列,通过Blastz将序列比对到基因组上[23]。每一个序列通过分析其序列位置、剪接位点个数、重复元件覆盖度以及外显子个数等生物特征,获得一个基于每个位点逆转录转座子发生可能性的分数。通过研究已知的假基因推断一个阈值,并根据阈值鉴定加工假基因。
上述鉴定方法虽然依赖于不同的流程,但都需要物种的基因组、转录组以及蛋白组信息,这些流程对于已知生物 EST及蛋白信息的物种很有效,但对非模式生物的假基因鉴定比较困难。Molineris等[3]提出了新的鉴定加工假基因方法REtrotransposed Gene EXPlorer (REGEXP)。REGEXP仅仅依赖DNA序列,不依赖mRNA、EST或蛋白信息,从而对于转录组注释缺乏的物种假基因鉴定有很重要的意义。其中心思想是编码基因和加工假基因能够通过一系列的两两同源基因比对 (Pairwise paralogous alignments)找到高分数对 (High score pairs,HSPs),根据加工假基因仅含有原始基因外显子的特点,寻找HSPs附近的簇,假基因对应的HSPs互相非常接近,而正常同源基因虽然较近,但却被内含子分割,依照此特点构建假基因数据库,又因一个祖先基因可以得到多个加工假基因,后续还包括对其分析并找到对应的原始唯一的祖先基因。为了降低假阳性以及对祖先基因不完整注释的概率,还需要至少 3个剪接缺口 (Splicing gaps)以获得 1个候选序列。将得到的结果与 Ensemble、VEGA以及Pseudogene.org上的假基因数据进行假基因比对,有很好的一致性。
除了针对生物体内普遍假基因鉴定的方法外,Zhang等[4]指出了一种新的鉴定无功能且无配对祖先基因——单一假基因 (Unitary pseudogenes)的方法。过程首先将人类与小鼠同源的基因找到,之后与小鼠的蛋白序列比对,找出不在同源基因内的小鼠蛋白序列后,将这些序列与人类基因组比对,能够匹配上的作为假基因候选序列,根据内含子与小鼠基因是否一致以及是否具有假基因的提前终止子、移码突变等特征进而鉴定人类假基因。利用获得的鉴定结果,Zhang等还比较了两个物种整体详细的直系同源基因信息,结合灵长类单一假基因的年龄,得到较早产生功能的基因在灵长类进化过程中不是突然消失功能,而是有规律性变化的结论。
结合之前假基因鉴定的方法和结果,Pei等[24]系统地进行了人类假基因的鉴定以及基于鉴定结果进行的表达水平、转录因子、RNA聚合酶Ⅱ结合位点以及染色质标记分析。其在过程中利用 HAVANA小组以及 PseudoPipe和RetroFinder获得的结果,在人类全基因组范围内鉴定了11 216个假基因以及138个单一假基因。同时,实验找到9 368个假基因对应的亲本基因3 391个 (1 848个假基因由于注释不精准得不到其亲本基因),其中2 071个仅对应1个假基因,另外一些如核糖体蛋白 (Ribosomal protein L21,RPL21)对应143个假基因,甘油醛-3-磷酸盐脱氢酶 (Glyceraldehyde-3-phosphate dehydrogenase,GAPDH)对应68个假基因,这也验证了之前管家基因拥有更多假基因的结论。文章还具体阐述了之所以较之前的鉴定结果有差别,主要的原因有两个:一是实验中未将有偏差的基因组区域加入,因为对于一个单一区域包含很大一簇专一作用的假基因 (例如Olfactory receptor假基因)来说,其并不能代表整个人类基因组内情况[22];二是随着基因注释的进步,注释基因整体数量的变化也会造成鉴定的假基因数量产生差异。
3 假基因的功能
假基因功能的研究主要经过了3个阶段:第1阶段是假基因发现的早期,这个阶段主要是在单个假基因鉴定的过程中由于基因内产生的缺陷而失去正常功能的报道;第2阶段是间接证据阶段,即通过研究证明了某些假基因在进化特点上有类似功能基因的特点,例如没有功能的假基因被证明突变的积累并不是完全中立等,并且也发现少量的假基因可以被转录;第3阶段是丰富的直接实验证据阶段,在这个阶段,主要涉及的是假基因对亲本基因的调控及其作用机理。下面就针对各阶段的研究进行总结 (表2)。
3.1 假基因可能有功能的进化证据
假基因由正常功能基因演化而来,在假基因化的漫长过程中,低表达的蛋白编码基因可能已经具有了假基因的特征,但由于处在过渡状态,这些基因仍然不会完全丧失功能:例如果蝇中假基因adh (Alcohol dehydrogenase)由于存在多个突变从而丧失编码蛋白的能力,但是Begun等[34]发现adh具有功能基因应有的特征,包括外显子核酸突变率比内含子低,密码子仍然保留偏好性以及沉默突变率明显高于替代突变率等。又如,鸡中IglV和IghV终止子出现的数量比正常核酸发生随机突变情况下产生的终止子少很多,而且大部分由于点突变产生的终止子能够通过修复机制还原为功能基因[27],此现象在老鼠中也被发现[35],说明替换机制可能使假基因恢复为原来的功能基因或者新的功能基因。Zou等[12]对植物中假基因进行了研究,比较了水稻和拟南芥中假基因的非同义替代/同义替代值,比值小于1的结果说明自然选择抑制有害突变发生,即许多植物的假基因在进化过程中并不是中立的,而是进行了很强的纯化选择[12]。通过对 685个拟南芥以及926个水稻的假基因——亲本基因研究,发现ω值≤0.2,说明这些假基因与大部分功能基因具有的选择限制一样强,进而表明其可能在相当长的时间为功能基因,后来才成为假基因。更进一步的研究还发现假基因的5′区域到第1个终止子比3′区域有更强的选择限制,说明假基因的 5′区域在提前终止子出现后的很长时间里仍有功能。

表2 假基因功能研究的3个阶段Table 2 Three stages on the function research of pseudogenes
3.2 假基因能够进行转录和表达
3.2.1 假基因的表达证据 (正义表达/反义表达)
假基因由于缺乏有功能的启动子和调节元件而不能编码蛋白,在单细胞低等生物体内,正在假基因化的基因能够被排除[36],但一些研究证实在具有比较大而复杂的基因组物种中,如小麦、大麦等,假基因存在十分普遍[37]。人们开始对多细胞生物体内存在的假基因进行功能研究,发现其具有转录功能。以一氧化氮合酶nos对应的假基因makorin1-p1为例:通过形成mRNA,makorin1-p1与 nos的 mRNA互补形成稳定的RNA-RNA杂合体,引起nos蛋白表达降低[29]。随后,Hirotsune等[38]把 sex-lethal基因插入到makorin1-p1假基因中部导致了小鼠的死亡。进一步生化及遗传学实验表明小鼠的死亡是由于makorin1-p1被破坏所致,第一次验证了假基因具有功能。而在同样的处理条件下,超表达makorin1-p1或者makorin1后转基因小鼠并未死亡,从而进一步证明假基因具有功能。随后,人类白细胞干扰素 (Leukocyte interferon)[39]、肿瘤抑制基因 pten[40]以及 oct4[41]当中的假基因都被发现能够进行转录,而且有的假基因比对应的功能基因有更多的转录子。王国亮等[42]应用RT-PCR技术检测50例良、恶性甲状腺病变中假基因hmga1l2 mRNA的表达。结果发现在12例结节性甲状腺肿、9例甲状腺腺瘤和15例甲状腺乳头状癌中,其阳性表达率均为100%。目前,通过 5′ RACE (Rapid Amplification of cDNA Ends),嵌合芯片分析 (Tiling Array Analysis)和高通量测序技术 (High-throughput Sequencing)[43]可以系统地分析整个生物体范围内的假基因转录功能,研究从单个基因走向全基因组范围。哺乳动物中利用相似表达标签分析发现2%~5%的假基因能够表达[43];GENCODE鉴定了11 224个人类假基因,其中863个能够进行转录[24]。RNA-seq技术的发展,解决了可变剪接[44](Alternative splicing)、等位基因特异性表达(Allelic-specific expression)[45]以 及 RNA 编 辑(RNA editing)[46]等复杂情形下的转录问题,人们也开始将其应用于假基因的功能研究中,如人类核糖体蛋白假基因的转录研究就利用了RNA-seq技术[47]:通过 RPKM (Reads Per Kilobase per Million mapped reads)预测基因表达量,对 RNA测序数据——Illumina Human Body Map 2.0 project中的16个人类组织进行研究,发现1个假基因的RPKM为170、3个假基因RPKM>10、13个假基因RPKM>5;而且与核糖体蛋白功能基因在几乎所有组织中表达不同的是,假基因的表达仅在特定组织中进行。紧随其后,通过RNA-seq技术,Shanker等[48]对鉴定出的293个代表人类13种癌症和正常组织的假基因进行了系统的功能研究,发现其在细胞分化和癌症的发展中起到重要作用。
对于假基因能够进行转录,目前主要有两种解释:一种是产生年代较新的假基因由于顺式调控元件区没有完全退化,完整的假基因编码区反应着完整的与之相连的启动子区,非同源基因的启动子可能由于与假基因相邻而驱动假基因转录。另一种认为其衍生于较早的基因,进化中有很强的纯化选择,最近才进入假基因化。但后一种情况的发生很少,所以还未有数据上的证据[12]。
随着假基因能够被转录的证据越来越多,人们将注意力集中在挖掘假基因转录与功能基因转录的区别。Zou等[12]对水稻和拟南芥EST/MPSS的分析表明,拟南芥和水稻中分别有73%和 49%的基因有 EST/MPSS表达证据,然而两者分别仅有2%~5%和2%~3%的假基因有表达证据。实验还比较了外显子在功能基因及假基因正反义链上的表达情况,发现拟南芥中外显子在正义链或反义链的表达均比假基因表达高,但水稻中 (正义链) 外显子的表达和假基因类似。另外,仅针对假基因正反义链的表达也被进行了研究,结果显示610个拟南芥 (16.79%) 与1 047个水稻 (22.91%)的假基因可能存在正义链表达[49],而523个拟南芥 (14.42%)和922个水稻 (20.17%)的假基因在反义方向表达[12],可以看到假基因在两种植物中的正义表达均比反义表达高一些,即正义链相比反义链更具有转录功能。因此,在转录的方式和特点上,假基因与功能基因有较大区别。
3.2.2 假基因的调控功能
自从假基因被发现能够转录,对其功能的探索就不断地进行。随着高通量链特异性RNA测序技术 (High-Throughput Illumina Strand-Specif i c RNA Sequencing)的发展,克服了传统RNA测序技术过程中由于缺少RNA极性信息而不能进行真核生物复杂转录组解码的缺陷,对基因组注释、新转录组收集、RNA正反义链鉴定以及精确基因表达分析都有很重要的意义[50]。通过链特异性RNA测序技术,人们将假基因与非编码小RNA联系在一起,通过测定非编码小RNA的两条链结构方面与假基因的联系并进行分析,获得了很多突破性的结论。目前,已确定的假基因功能表现为以下两点:
功能一:假基因通过产生siRNA影响亲本基因的表达。假基因之所以能够形成 siRNA,主要是因为其与功能基因具有序列相似性。通过两者互补配对,双链RNA能够与 Argonaute蛋白结合,在 Dicer酶作用下产生双链内源 siRNA(图2)[32-33,51-52],然后再在ATP的参与下,siRNA结合在 RISC (RNA-induced silencing complex)上,产生的复合物与靶标基因编码区或 UTR区配对进而降解靶标基因。利用此原理,人们首先以生物信息学方法找到假基因来源的siRNA,通过siRNA对靶基因作用过程中酶的敲除或抑制,然后利用实时定量 RCR检测靶基因表达量的变化进而间接挖掘假基因的调节作用。Tam等[32]不仅验证了假基因产生的内源 siRNA能调节基因的表达,而且描述了假基因衍生的siRNA作用的两种方式:假基因-功能基因配对方式和假基因-假基因方式。前者功能基因作为形成 siRNA的正义链,反义链来自于与功能基因互补的假基因[32];后者通过重复片段的插入,形成发夹结构进而得到双链siRNA。随后,Guo等[53]提出假基因可以通过顺式或反式作用产生小 RNA。其鉴定了145个能够形成siRNA的假基因,其中大于一半为24 nt,siRNA的形成依赖于RNA聚合酶Rdr2以及类Dicer蛋白3 (Dcl3),表明小RNA可能会进行顺式作用抑制假基因本身的转录;反式作用则主要通过假基因产生小 RNA作为反义siRNA干扰亲本功能基因。Wen等[54]对非洲布氏锥体虫African Trypanosoma brucei的研究表明,假基因能够产生siRNA,再通过RNA干扰作用对基因表达进行抑制,同时,抑制虫体内类Dicer蛋白 Tbdcl1产生过程中对应酶的基因,通过实时定量 PCR方法检测到靶基因表达量上升,从而确认了假基因介导的 siRNA对靶基因的调控功能 (图 2)。
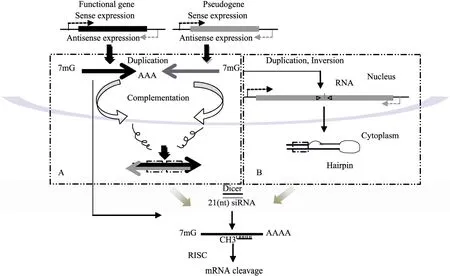
图2 假基因介导的内源siRNAs (endo-siRNAs) 产生过程示意图[32-33,51-52]Fig. 2 Pseudogene-derived endo-siRNAs formation process[32-33,51-52]. Pseudogenes could be formatted in two ways:duplication & retrotransposition. (A) The mRNA transcript of its parent gene and the antisense transcript from pseudogenes complemented with each other and formed the double-stranded small interfering RNA. (B)Single strand RNA was formed through retrotransposition. Hairpin structure was formed through its own complementation and the transcribed double strand mRNA formed 21 nt endo-siRNAs cut by Dicer, which degraded mRNA guided by the RISC complex.
功能二:假基因通过miRNA调控基因表达。MicroRNAs (miRNAs)是在真核生物中发现的一类内源性的具有调控功能的非编码 RNA,在动植物所有的细胞过程和细胞类型当中参与发育、细胞增殖和凋亡以及病毒防御等很多重要的调节途径。miRNA通过与编码序列互补或结合于目的基因的3′ UTR区从而减少目的蛋白的富集[55]。目前已进行的研究中,假基因与 siRNA的研究较多,因为siRNA为双链小干扰RNA,假基因作为其中一条链可以很好地解释 siRNA行使功能的方式。但鉴于miRNA为单链,且产生及作用机制已经被研究得较为透彻,针对miRNA与假基因之间关系的研究还较少。Megraw等[56]也认为 miRNA和假基因之间没有一个广泛存在的联系。然而,Poliseno等[30]研究了肿瘤抑制基因 pten的假基因 ptenp1与miRNA之间的关系,发现ptenp1的3′ UTR区有抑制肿瘤活性的作用,以pten为靶基因的miRNA同样会将ptenp1作为靶基因,即一个基因的假基因与基因竞争miRNA的结合。进一步的研究表明pten与假基因ptenp1的3′ UTR区前2/3是相近的,其中S1部分完全一致,而miRNA以pten的 S1部分为靶位点,故而同样以 ptenp1的 S1部分进行作用,即 miRNA同样作用于 ptenp1(图 3B,3C)。另外,由于 ptenp1保守性更差,一些miRNA仅以其作为靶基因,而完全不作用于pten。为了确定pten与ptenp1之间竞争miRNA结合是否为特例,Poliseno等[30]还将研究扩展到其他与癌症相关的基因及假基因中。结果表明miRNA与功能基因及其假基因的结合位点非常保守:例如miR-145结合位点在oct4及其假基因oct4-pg1,oct4-pg3,oct 4-pg4以及 oct4-pg5(oct4-pg1和oct4-pg5仅仅在癌症组织中表达,正常组织中不表达;oct4-pg5的5′端被截短,仅仅是跟着 3′ UTR的一小部分开放阅读框表达);miR-1家族结合于cx43及其假基因;miR-34家族结合在cdk4ps;miR-182结合于foxo3b;miR-17家族结合于e2f3p1以及miR-143和let-7家族结合于kras1p。而且根据pten与ptenp1的3′ UTR区类似,研究发现kras与假基因kras1p也有相似的关系,即kras1p 3′ UTR在DU145细胞中过表达会造成kras mRNA积累增加,换句话说,kras1p的增加会使得 miRNA与 kras的结合减弱。Poliseno等[30]将这一发现进行了扩展,认为任何两个共表达基因——分别命名为 g和 G,如果两者同时被同一个非编码RNA调控,那么g和G被称为具有诱捕 (Decoys)关系;如果某一RNA对g丰度有直接影响,那么也会影响G的丰度。

图3 假基因与功能基因竞争miRNA结合位点[30]Fig. 3 Pseudogenes were targeted by parent gene-targeting miRNAs[30]. (A) Different from the usually thought that miRNA regulated the abundance of the target gene, mRNA competed for the binding site of miRNA and the amount of one kind of mRNA influenced the other[57]. (B)pten is protected from miRNA binding by ptenp1. (C)The 3′ UTR region of pten and ptenp1, which contained a highly conserved part (Dark grey)and one with low conservation (Light grey). For the dark grey part (S1), pten-targeting miRNA seed matches within the high homology region are conserved between ptenp1 and pten, miRNA bond ptenp1 to keep it from binding on pten, thus kept pten free from miRNA cleavage.
此发现通过比较miRNA作用靶基因位点处功能基因与假基因的结构,找到假基因与miRNA的关系。另外,根据目前所知,作用位点位于3′UTR的miRNA仅占一小部分,miRNA还可以作用于生物体的5′ UTR或编码区,类似的方法探究这些位置上功能基因与假基因的关系可能会获得更多信息。相信此研究将作为后续研究的铺垫,未来基于功能基因及其假基因上的miRNA靶位点为突破口寻找假基因与miRNA之间关系的研究将会越来越多。
通过竞争性结合miRNA从而产生调节作用并不仅仅在上述基因中发生,此也不为假基因所特有。Fau等[58]首先在植物中提出了“target mimicry”的概念。通过对比 IPS1 (Induced by Phosphate Starvation1)和 PHO2 (Phosphate2)的mRNA,发现两者同时具有与miR399序列互补的相似性非常高的片段,IPS1能够通过结合miR399从而阻止其抑制PHO2 mRNA的积累和翻译。Salmena等[59]扩大了“target mimicry”的范围,认为在假基因、mRNA、长链非编码RNA(Long non-coding RNAs)以及其他能够作为miRNA结合位点的 RNA分子中都有竞争miRNA进而对功能基因进行调节的可能,改变了之前“蛋白编码 mRNA必须通过翻译成蛋白而发挥作用”的观点,并且将这类RNA命名为竞争性内源 RNAs (Competing endogenous RNAs,ceRNA)。随后,人们对 ceRNA 转录后水平的调节作用进行了更深入的研究[60-63],例如Cesana等[60]证明了针对肌肉组织的长链非编码RNA,linc-MD1在肌肉差异表达过程中的重要作用。因此,ceRNA的机制从植物到人类都广泛、保守地存在,假基因作为 ceRNA的一类,通过竞争miRNA进而对功能基因进行调控的机制也应广泛存在于各物种中。
但与上述相反,Chiefari等[64]在对假基因hmga1-p 3′ UTR进行功能研究时,发现其对维持功能基因表达稳定起很大作用。实验首先将 5′端缺少248 bp的区域 (与hmga1-p 5′ UTR区相关)连接载体后瞬时转染入 hela细胞中,发现hmga1 mRNA表达下降了50%。将hmga1-p全长与载体连接后转入细胞中,获得了相类似的结果;相反,将 hmga1-p 3′端缺失1 276 bp (与 hmga1 3′ UTR相关)的片段瞬时转入细胞中发现,hmga1表达几乎没有变化,从而得知hmga1-p 3′区域是影响 hmga1表达稳定性的关键因素。之后,为了寻找 hmga1-p 3′ UTR影响功能基因hmga1 mRNA稳定性的位置及方式,Chiefari将hmga1-p 3′ UTR进行突变,通过实时定量PCR发现:突变掉hmga1-p 3′ UTR 的291~1 026部分时,hmga1的表达轻微上升;突变掉hmga1-p 3′UTR的1 253~1 276部分,hmga1的表达显著下降;而突变掉hmga1-p 3′ UTR的3′末端152 bp片段后,发现hmga1的表达下降40%~50%,这个结果说明hmga1-p的RNA通过特定位置的反式调节方式对hmga1表达进行调控。不仅如此,Chiefari等[64]还在生理方面做了实验,以图通过实验找到假基因调节的机制。其发现了1个影响mRNA稳定性的蛋白αCP1。用siRNA抑制αCP1表达后,hmga1 mRNA表达下降。αCP1蛋白能够影响RNA-蛋白结合活性,含有一组KH-保守域的RNA-保守域结合蛋白能够特异性地结合于C富集区,进而控制 mRNA的稳定性[65]。而hmga1-p 3′ UTR区富含 C,能够竞争性地结合αCP1蛋白从而使hmga1 mRNA表达。
3.2.3 组织特异性表达
假基因作为结构具有缺陷的“不成功产物”,虽然转录过程与功能基因相同,但在转录结果及特点上存在区别。Zheng等[22]在研究假基因转录过程中,在编码区域找到 14个能够进行转录的假基因,其中5个在睾丸中进行表达,另外4个被发现也表达于特定组织中。不仅如此,有时特定组织或条件下假基因比同源功能基因转录更为普遍。例如,肌球蛋白轻链激酶假基因mylkp1(Myosin light chain kinase pseudogene)部分复制于 mylk (编码肌球蛋白轻链激酶 smooth muscle myosin light chain kinase亚型,smMLCK isoforms),mylkp1启动子在正常支气管上皮细胞中几乎没有活性,但是却在肺癌细胞中表现出了很高的活性,且在癌细胞中的过表达会抑制RNA稳定性进而抑制 smMLCK表达,从而促使细胞分裂增加[66]。之所以假基因的表达具有组织特异性,是因为虽然假基因与功能基因有很高的序列相似性,但作为“不成功产物”,在结构上,尤其是启动子上有突变的位点,在正常情况下可能造成假基因少量表达甚至不表达,但是当条件改变时,例如癌症等特定条件下的转录因子能够弥补假基因丧失的功能。除了上述情况外,不同生理条件下假基因表达也可能产生特异的变化[64],例如当酵母在新的胁迫环境下,假基因能够在特殊信号的刺激下恢复活性[67]。拟南芥胁迫过程中,基因及假基因表达也会随之发生变化[68]。
4 结语
假基因相关的研究发展迅速,目前已发现参与生物生长发育、调节生物与非生物胁迫等各个方面。然而,在对假基因功能研究的过程中,对同一假基因是否存在“功能性”却提出了不同的观点。例如,Hirotsune等[38]得出makorin1-p1基因具有“功能性”的结论。然而,Gray等[69]却否定了上述基因的“功能性”观点:认为之前归功于makorin1-p1产生的转录子实际上是被忽视的从makorin1位点得到的mRNA亚型;且其同时证明了 makorin1-p1 5′区域位点完全假基化,不能进行转录。相反的结论需要更加系统的分析和数据支持。相信随着对基因组鉴定的不断完善,未知基因逐渐减少,人们对于某一假基因特定功能的研究将具有更加确定的结论。
另外,针对假基因的功能大都是从假基因本身的单一层面进行研究,而对整体假基因功能机制研究还较少。假基因-小RNA-靶基因之间是相互联系密不可分的,假基因可以产生小 RNA,小RNA可以反过来抑制假基因表达,或者抑制靶功能基因表达;一个小RNA可以有较多靶基因,而单独的一个基因又可以拥有很多不同调节方向的假基因,且一般来讲,miRNA及 siRNA不会触发次级siRNA产生,但目前却证明22 nt的miRNA能够触发次级siRNA的合成[70]。人们对于研究生物体内这种复杂的网状情形还处于初级阶段。通过生物信息学的方法结合现今对RNA-seq技术逐渐成熟的使用进而挖掘参与调节途径的假基因与小 RNA、靶基因以及形成假基因的亲本基因之间的关系,并在不同的生物之间建立网络结构必将成为今后研究的热点。对假基因的研究也应从某个具体的性状扩展到细胞整体水平。
虽然假基因的调控功能主要发现于动物以及人类中,但植物中的研究也逐渐发展起来。目前,水稻和拟南芥等植物全基因范围内假基因鉴定数据已经发表,已有文献报道DCL对siRNA发挥功能起关键作用:水稻中类Dicer4在siRNA作用过程中为关键因素[71];Qian等[72]对玉米中的5个Dicer、18个Ago以及5个依赖RNA的RNA聚合酶进行全面的分析,发现 Dcl包含有DExD、Helicase-C、DUF283、PAZ,RNaseIII以及dsRB保守的domain区域,不同DCL包含的保守区域有一些区别。我们可以在找到假基因产生小干扰RNA的基础上,根据已知信息,通过抑制 siRNA起作用的关键因素,查看 siRNA靶基因的表达变化进而间接得到假基因在植物体内作用的证据。除此之外,在动物中发现的假基因功能很可能在植物中有类似的情况,例如:ceRNA的发现,早在2007年植物中就出现“target mimicry”的说法[58],而在这之后才在动物的研究中提出ceRNA,假基因转录的产物作为ceRNA的一种,在动物中的机制很可能会在植物中出现相类似的机制。另外,假基因的精确鉴定是研究其在生物体功能方面发挥作用的研究基础。但目前为止,对假基因鉴定方法仍然需要将基因组DNA与蛋白序列比对,去掉已知编码序列的过程中会同时删掉一些正在变成假基因过程中的“年轻假基因”,因为一个造成ORF破坏的突变要经历上百万年的时间才能变成稳定的缺少选择限制的基因,由于没有充足的时间积累提前终止子或者移码突变,这些“年轻假基因”就可能不被列入假基因名下造成数量被低估。同样地,非常古老的假基因由于已经没有同源的蛋白编码基因而不能被找到。因此,看似成熟的鉴定方法实际上丢掉了很多有用的信息。站在更广的角度上,鉴定方法都依据于假基因的定义,定义中的关键就是“无功能”。但这个无功能几乎在实验上不可能得到实践,发掘“无功能”比“有功能”难很多[13]。现在,越来越多的科研小组对所谓假基因在生物体内基因调节的意义进行发表[33],截止目前已经有很大一部分非编码序列,包括已注释的假基因能够产生转录子。另外还有研究称假基因不通过转录可直接发挥功能[13],假基因的定义越来越难。称呼一个有调节功能但无编码功能的基因假基因还对吗?其定义界限变得模糊。所以,随着研究的进步,很可能假基因这个名字会被赋予更多的含义,又或者将其中的一类分出并定义成为新的一类调控基因,但研究假基因无可争议具有重大的意义。
[1]Jacq C, Miller J, Brownlee G. A pseudogene structure in 5S DNA of Xenopus laevis. Cell, 1977,12(1): 109−120.
[2]Yao A, Charlab R, Li P. Systematic identification of pseudogenes through whole genome expression evidence profiling. Nucleic Acids Res, 2006,34(16): 4477−4485.
[3]Molineris I, Sales G, Bianchi F, et al. A new approach for the identification of processed pseudogenes. J Comput Biol, 2010, 17(5):755−765.
[4]Zhang ZD, Frankish A, Hunt T, et al. Identification and analysis of unitary pseudogenes: historic and contemporary gene losses in humans and other primates. Genome Biol, 2010, 11(3): R26.
[5]Wen YZ, Zheng LL, Qu LH, et al. Pseudogenes are not pseudo any more. RNA Biol, 2012, 9(1):27−32.
[6]Fang J. The processed pseudogene POU5F1P1 in 8q24 is expressed in tumor and shows oncogenicity. Cancer Res, 2012, 72(8): S1.
[7]Kreahling J, Graveley BR. The origins and implications of Aluternative spicing. Trends Geneti, 2004, 20(1): 4−11.
[8]Yang L, Takuno S, Waters ER, et al.Lowly-expressed genes in Arabidopsis thaliana bear the signature of possible pseudogenization by promoter degradation. Mol Biol Evol, 2011, 28(3):1193−1203.
[9]Harrison PM, Hegyi H, Balasubramanian S, et al.Molecular fossils in the human genome:identification and analysis of the pseudogenes in chromosomes 21 and 22. Genome Res, 2002, 12(2):272–280.
[10]Petrov D, Lozovskaya E, Hartl D. High intrinsic rate of DNA loss in Drosophila. Nature, 1996, 384(6607): 346−349.
[11]Benovoy D, Drouin G. Processed pseudogenes,processed genes, and spontaneous mutations in the Arabidopsis genome. J Mol Evol, 2006, 62(5):511−522.
[12]Zou C, LehtiShiu MD, ThibaudNissen F, et al.Evolutionary and expression signatures of pseudogenes in arabidopsis and rice plant physiol.PLoS Genetics, 2009, 151(1): 3−15.
[13]Podlaha O, Zhang JZ. Pseudogenes and Their Evolution [EB/OL]. [2012-04-05]. Encyclopedia of Life Sciences, 2010, a0005118. http://onlinelibrary.wiley.com/doi/10.100219780470015902.a0005118.pubz/abstract.
[14]Balasubramanian S, Zheng D, Liu YJ, et al.Comparative analysis of processed ribosomal protein pseudogenes in four mammalian genomes.Genome Biol, 2009, 10(1): R2.
[15]Harrison P, Kumar A. A small reservoir of disabled orfs in the yeast genome and its implications for the dynamics of proteome evolution. J Mol Biol, 2002,316(3): 409−419.
[16]Zhang Z, Harrison PM, Liu Y, et al. Millions of years of evolution preserved: a comprehensive catalog of the processed pseudogenes in the human genome. Genome Res, 2003, 13(12): 2541−2558.
[17]Zhang Z, Gerstein M. Large-scale analysis of pseudogenes in the human genome. Curr Opin Genet Dev, 2004, 14(4): 328−335.
[18]Liu GQ, Li H. The correlation of processed pseudogene distribution with recombination rate and gene density in human genome. Acta Biophysica Sin, 2008, 24(5).
[19]Chen SM, Ma KY, Zeng J. Pseudogene: lessons from PCR bias, identification and resurrection. Mol Biol Rep, 2011, 38(6): 3709−3715.
[20]Altschul S, Madden T, Schäffer A, et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs. Nucleic Acids Res, 1997, 25(17): 3389−3402.
[21]Zheng D, Gerstein MB. A computational approach for identifying pseudogenes in the ENCODE regions. Genome Biol, 2006, 7(1): S13.
[22]Zheng D, Gerstein MB, Frankish A. Pseudogenes in the ENCODE regions: consensus annotation,analysis of transcription, and evolution. Genome Res, 2007, 17(6): 839−851.
[23]Schwartz S, Kent WJ, Smit A, et al. Human-mouse alignments with BLASTZ. Genome Res, 2003, 13(1): 103−107.
[24]Pei B, Sisu C, Frankish A, et al. The GENCODE pseudogene resource. Genome Biol, 2012, 13:1465−6906.
[25]Graur D, Shuali Y, Li W. Deletions in processed pseudogenes accumulate faster in rodents than in humans. J Mol Evol, 1989, 28(4): 279−285.
[26]Li W, Gojobori T, Nei M. Pseudogenes as a paradigm of neutral evolution. Nature, 1981,292(5820): 237−239.
[27]Rothenfluh HS, Blanden RV, Steele EJ. Evolution of V genes: DNA sequence structure of functional germline genes and pseudogenes. Immunogenetics,1995, 42: 159−171.
[28]Frith MC, Wilming LG, Forrest A, et al.Pseudo-messenger RNA: phantoms of the transcriptome. PLOS Genetics, 2006, 2(4): e23.
[29]Korneev SA, Park J, O’Shea M. Neuronal expression of neural nitric oxide synthase (nNOS)protein is suppressed by an antisense RNA transcribed from an NOS pseudogene. J Neurosci,1999, 19(18): 7711−7720.
[30]Poliseno L, Salmena L, Zhang J, et al. A coding-independent function of gene and pseudogene mRNAs regulates tumour biology.Nature, 2010, 465(7301): 1033−1038.
[31]Pink RC, Wicks K, Caley DP, et al. Pseudogenes:pseudo-functional or key regulators in health and disease? RNA, 2011, 17(5): 792−798.
[32]Tam O, Aravin A, Stein P, et al.Pseudogene-derived small interfering RNAs regulate gene expression in mouse oocytes. Nature,2008, 453(7194): 534−538.
[33]Watanabe T, Totoki Y, Toyoda A, et al.Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes.Nature, 2008, 453(7194): 539−543.
[34]Begun DJ. Origin and evolution of a new gene descended from alcohol dehydrogenase in Drosophila. Genetics, 1997, 145(2): 375−382.
[35]Schiff C, Milili M, Fougereau M. Functional and pseudogenes are similarly organized and may equally contribute to the extensive antibody diversity of the IgVHII family. EMBO J, 1985,4(5): 1225−1230.
[36]Kuo C, Ochman H. The extinction dynamics of bacterial pseudogenes. PLoS Genetics, 2010,6(8):e1001050.
[37]Wicker T, Mayer KF, Gundlach H, et al. Frequent gene movement and pseudogene evolution is common to the large and complex genomes of wheat, barley, and their relatives. Plant Cell, 2011,23(5): 1706−1718.
[38]Hirotsune S, Yoshida N, Chen A, et al. An expressed pseudogene regulates the messenger-RNA stability of its homologous coding gene. Nature, 2003, 423(6935): 91−96.
[39]Goeddel DV, Leung DW, Dull TJ, et al. The structure of eight distinct cloned human leukocyte interferon cDNAs. Nature, 1981, 290(5801): 20−26.
[40]Fujii G, Morimoto A, Berson A, et al.Transcriptional analysis of the PTEN/MMAC1 pseudogene, psiPTEN. Oncogene, 1999, 18(9):1765−1769.
[41]Redshaw Z, Strain AJ. Human haematopoietic stem cells express Oct4 pseudogenes and lack the ability to initiate Oct4 promoter-driven gene expression. J Negat Results Biomed, 2010, 9(1): 2.
[42]Wang GL, Zhang GC, Li F, et al. The Expression of pseudogene HMGA1L2 in Thyroid Lesions.Hereditas(Beijing)2006, 28(11): 1365−1370.
[43]Harrison P, Zheng D, Zhang Z, et al. Transcribed processed pseudogenes in the human genome: an intermediate form of expressed retrosequence lacking protein-coding ability. Nucleic Acids Res,2005, 33(8): 2374−2383.
[44]Sultan M, Schulz MH, Richard H, et al. A global view of gene activity and alternative splicing by deep sequencing of the human transcriptome.Science, 2008, 321(5891): 956−960.
[45]Pastinen T. Genome-wide allele-specific analysis:insights into regulatory variation. Nat Rev Genet,2010, 11(8): 533−538.
[46]Li M, Wang IX, Li Y, et al. Widespread RNA and DNA sequence differences in the human transcriptome. Science, 2011, 333(6038): 53−58.
[47]Tonner P, Srinivasasainagendra V, Zhang S, et al.Detecting transcription of ribosomal protein pseudogenes in diverse human tissues from RNA-seq data. BMC Genomics, 2012, 13(1): 412.
[48]Shanker KS, Chandan KS, Sunita S, et al.Expressed pseudogenes in the transcriptional landscape of human cancers. Cell, 2012, 149(7):1622−1634.
[49]Yamada K, Lim J, Dale JM, et al. Empirical analysis of transcriptional activity in the Arabidopsis genome. Science, 2003, 302(5646):842−846.
[50]Zhong S, Joung JG, Zheng Y, et al.High-throughput illumina strand-specific RNA sequencing library preparation. Cold Spring Harb Protoc, 2011(8): 940−949.
[51]Okamura K, Chung W, Ruby J, et al. The Drosophila hairpin RNA pathway generates endogenous short interfering RNAs. Nature, 2008,453(7196): 803−806.
[52]Kawamura Y, Saito K, Kin T, et al. Drosophila endogenous small RNAs bind to Argonaute2 in somatic cells. Nature, 2008, 453(7196): 793−797.
[53]Guo X, Zhang Z, Gerstein MB, et al. Small RNAs originated from pseudogenes: cis- or trans-acting?PLoS Comput Biol, 2009, 5(7): e1000449.
[54]Wen YZ, Zheng LL, Liao JY, et al.Pseudogene-derived small interference RNAs regulate gene expression in African Trypanosoma brucei. Proc Natl Acad Sci USA, 2011, 108(20):8345−8350.
[55]Bartel DP. MicroRNAs: target recognition and regulatory functions. Cell, 2009, 136(2): 215−233.
[56]Megraw M, Sethupathy P, Corda B, et al. miRGen:a database for the study of animal microRNA genomic organization and function. Nucleic Acids Res, 2007, 35(Database issue): D149−155.
[57]Seitz H. Redefining microRNA targets. Curr Biol,2009, 19(10): 870−873.
[58]Fau F ZJ, Adrian V, Marco T, et al. Target mimicry provides a new mechanism for regulation of microRNA activity. Nat Genet, 2007, 39(8):1033−1037.
[59]Salmena L, Poliseno L, Tay Y, et al. A ceRNA hypothesis: the rosetta stone of a hidden RNA language? Cell, 2011, 146(3): 353−358.
[60]Cesana M, Cacchiarelli D, Legnini I, et al. A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA. Cell,2011, 147(2): 358−369.
[61]Karreth FA, Tay Y, Perna D, et al. In vivo identification of tumor-suppressive PTEN ceRNAs in an oncogenic BRAF-induced mouse model of melanoma. Cell, 2011, 147(2): 382−395.
[62]Tay Y, Kats L, Salmena L, et al.Coding-independent regulation of the tumor suppressor PTEN by competing endogenous mRNAs. Cell, 2011, 147(2): 344−357.
[63]Sumazin P, Yang X, Chiu HS, et al. An extensive microRNA-mediated network of RNA-RNA interactions regulates established oncogenic pathways in glioblastoma. Cell, 2011, 147(2):370−381.
[64]Chiefari E, Iiritano S, Paonessa F, et al.Pseudogene-mediated posttranscriptional silencing of HMGA1 can result in insulin resistance and type 2 diabetes. Nat Commun, 2010, 1: 40.
[65]Liebhaber SA. mRNA stability and the control of gene expression. Nucleic Acids Symp, 1997, 36:29−32.
[66]Han YJ, Ma SF, Yourek G, et al. A transcribed pseudogene of MYLK promotes cell proliferation.FASEB J, 2011, 25(7): 2305−2312.
[67]Gerstein M, Zheng D. The real life of pseudogenes.Sci Am, 2006, 295(2): 48−55.
[68]Zeller G, Henz SR, Widmer CK, et al.Stress-induced changes in the Arabidopsis thaliana transcriptome analyzed using whole-genome tiling arrays. Plant J, 2009, 58(6): 1068−1082.
[69]Gray TA, Wilson A, Fortin PJ, et al. The putatively functional Mkrn1-p1 pseudogene is neither expressed nor imprinted, nor does it regulate its source gene in trans. Proc Natl Acad Sci USA,2006, 103(32): 12039−12044.
[70]Chen HM, Chen LT, Patel K, et al. 22-Nucleotide RNAs trigger secondary siRNA biogenesis in plants. Proc Natl Acad Sci USA, 2010, 107(34):15269−15274.
[71]Liu B, Chen Z, Song X, et al. Oryza sativa dicer-like4 reveals a key role for small interfering RNA silencing in plant development. Plant Cell,2007, 19(9): 2705−2718.
[72]Qian Y, Cheng Y, Cheng X, et al. Identification and characterization of Dicer-like, Argonaute and RNA-dependent RNA polymerase gene families in maize. Plant Cell Rep, 2011, 30(7): 1347−1363.
