肿瘤坏死因子受体基因单核苷酸多态性与肺炎严重程度的相关性*
2013-10-25郑少强袁伟锋黄文杰
李 理, 郑少强, 袁伟锋, 黄文杰△
(1广州军区广州总医院呼吸内科,广东 广州 510010; 2南方医科大学第三附属医院呼吸内科,广东 广州 510630)
肿瘤坏死因子受体基因单核苷酸多态性与肺炎严重程度的相关性*
李 理1, 郑少强2, 袁伟锋1, 黄文杰1△
(1广州军区广州总医院呼吸内科,广东 广州 510010;2南方医科大学第三附属医院呼吸内科,广东 广州 510630)
目的比较肿瘤坏死因子受体(tumor necrosis factor receptor,TNFR)基因多个等位基因在肺炎人群中的分布频率,分析基因多态性与肺炎发病率和病情严重程度的相关性。方法纳入66例肺炎患者与66例既往无肺炎的健康体检者,抽提各研究对象外周血DNA,通过聚合酶链式反应-限制性片段长度多态性或基因测序的方法检测TNFR1+36A/G、TNFR1-609G/T、TNFR2+676T/G、TNFR2 +1663T/G、TNFR2 +1668A/G和TNFR2 +1690C/T各多态性位点在肺炎患者与健康体检者、重症肺炎与非重症肺炎患者中的分布频率,并统计分析各基因分布频率与肺炎发生率和严重程度的相关性。结果TNFR1-609G与T等位基因在肺炎患者中分布频率分别为40.9%与59.1%,在健康体检者中的分布频率分别为53.8%与46.2%;TNFR1-609T等位基因在肺炎患者中的分布频率较高(P<0.05)。余基因的各等位基因在肺炎患者与健康体检者中的分布频率差异无统计学意义。TNFR1-609G与T等位基因在重症肺炎患者中分布频率分别为25.0%与75.0%,在非重症肺炎患者中的分布频率分别为46.0%与54.0%,T等位基因在重症肺炎患者中的分布频率较高(P<0.05)。TNFR2 +1690C与T等位基因在重症肺炎患者中分布频率分别为81.1%与18.9%,在非重症肺炎患者中的分布频率分别为61.0%与39.0%,C等位基因在重症肺炎患者中分布频率较高(P<0.05),余基因的各等位基因在重症肺炎与非重症肺炎患者中的分布频率差异无统计学意义。结论携带TNFR1-609T等位基因的个体更易罹患肺炎,而携带TNFR1-609T及TNFR2 +1690C等位基因的个体肺炎病情较严重,易进展为重症肺炎。
受体,肿瘤坏死因子; 多态性,单核苷酸; 肺炎
当前众多研究均显示重症肺炎的发病机制与肺局部炎症反应调节失衡有关,促炎反应过度活化与抑炎反应活化相对不足,导致机体正常组织与细胞的炎症性破坏,损伤肺脏的通气与换气功能,在宏观上表现为重症肺炎。 本实验室前期研究已证实炎症反应通路中不同分子的基因多态性可能影响炎症反应强度,进而影响机体对肺炎的易感性与罹患重症肺炎的风险,如携带TNF-α-308A等位基因的患者在感染后体内炎症反应水平较高,更容易发展成感染性休克[1]。
TNF-α与其受体TNFR结合将向细胞内传递炎症反应活化的信号,二者质或量的改变均将影响炎症反应强度。TNFR作为TNF-α的受体,是TNF-α/NF-κB/TNF-α炎症反应放大途径的重要分子。但对于在感染性疾病TNFR基因多态性的研究不多,在肺炎中的研究仅见个别报道。本研究为明确TNFR基因多态性与肺炎易感性、病情严重程度的相关性,通过聚合酶链式反应-限制性片段长度多态性(po-lymerase chain reaction-restriction fragment length polymorphism,PCR-RFLP)或基因测序的方法检测肺炎患者与健康体检者TNFR基因中多个多态性位点不同等位基因的分布,并分析其分布规律与病情严重程度的相关性,为通过检测TNFR基因多态性预测肺炎患者病情与预后提供实验室依据。
材 料 和 方 法
1研究对象
研究对象为2010年10月~2011年 6月就诊于广州军区广州总医院呼吸科的66例成年肺炎患者(男性37名,女性29名,年龄60.59岁±14.67岁)及66例既往无肺炎病史的健康体检者(男性31名,女性35名,平均年龄54.62岁±15.45岁)。各受试者之间无血缘关系。在肺炎患者中,16例诊断为重症肺炎,死亡7例。
肺炎诊断标准:(1)新近出现的咳嗽、咳痰,或原有呼吸道疾病症状加重,并出现脓性痰;伴或不伴胸痛、气急等症状;(2) 发热;(3) 肺实变体征和(或) 湿性罗音;(4)白细胞>10×109/L 或<4×109/L,伴或不伴核左移;(5) 胸部X 线检查显示片状、斑片状浸润性阴影或间质性改变,伴或不伴胸腔积液。以上1~4 项中任何1项加第5项,并排除肺结核、肺部肿瘤、非感染性肺间质性疾病、肺水肿、肺不张、肺栓塞、肺嗜酸性粒细胞浸润症、肺血管炎外等,可建立肺炎临床诊断。重症肺炎诊断标准参照2007年IDSA/ATS关于成人社区获得性肺炎的诊治指南。主要标准:(1)需要机械通气;(2)感染性休克或需要应用血管活性物质;次要标准(1)呼吸频率≥30 min-1;(2)氧合指数(PaO2/FiO2)<250;(3)双侧或多叶性肺炎;(4)昏迷或定向力缺失;(5)尿毒症(血尿素氮≥20 mg/dL);(6)白细胞计数<4×109/L;(7)血小板计数<100×109/L;(8)低体温<36 ℃;(9)低血压需要液体复苏。符合3条次要标准或1条主要标准[2]。排除标准:(1)伴有代谢性疾病,如糖尿病、甲状腺功能亢进、甲状腺功能减低、原发性醛固酮增多症等;(2)伴良性或恶性肿瘤;(3)伴自身免疫性疾病;(4)伴病毒感染。
本研究以肺炎患者为肺炎组(community-acquired pneumonia group, CAP组),以健康体检者为对照组(control group);其中CAP组按病情严重程度分为重症肺炎组(severe community-acquired pneumonia group, SCAP组)与非重症肺炎组(non-severe community-acquired pneumonia group, NSCAP组)。
2TNFR的单核苷酸多态性(singlenucleotidepo-lymorphism,SNP)检测
收集研究对象的外周血,EDTA抗凝,取200 μL提取外周血DNA,按照Fermentas公司DNA抽提试剂盒(Genomic DNA Purification Kit,#K0512)说明书进行。
以抽提的DNA作为模板,使用PCR-RFLP或基因测序的方法对各研究对象的以下SNP进行分析:TNFR1+36A/G、TNFR1-609G/T、TNFR2+676T/G、TNFR2+1663T/G、TNFR2+1668A/G与TNFR2+1690C/T。PCR试剂采用DreamTaqTMPCR Master Mix 2×(Fermentas,#K1071)。PCR体系:DreamTaqTMPCR Master Mix 10 μL,上游及下游引物各0.8 μL(浓度10 μmol/L,引物序列见表1),DNA模板3 μL,去离子水5.4 μL。PCR条件:95 ℃预变性5 min,95 ℃变性、退火(温度见表1)与72 ℃延伸各30 s,终延伸5 min。
以各PCR扩增产物作为底物,采用限制性内切酶进行酶切反应。携带TNFR1+36A/G、TNFR1 -609G/T与TNFR2+676T/G 这3个SNP的PCR产物酶切体系、条件、酶切产物片段大小与基因型判断见表2。
TNFR2 +1663T/G、 +1668A/G和 +1690C/T这3个SNP在序列上位置接近,采用基因测序的方法共同对上述3个SNP进行检测。
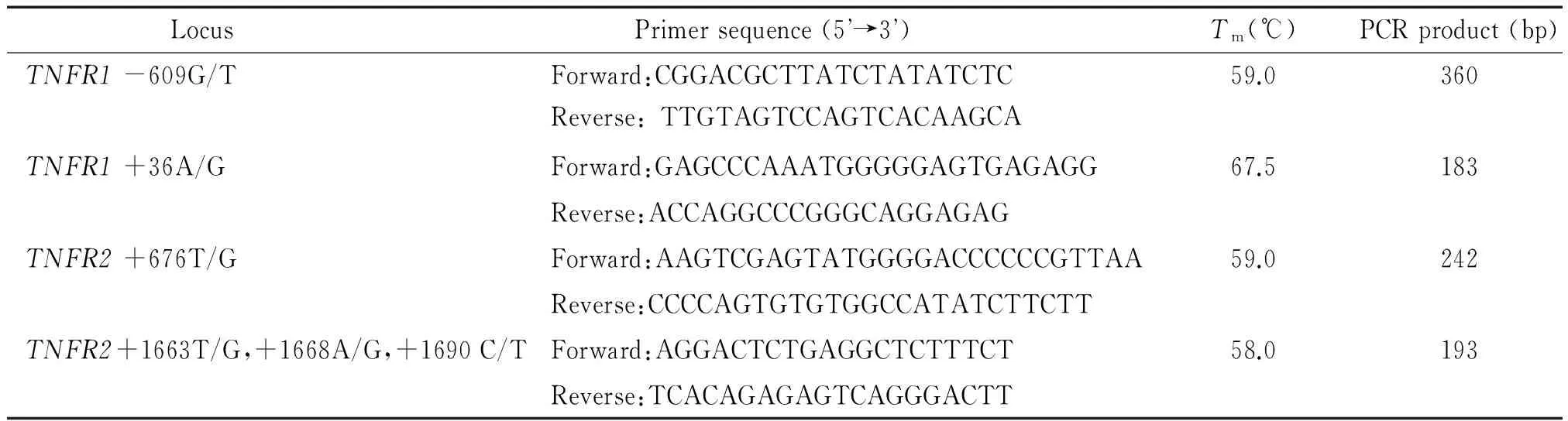
表1 携带目标SNP的DNA片段PCR扩增
表2酶切反应条件与基因型判断
Table 2. Outline of genotypes ofTNFRand the reaction condition of restriction enzyme

LocusSubstrate(bp)EndonucleaseTemperature(℃)Allele(bp)TNFR1-609G/T360TaaI65GT:360+263+97TT:263+97GG:108+75TNFR1+36A/G183MspAII37GA:183+108+75AA:183TNFR2+676T/G242NlaIII37TT:109+133GT:242+109+133GG:242
3统计学处理
计量资料行正态性检验,正态性分布资料以均数±标准差(mean±SD)表示,非正态资料以中位数表示。计数资料的组间比较采用2检验。采用SPSS 11.5软件完成,以P<0.05为差异有统计学意义。
结 果
1TNFR基因各SNP检测结果
在所有研究对象中,除TNFR1+36A/G未发现突变纯合子外,TNFR1-609G/T、TNFR2+676T/G、TNFR2+1663T/G、TNFR2+1668A/G与TNFR2 +1690C/T均发现野生纯合子与杂合子。TNFR1 -609G/T、TNFR1 +36A/G与TNFR2 +676T/G的基因型判断参照表2,上述不同基因型所在PCR片段经酶切后进行琼脂糖电泳分析,杂合子的基因型将被酶切为3段核苷酸片段,酶切产物电泳示意图见图1。TNFR2+1663、+1668和+1690基因测序结果示意图见图2。
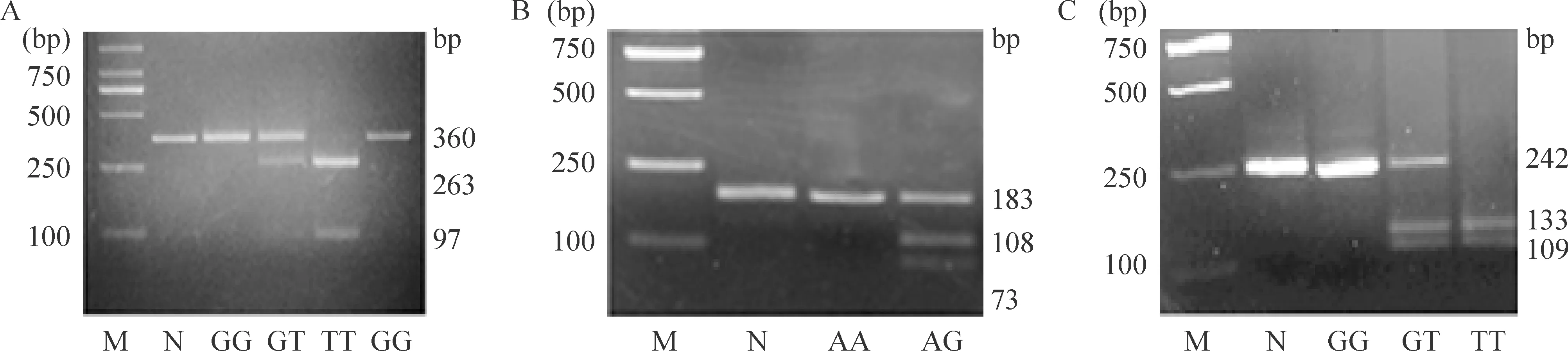
Figure 1. Restriction enzyme analysis ofTNFR1-609(A),TNFR1+36 (B) andTNFR2+676 (C) PCR products.M: DNA marker;N:normal control (wild type).
图1TNFR1-609、TNFR1+36与TNFR2+676的PCR片段酶切分析
2TNFR基因多态性在CAP组和对照组中的分布
TNFR1-609GG、GT与TT基因型在CAP组中分布频率分别为18.2%、45.5%与36.3%,在对照组中的分布频率分别为30.3%、47.0%与22.7%,上述3个基因型在两组中的分布差异无统计学意义(P>0.05)。TNFR1 -609G与T等位基因在CAP组中分布频率分别为40.9%与59.1%,在对照组中的分布频率分别为53.8%与46.2%,T等位基因在CAP组中的分布频率较高,其差异具统计学意义(P<0.05),见表3。TNFR1+36、TNFR2+676、TNFR2 +1663、TNFR2+1668与TNFR2+1690的各基因型与等位基因在CAP组与对照组中的分布频率差异差异无统计学意义,见表3。
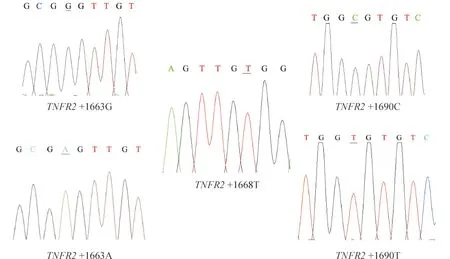
Figure 2. The schematic diagrams of sequencing ofTNFR2 +1663, +1668 and +1690.
图2TNFR2+1663、+1668和+1690基因测序结果示意图
3TNFR基因多态性在SCAP组和NSCAP组中的分布
TNFR1-609GG、GT与TT基因型在SCAP组中分布频率分别为0.0%、50.0%与50.0%,在NSCAP组中的分布频率分别为24.0%、44.0%与32.0%,上述3个基因型在两组中的分布差异无统计学意义(P>0.05)。TNFR1-609G与T等位基因在SCAP组中分布频率分别为25.0%与75.0%,在NSCAP组中的分布频率分别为46.0%与54.0%,T等位基因在SCAP组中的分布频率较高,其差异有统计学意义(P<0.05)。
TNFR2+1690CC、CT与TT基因型在SCAP组中分布频率分别为62.5%、37.5%与0.0%,在NSCAP组中的分布频率分别为40.0%、42.0%与18.0%,上述3个基因型在两组中的分布差异无统计学意义(P>0.05)。TNFR2+1690C与T等位基因在SCAP组中分布频率分别为81.1%与18.9%,在NSCAP组中的分布频率分别为61.0%与39.0%,C等位基因在SCAP组中分布频率较高,其差异有统计学意义(P<0.05)。
TNFR1+36、TNFR2+676、TNFR2+1663与TNFR2+1668的各基因型与等位基因在SCAP组与NSCAP组中的分布频率差异无统计学意义,见表4。
表3TNFR基因多态性在CAP组和对照组中的分布
Table 3. Genotype and allele frequencies ofTNFRgene polymorphisms between CAP group and control group
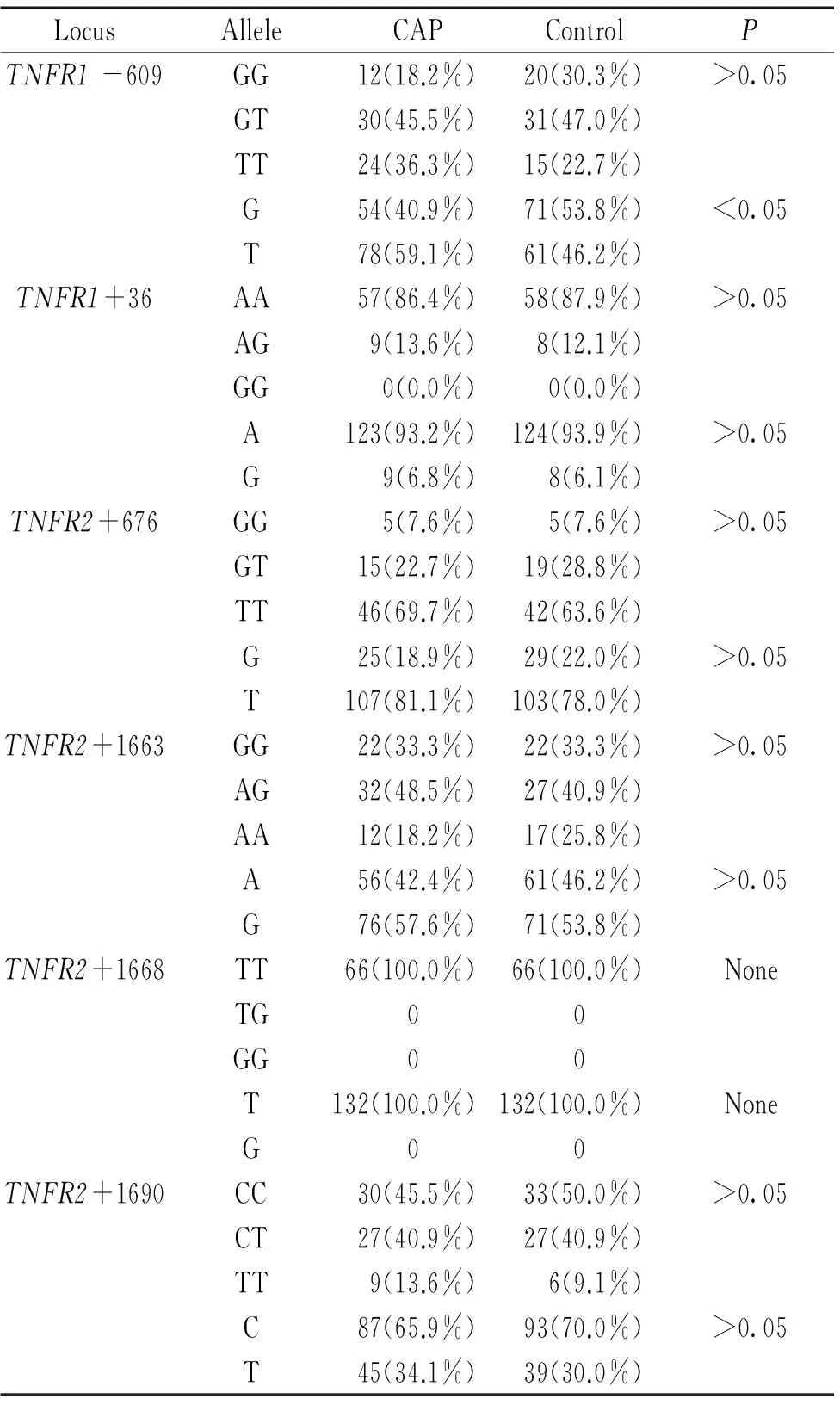
LocusAlleleCAPControlPTNFR1-609GG12(18.2%)20(30.3%)>0.05GT30(45.5%)31(47.0%)TT24(36.3%)15(22.7%)G54(40.9%)71(53.8%)<0.05T78(59.1%)61(46.2%)TNFR1+36AA57(86.4%)58(87.9%)>0.05AG9(13.6%)8(12.1%)GG0(0.0%)0(0.0%)A123(93.2%)124(93.9%)>0.05G9(6.8%)8(6.1%)TNFR2+676GG5(7.6%)5(7.6%)>0.05GT15(22.7%)19(28.8%)TT46(69.7%)42(63.6%)G25(18.9%)29(22.0%)>0.05T107(81.1%)103(78.0%)TNFR2+1663GG22(33.3%)22(33.3%)>0.05AG32(48.5%)27(40.9%)AA12(18.2%)17(25.8%)A56(42.4%)61(46.2%)>0.05G76(57.6%)71(53.8%)TNFR2+1668TT66(100.0%)66(100.0%)NoneTG00GG00T132(100.0%)132(100.0%)NoneG00TNFR2+1690CC30(45.5%)33(50.0%)>0.05CT27(40.9%)27(40.9%)TT9(13.6%)6(9.1%)C87(65.9%)93(70.0%)>0.05T45(34.1%)39(30.0%)
表4TNFR基因多态性在SCAP组和NSCAP组中的分布
Table 4. Genotype and allele frequencies ofTNFRgene polymorphisms between SCAP group and NSCAP group
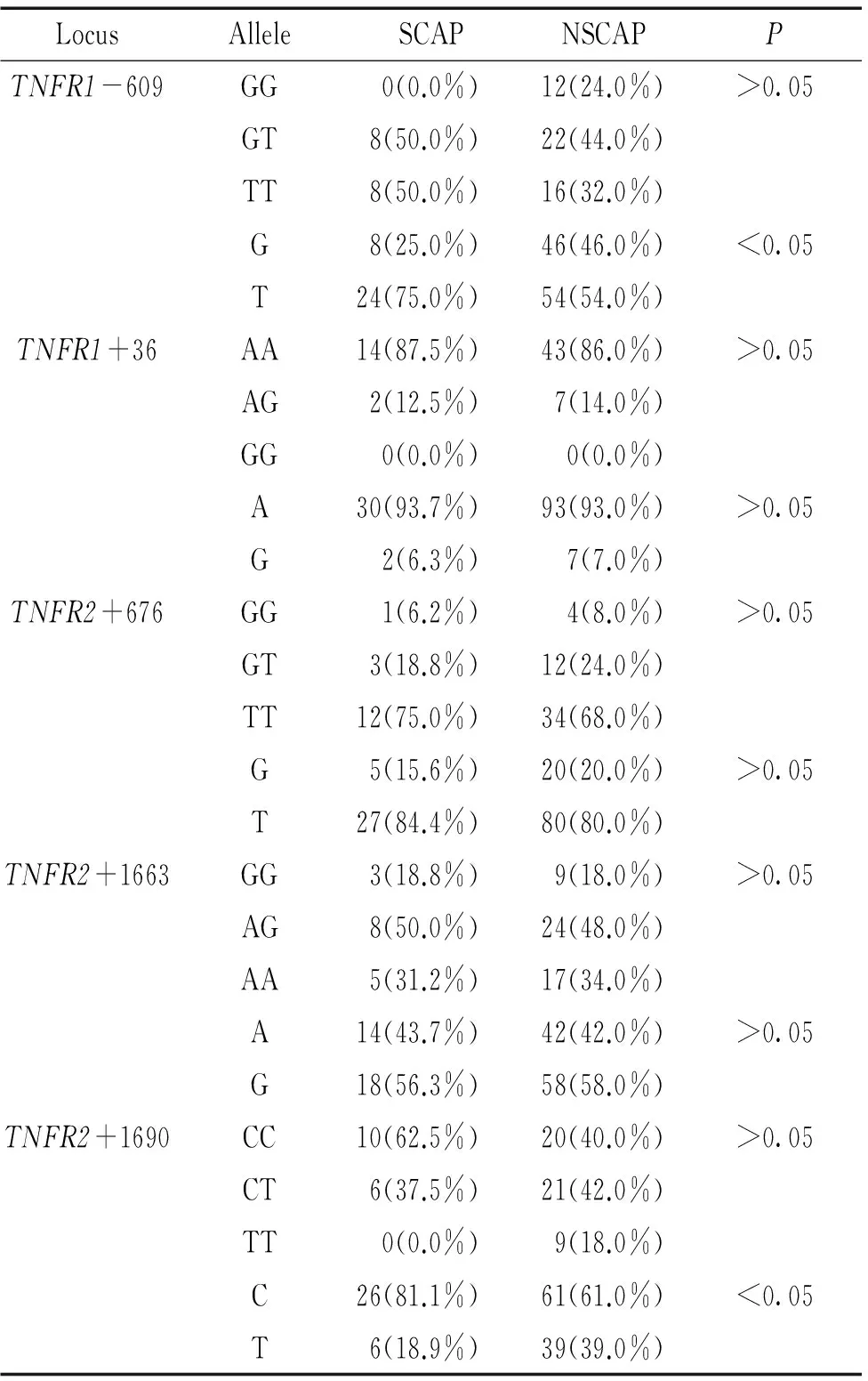
LocusAlleleSCAPNSCAPPTNFR1-609GG0(0.0%)12(24.0%)>0.05GT8(50.0%)22(44.0%)TT8(50.0%)16(32.0%)G8(25.0%)46(46.0%)<0.05T24(75.0%)54(54.0%)TNFR1+36AA14(87.5%)43(86.0%)>0.05AG2(12.5%)7(14.0%)GG0(0.0%)0(0.0%)A30(93.7%)93(93.0%)>0.05G2(6.3%)7(7.0%)TNFR2+676GG1(6.2%)4(8.0%)>0.05GT3(18.8%)12(24.0%)TT12(75.0%)34(68.0%)G5(15.6%)20(20.0%)>0.05T27(84.4%)80(80.0%)TNFR2+1663GG3(18.8%)9(18.0%)>0.05AG8(50.0%)24(48.0%)AA5(31.2%)17(34.0%)A14(43.7%)42(42.0%)>0.05G18(56.3%)58(58.0%)TNFR2+1690CC10(62.5%)20(40.0%)>0.05CT6(37.5%)21(42.0%)TT0(0.0%)9(18.0%)C26(81.1%)61(61.0%)<0.05T6(18.9%)39(39.0%)
讨 论
多项研究已证实重症肺炎患者体内存在炎症反应失衡。本实验室既往的动物实验已证实重症肺炎大鼠体内存在促炎细胞因子的过度释放与抑炎细胞因子的表达不足[3-4]。后续研究证实TNF-α启动子区单核苷酸多态性与肺炎的易感性、重症肺炎的发生率有关[5]。我们对TNF-α的后续扩展研究发现,重症肺炎时TNF-α/NF-κB形成正反馈环路,放大炎症反应,当抑制TNF-α信号后,上述正反馈环路中断,肺内炎症损伤减轻[6-7]。因此,TNF-α信号与肺内炎症反应强度密切相关,并可能影响肺炎的进一步恶化。
TNFR接受TNF-α的信号而启动细胞的炎症反应。TNF-α作为一种重要的炎症介质,与其受体TNFR结合,启动细胞内的炎症反应,二者表达量或活化程度的改变均可影响炎症反应的强度。TNFR主要分两型,TNFR1与TNFR2,前者由TNFRSF1A编码,后者由TNFRSF1B编码。TNFR1广泛存在于有核细胞表面,其胞内段具有死亡结构域,可通过与TNF受体相关死亡结构域(TNF receptor-associated death domain,TRADD)相结合,激活半胱天冬酶及活化NF-κB,引起炎性因子、趋化因子的转录[8]。TNFR2主要在淋巴细胞表达,其胞内段无死亡结构域,但可通过TNF受体相关因子2(TNF receptor-asso-ciated factor 2,TRAF2)介导细胞凋亡与NF-κB的激活。TNFR2与TNF-α的亲合力较高,即使较低的浓度也可以与TNF-α结合[9]。
TNFR与炎症反应关系密切,其基因的改变也将从TNFR表达水平或功能上影响炎症反应强度。已知TNFRSF1A基因与TNFRSF1B基因均存在多个基因多态性位点。有研究表明在伴有血液疾病的侵袭性肺曲霉病患者中,如携带TNFRSF1A+36G或-609G等位基因则易于罹患侵袭性肺曲霉病[10]。Cipriano等[11]则发现在CAP患者中,携带TNFRSF1B+676TG基因型的患者比携带TT或GG基因型患者死亡率低。但Gordon等[12]的研究却认为TNFRSF1A-609G/T、+36A/G,TNFRSF1B+676T/G、+1663A/G基因多态性与脓毒症的易感性、严重程度不相关。
TNFR基因的多态性是否影响炎症反应强度进而影响肺炎的的严重性或机体对肺炎的易感性仍存争论,探讨TNFR基因多态性与肺炎的相关性,是更好地利用基因进行疾病严重性与预后判断的基础。本研究通过PCR-RFLP及基因测序的方法检测了66例成年肺炎患者与66例健康体检者TNFR基因上多个多态性位点的等位基因分布情况,并分析TNFR基因多态性与肺炎严重程度、肺炎易感性的相关性。
在本研究中,我们发现TNFR1-609T等位基因在肺炎患者与重症肺炎中的分布频率较高,提示TNFR1-609T与肺炎的易感性、病情严重程度有关。目前已有研究证实TNFR1-609G/T多态性位点与侵袭性肺曲霉病有关,其机制是TNFR1-609位点G到T的变异将影响ICSBP等转录因子与TNFR基因的结合能力,进而影响TNFR1的表达强度[10]。另有研究证实携带 TT 基因型患者的TNFR1 mRNA转录水平比携带GT、GG基因型高[13]。因此我们认为携带TNFR1-609T等位基因的肺炎患者体内表达更多的TNFR1,TNF-α的结合位点增多,炎症信号相应更易向细胞内转导,故此类个体更易罹患肺炎,且当此类患者罹患肺炎后,体内炎症反应强度也较剧烈,易进展为重症肺炎。
本研究还发现TNFR2 +1690C等位基因的分布频率在肺炎患者与健康体检者中的分布频率无明显差异,但在重症肺炎患者中的分布频率较高,提示TNFR2+1690C等位基因影响炎症性疾病严重程度。而Puga等[14]的研究却有不同结果,认为TNFR2 +1690T/C多态性并不影响TNFR2的表达强度。我们已知膜蛋白的结构将影响其下传信号的强度,甚至不需结合配体即可启动下游的信号通路,因此我们推测编码区中TNFR2 +1690T到C的变异通过影响TNFR的活性而使机体对TNF-α更“敏感”,从而加重炎症性疾病的严重程度。此外,有研究认为TNFR2 +1690T/C与TNFR mRNA的稳定性有关,故携带TNFR2+1690C的肺炎患者炎症反应时间延长,这可能是导致病情加重、迁延的重要原因之一。
基因多态性伴随机体的一生,不受机体生理与病理状态影响。通过对稳定的基因多态性进行检测以准确评估机体对疾病的反应性,而准确地评估则建立在明确基因多态性与疾病相关性的基础上。我们的研究发现携带TNFR1-609T等位基因的患者肺炎及重症肺炎发病率高,携带TNFR2+1690C等位基因的肺炎患者易于发展为重症肺炎,提示可通过对上述基因多态性的检测判断机体对肺炎易感性与严重程度进行预测,以尽早干预,提高重症病人救治成功率。
[1] O’Keefe GE, Hybki DL, Munford RS. The G→A single nucleotide polymorphism at the -308 position in the tumor necrosis factor-alpha promoter increases the risk for severe sepsis after trauma[J]. J Trauma, 2002, 52(5):817-825.
[2] Mandell LA, Wunderink RG, Anzueto A, et al. Infectious Diseases Society of America/American Thoracic Society consensus guidelines on the management of community-acquired pneumonia in Adults[J]. Clin Infect Dis, 2007, 44(Suppl 2):S27-S72.
[3] 陈业民, 黄文杰, 李胜利, 等. 重症肺炎大鼠干扰素-γ、白细胞介素-6和肿瘤坏死因子-α含量变化[J]. 中国病理生理杂志, 2007, 23(3):492-494.
[4] 李胜利, 黄文杰. 地塞米松对SD大鼠肺炎克雷伯杆菌性重症肺炎模型血清白细胞介素-8、10的影响[J]. 实用医学杂志, 2007, 23(17):2634-2635.
[5] 袁伟锋, 黄文杰, 梁 坤.TNF-α基因单核苷酸多态性与肺炎的相关性研究[J]. 中国病理生理杂志, 2008, 24(12):2391-2395.
[6] 吴炜景, 李 理, 袁伟锋, 等. NF-κB p65基因在TNF-α诱导的肺泡上皮细胞氧化损伤中的作用[J]. 中国呼吸与危重监护杂志, 2012, 11(1):46-51.
[7] 袁伟锋, 李 理, 徐 虹, 等. TNFR-Fc减轻 LPS所致小鼠 ALI炎症反应损伤[J]. 中国病理生理杂志, 2011, 27(12):2387-2390.
[8] Jones SJ, Ledgerwood EC, Prins JB, et al. TNF recruits TRADD to the plasma membrane but not the trans-Golgi network, the principal subcellular location of TNF-R1[J]. J Immunol, 1999, 162(2):1042-1048.
[9] Rothe M, Wang SC, Henzel WJ, et al. A novel family of putative signal transducers associated with the cytoplasmic domain of the 75 kDa tumor necrosis factor receptor[J]. Cell, 1994, 78(4):681-692.
[10] Sainz J, Salas-Alvarado I, López-Fernández E, et al.TNFR1 mRNA expression level andTNFR1 gene polymorphisms are predictive markers for susceptibility to develop invasive pulmonary aspergillosis[J]. Int J Immunopathol Pharmacol, 2010, 23(2):423-436.
[11] Cipriano C, Caruso C, Lio D, et al. The -308G/A polymorphism of TNF-α influences immunological parameters in old subjects affected by infectious diseases[J]. Int J Immunogenet, 2005, 32(1):13-18.
[12] Gordon AC, Lagan AL, Aganna E, et al. TNF and TNFR polymorphisms in severe sepsis and septic shock:a prospective multicentre study[J]. Genes Immun, 2004, 5(8):631-640.
[14] Puga I, Lainez B, Fernández-Real JM, et al. A polymorphism in the 3’ untranslated region of the gene for tumor necrosis factor receptor 2 modulates reporter gene expression[J]. Endocrinology, 2005, 146(5):2210-2220.
RelationshipbetweenSNPofTNFRgeneandincidence/severityofpneumonia
LI Li1, ZHENG Shao-qiang2, YUAN Wei-feng1, HUANG Wen-jie1
(1DepartmentofRespiratoryMedicine,GuangzhouGeneralHospitalofGuangzhouMilitaryCommand,Guangzhou510010,China;2DepartmentofRespiratoryMedicine,theThirdAffiliatedHospitalofSouthernMedicalUniversity,Guangzhou510630,China.E-mail:huangyelu1029@vip.163.com)
AIM: To analyze the relationship between the single nucleotide polymorphism (SNP) of tumor necrosis factor receptor (TNFR) gene and the incidence and severity of pneumonia.METHODSTotal 132 Chinese individuals were enrolled in this study. There were 66 patients with pneumonia and 66 healthy subjects. The SNPs ofTNFRgene includingTNFR1+36A/G,TNFR1-609G/T,TNFR2+676T/G,TNFR2+1663T/G,TNFR2 +1668A/G andTNFR2 +1690C/T were genotyped by polymerase chain reaction-restriction fragment length polymorphism or gene sequencing for all subjects. Polymorphisms affecting pneumonia incidence and severity were calculated by SPSS.RESULTSThe frequencies ofTNFR1-609G and T alleles in pneumonia patients were 40.9% and 59.1%, while those in healthy subjects were 53.8% and 46.2%. The frequency ofTNFR1-609T in pneumonia patients was higher than that in healthy subjects (P<0.05). Besides, the frequencies ofTNFR1-609G and T alleles in severe pneumonia patients were 25.0% and 75.0%, while those were 46.0% and 54.0% in non-severe pneumonia patients. The frequencies ofTNFR2 +1690C and T alleles in severe pneumonia patients were 81.1% and 18.9%, while those were 61.0% and 39.0% in non-severe pneumonia patients. The frequencies ofTNFR1-609T andTNFR2 +1690C in severity pneumonia subjects were higher than those in mild subjects (P<0.05).CONCLUSIONIt appears thatTNFR1-609T is associated with high incidence of pneumonia.TNFR1-609T andTNFR2+1690C are the risk factors of severity in pneumonia in Chinese.
Receptors,tumor necrosis factor; Polymorphism,single nucleotide; Pneumonia
R363
A
10.3969/j.issn.1000- 4718.2013.04.021
1000- 4718(2013)04- 0695- 06
2012- 11- 16
2013- 02- 22
广东省自然科学基金资助项目(No.10151001002000005)
△通讯作者 Tel: 020-36653555; E-mail: huangyelu1029@vip.163.com
