帕金森病相关血清蛋白质标志物的筛选及鉴定*
2013-10-25刘池波徐小辉梁海燕
周 婷, 刘池波, 徐小辉, 梁海燕
(台州学院医学院附属台州市立医院 1神经内科, 2检验科,浙江 台州 318000)
帕金森病相关血清蛋白质标志物的筛选及鉴定*
周 婷1△, 刘池波2, 徐小辉1, 梁海燕1
(台州学院医学院附属台州市立医院1神经内科,2检验科,浙江 台州 318000)
目的探讨并初步鉴定帕金森病患者血清中相关蛋白质作为特异标志物的可能性。方法应用表面增强激光解吸电离飞行时间质谱(SELDI-TOF-MS)结合纳米磁珠技术检测44例帕金森病和60例健康对照的血清标本,应用生物信息学方法筛选差异蛋白峰,经高效液相色谱(HPLC)分离出差异蛋白,酶解后进行液质联用串联质谱(LC-MS/MS)分析,利用Xcalibur的程序组件BioWorks 3.2完成蛋白质序列数据库鉴定分析。结果经SELDI-TOF-MS结合纳米磁珠技术筛选出质荷比m/z位于8 937的蛋白质在帕金森病中高表达(帕金森病组表达强度为27.47±16.58,正常组表达强度为5.01±3.47),有显著差异(P<0.01);6 636和8 697的蛋白质在帕金森病中低表达(帕金森病组表达强度为5.43±2.66和20.22±9.57,正常组表达强度为18.85±7.56和51.13±26.22), 有显著差异(P<0.01)。联合上述3种潜在蛋白质标志物,可区分帕金森病组和对照组,其中帕金森病患者检出率为90.0%(27/30),健康者检出率为92.5%(37/40)。对m/z为6 636、8 697和8 937的标志物进行鉴定,结果分别为载脂蛋白C-I、载脂蛋白 C-III 和补体成分3a。结论鉴定出的载脂蛋白 C-I、载脂蛋白C-III和补体成分3a在帕金森病的诊断中具有一定价值,值得进一步研究和探讨。
帕金森病; 蛋白质组; 表面增强激光解吸电离飞行时间质谱法; 液相色谱-质谱联用串联质谱
帕金森病(Parkinson’s disease,PD),又名震颤麻痹(shaking palsy),是一种中老年人常见的缓慢进展的神经系统疾病,是中老年人致残的主要原因之一。临床上以静止性震颤、运动迟缓、肌强直和姿势步态障碍为主要特征,患病率与发病率随着年龄的增加而递增[1]。目前临床上诊断帕金森病尚无特异敏感的生化指标和影像学检查可作为确诊的依据。所以诊断主要依据临床症状和对左旋多巴治疗的反应,但这些方法往往只能发现中晚期病例,且容易漏诊和误诊。目前帕金森病的治疗主要在于改善运动症状,而非延缓疾病的进程,所以早期诊断帕金森病,为帕金森病的早期干预及延缓病损提供了可能。因此,探索和建立一种简单、快速、敏感性高和准确性强的帕金森病快速诊断技术已经成为临床医学上亟待解决的问题。表面增强激光解吸电离飞行时间质谱(surface-enhanced laser desorption ionization time-of-flight mass spectrometry,SELDI-TOF-MS) 是近年来出现的一种新型的蛋白质组学研究方法[2-4],可用于直接检测临床各种类型标本,如血清、胸腹水、尿液、脑脊液等,实现了质谱技术用于临床标本检测的飞跃[5-6]。我们利用SELDI-TOF-MS结合纳米磁珠技术对44例帕金森病和60例健康对照的血清标本进行差异蛋白筛选,再结合高效液相色谱(high-performance liquid chromatography,HPLC)、基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption ionization time-of-flight mass spectrometry,MALDI-TOF-MS)及液质联用串联质谱(liquid chromatograph-mass spectrometry tandem mass spectrometry,LC-MS/MS)对帕金森病差异蛋白进行鉴定分析。
材 料 和 方 法
1研究对象
收集2011年1月~2012年2月我院住院及门诊的PD患者44例,男性24例,女性20例,年龄(67.41±12.87)岁(54~82岁);病程(3.25±4.36)年(0.5~16年)。入选标准:(1)符合2006年中华医学会神经病学分会运动障碍及PD学组所制订的诊断标准[7];(2)均经CT或MRI检查,除有老年性脑改变外,未见特殊异常;(3)排除脑血管疾病、脑炎等原因所导致的帕金森综合征。所有帕金森病患者和健康者血清均来自浙江省台州市立医院,随机分成实验组与验证组(training set为实验组,blind set为验证组,见表1)。本实验经本院医学伦理委员会批准,受试者均签署知情同意书。所有外周血标本均在清晨空腹采血,采集后立即放入4 ℃冰箱内静置1~2 h,然后4 ℃ 3 000 r/min离心10 min,分离血清后,在冰上将血清转移到新的PCR管中分装,放入-80 ℃内冰箱保存待检。
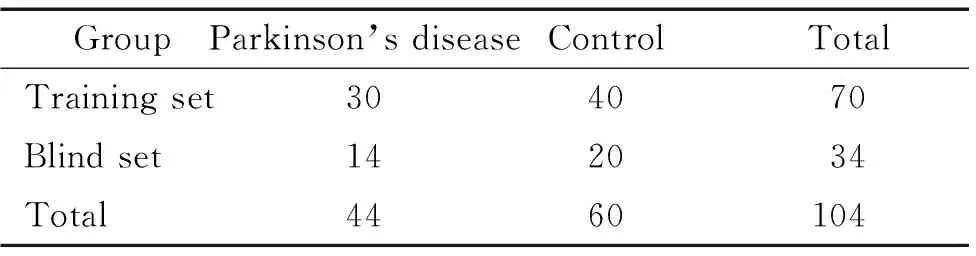
表1 44例帕金森病患者和60例对照组分组情况
2仪器和试剂
乙腈(acetonitrile,ACN)、三氟乙酸(trifluoroacetic acid,TFA)、芥子酸(sinapinic acid,SPA)、尿素、二硫苏糖醇(DL-dithiothreitol,DTT)、3-[3-(胆酰氨丙基)二甲氨基]丙磺酸内盐{3-[(3-cholamidopropyl)dimethylammonio]-1-propanesulfonate,CHAPS}、Tris-HCl、H2O等购自Sigma,SELDI-TOF-MS (PBS IIC)质谱仪购自Ciphergen Biosystems,弱阳离子交换型(WCX)纳米磁珠、结合缓冲液、洗脱液等购自北京赛尔迪公司。MALDI-TOF-MS质谱仪(AXIMA-CFRTMPlus)购于Kratos Analytical,HPLC购于Shimadzu,Xcalibur的程序组件BioWorks 3.2数据库购自Thermo Finnigan。
3帕金森病SELDI-TOF-MS分析
将WCX纳米磁珠分装于PCR管中,置于磁性处理器上,吸取液体。再依次加入100 μL结合缓冲液,混匀后放置5 min,然后把PCR管置于磁性处理器上,吸取液体,重复上述操作1次。向每个装有纳米磁珠的PCR管中加入100 μL稀释好的血清样品,混匀后室温放置15 min。将PCR管置于磁性处理器上,吸去未结合样品。每管加入100 μL结合缓冲液,混匀后放置5 min,然后将PCR管置于磁性处理器上,吸取液体,重复上述操作1次。每管加入10 μL洗脱液混匀,放置5 min后置于磁性处理器上。取5 μL上清液移到另一个PCR管中,加人5 μL饱和SPA溶液充分混匀,吸取1 μL点样到Au芯片上,待干后放入仪器读取。SPA饱和溶液为芥子酸在50% ACN和0.5% TFA的饱和溶液。质谱仪参数设定为激光强度190,灵敏度8,优化范围2 000~20 000质荷比(mass/charge rato,m/z)。每条芯片取1点用同一正常人血清作内参照,芯片间 CV≤10%。检测前用ALL-IN-ONE多肽标准芯片校正,系统质量偏差≤0.1%。原始数据先以ProteinChip 3.0软件校正。
4蛋白质的纯化分析
将血清溶解后取出100 μL血清,加入350 μL水,700 μL纯乙腈。将上述混合液体置入-20 ℃的冰箱内,30 min后取出,3 000 r/min离心10 min。上清液转入PCR管中冻干20 min。将冻干的样品上样至HPLC中。收集不同时点的纯化液体,将纯化出的蛋白液置入PCR管中冻干约20 μL。各取所收集组分0.5 μL,分别加入1.5 μL基质溶液[10 g/Lα-氰基-4-羟基肉桂酸(α-cyano-4-hydroxycinnamic acid, CHCA)],混合后点于靶板上,静置结晶、干燥后用MALDI-TOF-MS检测。检测条件:脉冲氮激光(337 nm)作为离子解吸电离源,加速电压20 kV,采用线性分析模式,质量范围:3 000~20 000 (m/z)。找出SELDI-TOF-MS所筛选质荷峰值的特异样本。
5目标蛋白质的LC-MS/MS分析
取20 μL纯化的目标蛋白质,加60 μL 8 mol/L尿素,使尿素的终浓度为6 mol/L,室温振荡20 min;加入0.8 μL 1 mol/L DTT,使其终浓度为10 mol/L,室温振荡1 h;再加入3.2 μL 1 mol/L碘乙酰胺,使其终浓度为40 mol/L,避光放置45 min;加入3.2 μL 1 mol/L DTT,终浓度为40 mol/L,振荡20 min;加入400 μL 50 mol/L NH4HCO3稀释样品溶液,使尿素终浓度降为1 mol/L,pH 约8.0。每支样品中加入0.1 μg蛋白酶K,37 ℃水浴1 h,加入甲酸调溶液pH<3,终止酶解反应。目标蛋白的酶解产物用LC-MS/MS进行分析。样品溶液可以通过特定装置直接上样于自制的C18毛细管液相色谱柱。色谱柱及色谱条件:自制毛细管柱内径为100 μm,填料部分长100 mol/L,填料粒径5 μm。流动相A:水,0.1%甲酸;流动相B:乙腈,0.1%甲酸;洗脱程序:100% A(0 min)→100%A (5 min)→5% B(5.1min)→65%B(60 min) →100% B (75 min) →100% B (85 min);流速:200~800 nL/min。质谱条件:采用数据依赖模式(data-dependent mode),扫描质量范围:400~2 000 (m/z) ,每次选取全扫描(full scan)中5个信号最强的离子峰(母离子)进行二级质谱(MS2)分析。
6LC-MS/MS分析数据的检索
数据检索利用Xcalibur的程序组件BioWorks 3.2完成,根据二级离子谱图在NCBI的人类蛋白数据库中检索肽段序列。检索参数设置见参考文献[8]。
7统计学处理
帕金森病SELDI蛋白指纹图谱用Biomarker Wizard 3.1软件和Biomarker Patterns 4.0.1软件进行分组及相关性分析,帕金森病组与对照组之间蛋白质峰强度比较采用t检验,以P<0.05为差异有统计学意义。
结 果
1帕金森病血清蛋白指纹图谱分析
30例帕金森病患者和40例对照者血清的蛋白指纹图谱经标准化后,用 Biomarker Wizard软件分析,在分子质量2 000~20 000范围内共检测到143个蛋白峰,见图1。
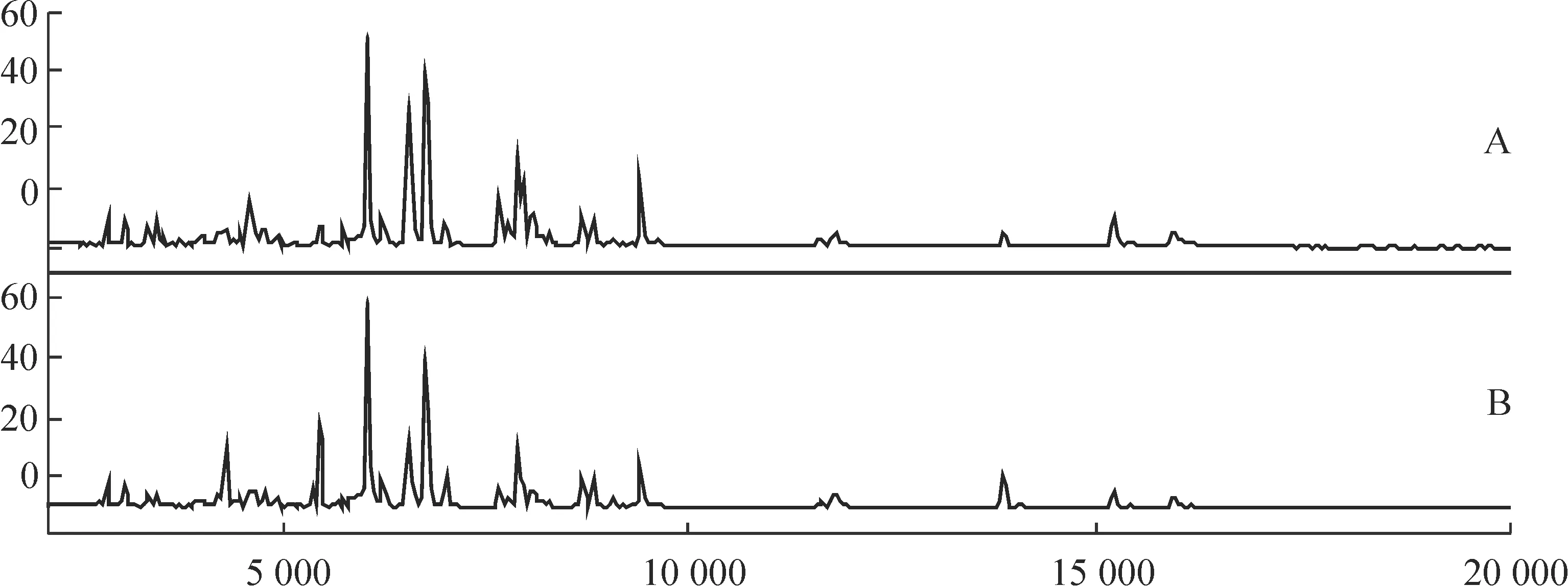
Figure 1. Representative protein spectrum of a Parkinson disease sample detected by SELDI-TOF-MS combined with WCX magnetic beads,showing the protein mass/charge ratio between 2 000 and 20 000.A:healthy control; B: Parkinson’s disease.
图1经SELDI-TOF-MS结合WCX纳米磁珠检测帕金森病的血清蛋白质指纹表达图谱
2帕金森病血清蛋白指纹图谱数据分析及帕金森病诊断模型的建立
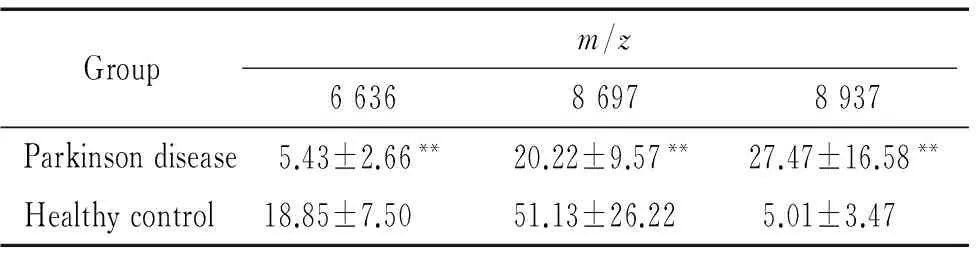
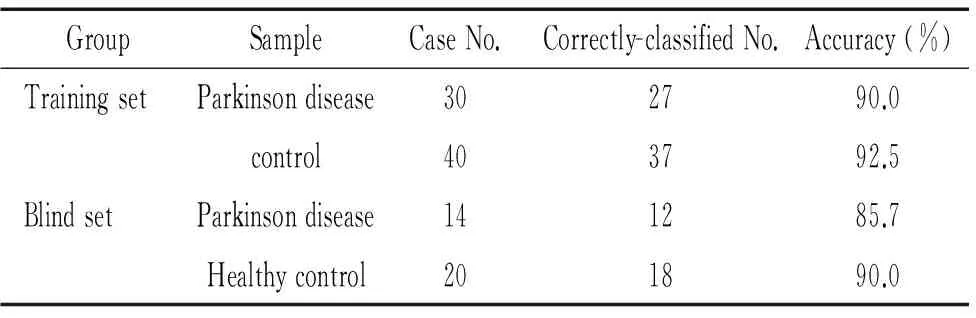
经Biomarker Patterns软件进一步分析显示,帕金森病患者外周血清蛋白质表达谱与对照组相比,m/z为6 636、8 697和8 937的3个蛋白峰的差异有统计学意义(P<0.01),其中m/z为8 937的蛋白峰在帕金森病组中明显高于对照组,而m/z为6 636和8 697的蛋白峰则在对照组中相对高表达(P<0.01),见表2。用此模型分析30例帕金森病患者与40例对照者的质谱结果,帕金森病患者检出率为90.0%(27/30),健康者检出率为92.5%(37/40),见表3。根据差异蛋白及其特定组合,理论上可以区分帕金森病组与对照组。
表2帕金森病组与对照组血清蛋白质峰强度的比较
Table 2. Conparison of signal intensity of various serum protein peaks between Parkinson’s disease and healthy control
Groupm/z663686978937Parkinsondisease5.43±2.66**20.22±9.57**27.47±16.58**Healthycontrol18.85±7.5051.13±26.225.01±3.47
**P<0.01vshealthy control.
3诊断模型的验证
采用14例帕金森病患者与20例对照者检测得到的蛋白指纹图谱对已建立的帕金森病诊断模型进行验证,验证结果表明其对帕金森病诊断的帕金森病患者检出率为85.7%(12/14),健康者检出率为90.0%(18/20),见表3。
表3帕金森病诊断模型的诊断特性
Table 3. Prediction results of the diagnostic model for Parkinson’s disease
GroupSampleCaseNo.Correctly-classifiedNo.Accuracy(%)TrainingsetParkinsondisease302790.0control403792.5BlindsetParkinsondisease141285.7Healthycontrol201890.0
4差异蛋白质峰的纯化
将帕金森病的3个差异蛋白质峰(6 636、8 697和8 937)经HPLC分离纯化后,收集到不同的PCR管中,经MALDI-TOF-MS检测,发现3个纯化样本分别是相对分子质量为6 632.9、8 697.2和8 933.1的特异蛋白质,见图2。
5质谱分析及数据库检索
将上述3管的蛋白质样品酶解后,采用LC-MS/MS测定该蛋白质的酶解肽段,数据经过Xcalibur的程序组件BioWorks 3.2 完成,根据二级离子谱图在NCBI的人类蛋白数据库中检索肽段序列。最终鉴定m/z为6 636、8 697和8 937的标志物分别为载脂蛋白C-I、载脂蛋白 C-III 和补体3a(complement 3a,C3a),见表4。
讨 论
SELDI-TOF结合HPLC及LC-MS/MS技术组合是近年出现的一种新的蛋白质研究方法,它集临床蛋白质组学差异蛋白筛选、鉴定为一体的较理想的技术平台[9]。目前已在自身免疫性疾病[10]、感染性疾病[11]及恶性肿瘤[12-14]等多种疾病标志物筛选方面取得了突破性的进展,并且筛选出许多与疾病相关的新型生物标志物为临床疾病的诊断和治疗提供新的选择[15-16]。
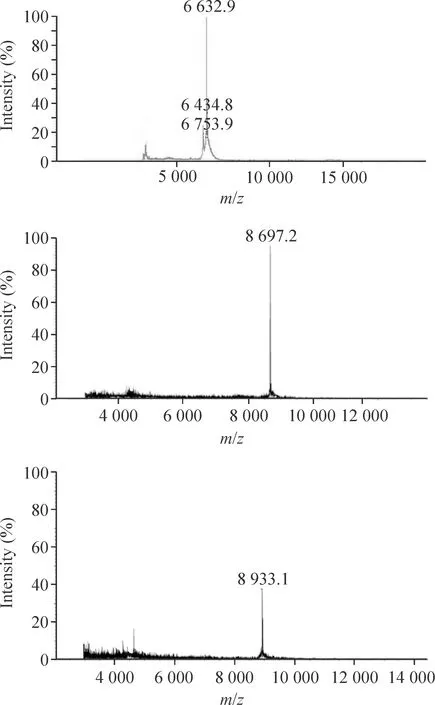
Figure 2. MALDI-TOF-MS atm/zof spectra of the three purified potential protein markers atm/zof 6 636, 8 697 and 8 937.
图2m/z6636、8697和8937的3个差异蛋白经蛋白纯化后的MALDI-TOF-MS图
在本研究中,我们通过SLEDI-TOF-MS技术结合WCX纳米磁珠筛选出m/z6 636、8 697和8 937的蛋白质标志物3个,其中8 937在帕金森病血清中高表达,6 636和8 697在帕金森病血清中低表达。由这3个标志物组合的诊断模型对帕金森病患者检出率为90.0%(27/30),健康者检出率为92.5%(37/40)。提示可以将这3个蛋白质标志物作为帕金森病血清学诊断的指标。通过本研究建立的诊断模型对14例帕金森病组与20例对照组进行验证,验证结果对帕金森病患者检出率为85.7%(12/14),健康者检出率为90.0%(18/20)。结合HPLC及LC-MS/MS技术对m/z为6 636、8 697和8 937的标志物进行鉴定,结果为载脂蛋白 C-I、载脂蛋白C-III 和C3a。
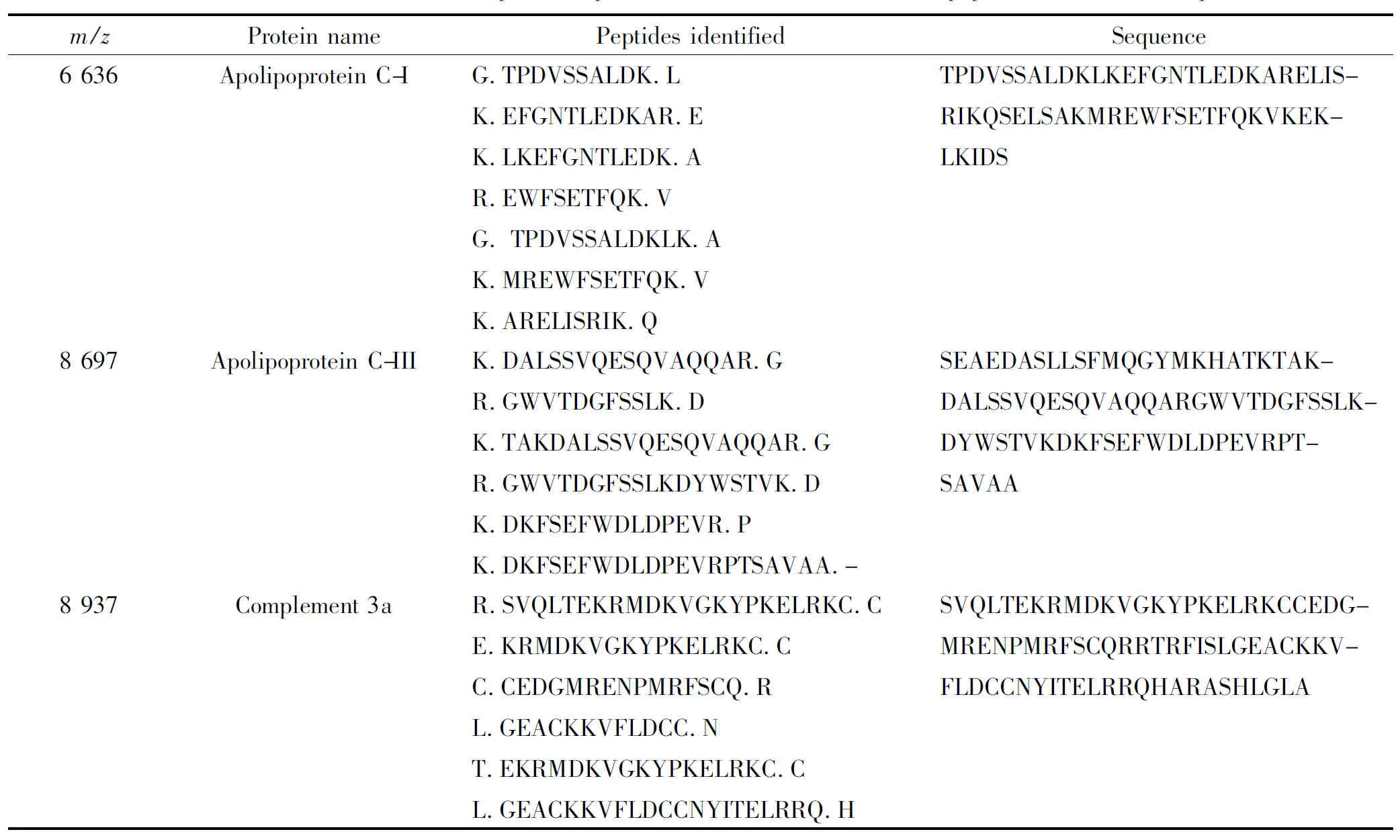
表4 帕金森病差异蛋白质的氨基酸序列
m/z为6 636的蛋白为载脂蛋白 C-I,主要是在肝脏合成,极少一部分是由小肠合成,其主要的生物学功能是参与脂类代谢。目前已有报道关于应用SELDI-TOF技术筛选胰腺癌[17]和非小细胞肺癌[18]患者血清中的差异蛋白,发现m/z6 636的蛋白在胰腺癌和非小细胞肺癌患者血清中低表达,并鉴定为载脂蛋白 C-I,与本研究中m/z6 636的蛋白为同一蛋白。但至今,国内外有关载脂蛋白 C-I与帕金森病的相关性研究报道甚少,有待进一步研究。m/z为8 697的蛋白为载脂蛋白C-III,载脂蛋白 C-III是由79个氨基酸残基组成的分子量为8 800的糖蛋白,主要在肝脏合成,其次在小肠。载脂蛋白C-III主要分布于富三酰甘油脂蛋白和高密度脂蛋白(HDL)中。目前已有报道载脂蛋白C-III是心血管疾病的重要危险因素,可引起脂质代谢异常,其直接参与致血管内皮细胞功能失调过程,几乎参与动脉粥样硬化形成的全过程,被视为一个独立的促炎和促粥样硬化因素。此外,载脂蛋白C-III还具有阻止肝细胞摄取含载脂蛋白E的乳糜微粒残体的作用。同时,Fan等[12]通过SELDI技术筛选乳头状甲状腺癌,发现m/z为8 697的蛋白在乳头状甲状腺癌中低表达,与本研究中m/z8 697的蛋白相同。载脂蛋白C-III在帕金森病患者血清中呈低表达,提示帕金森病的发生可能对载脂蛋白C-III的表达起抑制作用。这可能与帕金森病的发生对载脂蛋白C-III合成部位的损害有关,载脂蛋白C-III表达下调的原因有待于进一步研究。m/z为8 937的蛋白峰鉴定为C3a。补体具有调节炎症及免疫反应的功能,C3是由α和β链组成的,是血清中含量最丰富的补体成分,C3可裂解为C3a和C3b, C3a具有作为配体与细胞表面相应受体结合后,激发细胞脱颗粒,释放组胺之类的血管活性介质,从而增强血管通透性并刺激内脏平滑肌收缩的功能,是一种过敏毒素。同时可对免疫应答的各个环节发挥调节作用,可成为炎症的一种生物标志物。在本研究中,C3a在帕金森病中高表达,其机制尚不清楚,可能作为一种因子参与帕金森的发生与发展。
本研究鉴定出的载脂蛋白C-I、载脂蛋白C-III 和C3a在帕金森病诊断中的联合应用具有较高的准确性,该联合检测模型可作为帕金森病的一种新的诊断方法。但由于这3个蛋白分别在胰腺癌、非小细胞肺癌以及乳头状甲状腺癌中均有不同表现,并非帕金森病特有蛋白,因此在下一步研究中,我们将利用这3个蛋白组成的诊断模型扩大在帕金森病患者中的临床早期诊断应用,并将该模型用于帕金森病的疗效观察中,以了解该模型在帕金森病诊断、疗效观察中的更多临床意义。
[1] Pahwa R, Lyons KE. Early diagnosis of Parkinson’s disease: recomendations from diagnostic clinical guidelines[J]. Am J Manag Care,2010,16(4):S94-S99.
[2] Xiao X, Zhao X, Liu J, et al. Discovery of laryngeal carcinoma by serum proteomic pattern analysis[J]. Sci China C Life Sci, 2004,47(3):219-223.
[3] Pang RT, Poon TC, Chan KC, et al. Serum proteomic fingerprints of adult patients with severe acute respiratory syndrome[J]. Clin Chem, 2006, 52(3):421-429.
[4] Liang Y, Fang M, Li J, et al. Serum proteomic patterns for gastric lesions as revealed by SELDI mass spectrometry[J]. Exp Mol Pathol, 2006, 81(2):176-180.
[5] Zhang Q, Wang J, Dong R, et al. Identification of novel serum biomarkers in child nephroblastoma using proteomics technology[J]. Mol Biol Rep, 2011,38(1):631-638.
[6] Cho WC. Proteomics technologies and challenges[J]. Genomics Proteomics Bioinformatics, 2007, 5(2):77-85.
[7] 中华医学会神经病学分会运动障碍及帕金森病学组.帕金森病的诊断[J].中华神经科杂志,2006,39(6):408-409.
[8] 王伟平,刘池波.胃腺癌血清蛋白质标志物的筛选及鉴定[J].中国病理生理杂志,2012,28(2):274-280.
[9] Li J, Xie Z, Shi L,et al. Purification, identification and profiling of serum amyloid A proteins from sera of advanced-stage cancer patients[J]. J Chromatogr B Analyt Technol Biomed Life Sci, 2012,889-890:3-9.
[10] Li YZ, Hu CJ, Leng XM,et al. Promising diagnostic biomarkers for primary biliary cirrhosis identified with magnetic beads and MALDI-TOF-MS[J]. Anat Rec (Hoboken), 2009,292(3):455-460.
[11] Kanmura S, Uto H, Sato Y,et al. The complement component C3a fragment is a potential biomarker for hepatitis C virus-related hepatocellular carcinoma[J]. J Gastroenterol, 2010,45(4):459-467.
[12] Fan Y, Shi L, Liu Q,et al. Discovery and identification of potential biomarkers of papillary thyroid carcinoma[J]. Mol Cancer, 2009,8:79.
[13] Shi L, Zhang J, Wu P,et al. Discovery and identification of potential biomarkers of pediatric acute lymphoblastic leukemia[J]. Proteome Sci,2009,7: 7.
[14] Zhang Q, Wang J, Dong R,et al. Identification of novel serum biomarkers in child nephroblastoma using proteomics technology[J]. Mol Biol Rep, 2011,38(1):631-638.
[15] Liu C, Pan C, Shen J,et al. MALDI-TOF MS combined with magnetic beads for detecting serum protein biomarkers and establishment of boosting decision tree model for diagnosis of colorectal cancer[J]. Int J Med Sci, 2011, 8(1):39-47.
[16] Liu C. The application of SELDI-TOF-MS in clinical diagnosis of cancers[J]. J Biomed Biotechnol, 2011,2011:245821.
[17] Takano S, Yoshitomi H, Togawa A. Apolipoprotein C-1 maintains cell survival by preventing from apoptosis in pancreatic cancer cells[J]. Oncogene, 2008,27(20):2810-2822.
[18] Yang Y, Zhao S, Fan Y,et al. Detection and identification of potential biomarkers of non-small cell lung cancer[J]. Technol Cancer Res Treat, 2009, 8(6):455-466.
ScreeningandidentificationofspecificserumbiomarkersofParkinsondisease
ZHOU Ting1, LIU Chi-bo2, XU Xiao-hui1, LIANG Hai-yan1
(1DepartmentofNeurology,2DepartmentofClinicalLaboratory,TaizhouMunicipalHospitalAffiliatedtoTaizhouUniversitySchoolofMedicine,Taizhou318000,China.E-mail:dr_zhouting@yahoo.com.cn)
AIM: To screen the possible serum biomarkers of Parkinson’s disease.METHODSThe surface-enhanced laser desorption ionization time-of-flight mass spectrometry (SELDI-TOF-MS) was used to screen the serum samples from 44 cases of Parkinson’s disease and 60 control subjects. The differentially expressed protein peaks were selected and isolated with high-performance liquid chromatography (HPLC), and processed with enzyme before analysis by liquid chromatography-mass spectrometry tandem mass spectrometry (LC-MS/MS). The data mining was performed by Xcalibur program component BioWorks 3.2.RESULTSThree differentially expressed protein peaks were selected as potential serum biomarkers from the patients of Parkinson’s disease: the protein at 8 937m/zpeak showed significant increase (27.47±16.58 in Parkinson’s disease group, and only 5.01±3.47 in control group), and the proteins at 6 636 and 8 697m/zpeaks showed significant decreases (5.43±2.66 and 20.22±9.57, respectively, in Parkinson’s disease group, and 18.85±7.56 and 51.13±26.22, respectively, in control group). The proteins at 6 636, 8 697 and 8 937m/zpeaks were identified as apolipoprotein C-I, apolipoprotein C-III and complement 3a,respectively. Combined use of these 3 biomarkers effectively distinguished the subjects between Parkinson’s disease group and control group. The detection rate of the patients with Parkinson’s disease was 90.0% (27/30), and the detection rate of the healthy sibkects was 92.5% (37/40).CONCLUSIONThe apolipoprotein C-I, apolipoprotein C-III and complement component 3a identified as potential markers of Parkinson’s disease have diagnostic value in clinical application.
Parkinson disease; Proteome; Surface-enhanced laser desorption ionization time-of-flight mass spectrometry; Liquid chromatography-mass spectrometry tandem mass spectromery
R741.04
A
10.3969/j.issn.1000- 4718.2013.04.016
1000- 4718(2013)04- 0664- 06
2012- 08- 31
2013- 03- 04
台州市市级科技资金资助项目(No.102KY11)
△通讯作者 Tel: 0576-88858166; E-mail: dr_zhouting@yahoo.com.cn
