胚胎干细胞分化为表皮样细胞基因启动子区差异甲基化的研究*
2013-10-24张仁礼刘彩霞孟锦绣张丽丽蔡佳杰闻安民
张仁礼, 刘彩霞, 孟锦绣, 张丽丽, 韩 冬, 蔡佳杰, 闻安民
(广东省医学科学院,广东省人民医院 1生殖医学中心, 2医学研究中心,广东 广州 510080)
·论著·
胚胎干细胞分化为表皮样细胞基因启动子区差异甲基化的研究*
张仁礼1△, 刘彩霞1, 孟锦绣2, 张丽丽1, 韩 冬1, 蔡佳杰1, 闻安民1
(广东省医学科学院,广东省人民医院1生殖医学中心,2医学研究中心,广东 广州 510080)
目的用全基因组甲基化芯片探讨可能参与小鼠胚胎干细胞(ESCs)分化为表皮样细胞(ELCs)的基因DNA甲基化调控机制。方法利用人羊膜建立体外诱导体系,将小鼠ESCs诱导定向分化为ELCs,分别取未分化的ESCs和诱导分化后的ELCs进行芯片分析,利用甲基化免疫共沉淀技术将每组染色质DNA的甲基化片段共沉淀下来,和本底对照 (input)分别标记Cy3和Cy5荧光,一同上样于Roche NimbleGen高密度(2.1M,芯片覆盖22 425个启动子)甲基化芯片,启动子区采用UCSC数据库进行注释,启动子覆盖转录起始位点上游8 200 bp、下游3 000 bp,通过对这些启动子区甲基化谱的分析,筛选出甲基化调控可能与ESCs向ELCs定向分化相关的基因。结果小鼠ESCs和ELCs 两组细胞在基因组水平上有17 500个启动子存在甲基化,其中有3 435个启动子发生甲基化差异变化,高CpG岛的启动子有894个发生差异甲基化,中度CpG岛的启动子有974个发生差异甲基化,低CpG岛的启动子有1 567个发生差异甲基化。结论在ESCs向ELCs定向分化的过程中,众多的基因启动子区发生了甲基化程度的变化,说明细胞分化是一个复杂的表观遗传学事件。这些基因启动子的差异甲基化在ESCs向ELCs定向分化过程中所起的作用及其机制尚有待进一步的功能学研究阐明。
DNA甲基化; 胚胎干细胞; 表皮样细胞; 启动子; 甲基化DNA免疫共沉淀
在前期研究中,我们利用人羊膜接触或非接触均能将小鼠胚胎干细胞(embryonic stem cells, ESCs)定向诱导为表皮样细胞(epidermal-like cells, ELCs),分化后的ELCs呈现典型单层表皮样细胞形态改变,表达表皮特异标志物β1整合素(β1-integrin)、角蛋白19(cytokeratin 19, CK19)和角蛋白15(CK15)[1]。诱导后的细胞植入裸鼠皮下,可形成边界清晰的瘤体,瘤体内可见大量表皮样和表皮附属器样结构。这表明人羊膜能够将ESCs定向诱导分化为ELCs,诱导后的细胞具有分化为表皮及表皮附属器的潜能[2]。但在ESCs分化为ELCs的过程中,基因表达谱的变化和相关的表观遗传学调控机制等尚不清楚。本研究通过甲基化DNA免疫共沉淀(methylated DNA immunoprecipitation, MeDIP)与甲基化芯片(methylation microarray)技术相结合,比较诱导分化前后两组细胞样本间DNA甲基化的差异,快速高通量筛选出与定向分化相关关键基因,并分析这些基因的功能,以进一步阐述ESCs分化为表皮样干细胞的发生机制。
材 料 和 方 法
1材料
1.1主要试剂 DMEM(Dulbecco’s Modified Eagle’s Medium)、小鼠白血病抑制因子(leukemia inhibitory factor, LIF)、胰蛋白酶和胎牛血清均为Gibco产品;DNA纯化试剂盒(DNeasy Blood & Tissue Kit ,Qiagen);磁珠(BioMagTMmagnetic beads, Bangs laboratories);5-甲基胞嘧啶抗体[anti-5-methylcytidine(mouse) (1 g/L), Diagenode];小鼠IgG [mouse IgG (1 g/L), Jackson];酚/氯仿/异戊醇25∶24∶1;全基因组扩增试剂盒(Sigma);QIA快速聚合酶链反应(polymerase chain reaction, PCR)扩增试剂盒(QIAquick PCR purification kit,Qiagen);β-巯基乙醇 (Sigma);100 mmol/L dNTPs (Invitrogen);Cy3/Cy5 染料标记随机引物 (TriLink Biotechnologies);Klenow片段(NEB);杂交试剂盒40(NimbleGen);Nimble芯片杂交炉(NimbleGen);超声破碎仪(Bioruptor, Diagenode);热循环仪(Thermal cycler);分光光度计(Nanodrop ND-1000);芯片扫描仪(GenePix 4000B)。
1.2ES-BALB/c小鼠胚胎干细胞 中山大学附属眼科中心黄冰教授惠赠。
2方法
2.1ESCs体外诱导分化 首先制备羊膜取足月妊娠剖腹产的胎膜,钝性分离除去绒毛膜,用含庆大霉素的生理盐水反复冲洗,去除血污,剪切成大小约为3 mm×5 mm方形,浸泡于ESCs培养液中备用。取传代培养48 h的ES-BALB/c细胞,以0.125%胰蛋白酶-EDTA消化液消化,按5×108cells/L接种于双层6孔培养板的下层,然后将制备好的羊膜置于双层培养板的上层,用无LIF的ESCs培养液培养。每隔24 h将培养液换掉一半,培养3 d,倒置镜下观察细胞形态变化。
2.2DNA样本的制备 采用Qiagen DNA抽提试剂盒抽提DNA(按DNeasy Blood & Tissue Kit实验手册进行),收集细胞沉淀去上清加入180 μL ATL缓冲液,加入20 μL蛋白酶K涡旋振荡均匀,56 ℃孵育直至细胞充分裂解;加入2 μL RNA酶A涡旋振荡均匀,室温孵育2 min;涡旋15 s,加入AL缓冲液到样本中,涡旋混匀,而后加入200 μL 乙醇(96%~100%),再次混匀;过柱6 000×g离心1 min收集;转移到新的2 mL收集管,加入500 μL的AW1缓冲液,6 000×g离心1 min 收集;500 μL的AW2缓冲液处理,20 000×g离心3 min 收集;将DNA纯化柱转移至干净的1.5 mL的离心管,加入200 μL的AE缓冲液室温孵育1 min,6 000×g离心1 min洗脱收集纯化后的DNA。对DNA浓度和纯度进行分光光度检测。
2.3启动子区差异性甲基化芯片分析
2.3.1超声打断基因组 每份样本11 μg DNA溶入300 μL的缓冲液进行超声打断;超声仪调至L档(低速档);将DNA试管周围的容器装满冷水,不断补充的0.5 cm大小的碎冰降温;超声10个循环,每个循环设置30开,30停;取5 μL DNA琼脂糖凝胶电泳检测超声效果。
2.3.2甲基化DNA免疫共沉淀 MeDIP样本和拟免疫共沉淀样本(mock immunoprecipitation,mock IP)(各5 μg基因组DNA)用IP缓冲液调到500 μL,本底对照样本(input)用IP缓冲液调到200 μL;将DNA在250 r/min转速下,94 ℃孵育10 min;然后在4 ℃下迅速冷却并做一个迅速的旋转;加入5 μg抗5-甲基胞嘧啶抗体于MeDIP样本中,5 μg鼠IgG抗体于拟免疫共沉淀样本中,本底对照样本不加任何试剂;在4 ℃旋转震动孵育过夜;加入50 μL偶联抗鼠IgG抗体的磁珠混匀,在4 ℃反复涡旋2 h;孵育后,在4 ℃用磁珠分离试剂富集磁珠2 min;去上清和废弃液;采用3种冲洗液按顺序冲洗磁珠。采用1 mmol/L Tris-HCl(pH 8.0)、0.5 mmol/L EDTA(pH 8.0)和10% 十二烷基磺酸钠(sodium dodecyl sulfate, SDS)配制洗脱液,向MeDIP和mock IP管各加入200 μL的洗脱液;65 ℃洗脱15 min;分离的磁珠转移到另外的管子里;酚氯仿法提取MeDIP-DNA和实验参照DNA;用Nanodrop ND-1000测定其质量和浓度。
2.3.3MeDIP与input DNA片段线性扩增
① 文库制备 MeDIP和input样本各10 ng,用水调至各10 μL总量;在MeDIP和input样本中再各加入2 μL的文库制备缓冲液,然后贴壁转移至0.2 mL的PCR反应管中;各加入1 μL文库稳定液、混匀,快速旋转、转移热循环仪95 ℃ 2 min;立刻在冰上冷却,再次快速旋转;再加1 μL文库制备酶,混匀;热循环仪孵育,反应设置:16 ℃ 20 s,24 ℃ 20 s,37 ℃ 20 s,75 ℃ 5 s,4 ℃保温;快速旋转,-20 ℃保温3 d。
②扩增及纯化片段 按Sigma全基因组扩增试剂盒的操作流程来进行扩增,配好60 μL的反应混合物加入到每份样本中,涡旋混匀。按以下热反应流程来孵育扩增:95 ℃预变性3 min,1个循环;94 ℃变性15 s,65 ℃退火、延伸20 s,设置14个循环;4 ℃保温。最后采用Qiagen的QIA快速PCR纯化试剂盒,对扩增的DNA进行纯化回收。
2.3.4荧光标记 按照罗氏芯片标记手册步骤操作,用1 OD/42 μL 的9 mer随机探针缓冲液来稀释Cy3(绿色荧光)和Cy5(红色荧光)标记的9 mer长的探针,将稀释液加入0.2 mL薄壁的PCR管中,等分每管稀释液到40 μL,-20 ℃避光保存。将MeDIP样本和Input样本分别放入到PCR管中。98 ℃ 10 min热变性,快速转移冰上10 min,加入20 μL dNTP/Klenow 混合物到各管中,充分吸打10次,并将底部振荡防止DNA沉淀到管底。避光37 ℃孵育2 h。之后用0.5 mol/L EDTA终止反应,每个管中加入5 mol/L NaCl ,简单的涡旋振荡后转移至含有异丙醇的1.5 mL管中,避光室温孵育10 min, 12 000×g离心10 min去上清,用500 μL的80%冰乙醇悬浮沉淀物,12 000×g离心10 min去上清。在真空离心蒸发浓缩器中,地热避光干燥5 min。在打开前简单地振荡环氧树脂管,之后用25 μL的纯化水对干燥颗粒进行涡旋,振荡水化处理。采用 Nanodrop ND-1000检测标记的DNA,并用6 μg标记的MeDIP/Input DNA进行芯片杂交反应。
2.3.5芯片杂交 设置杂交系统恒温到42 ℃,将6 μg的MeDIP样本和6 μg的Input样本悬浮与5 μL水中。加入合适的NimbleGen杂交液到每个管中,涡旋振荡15 s,转移到收集管中,95 ℃避光孵育5 min。涡旋振荡后将样本管放置于预热到42 ℃的杂交系统中。将NimbleGen Chip 混合液迅速放入精密混合物比对仪(Precision Mixer Alignment Tool, PMAT)中,并将芯片放入PMAT中。使用Gilson Microman M100 移液器将15 μL样本慢慢地加入到芯片槽中。MeDIP样本和Input样本在42 ℃芯片杂交反应16~20 h。杂交完成后,用洗脱液进行洗脱。
2.3.6图像采集和数据分析 芯片结果由Axon的GenePix 4000B双通道扫描仪进行扫描读取,获得原始信号值。使用GenePix Pro 6.0软件分析芯片结果原始信号值进行标准化等数据分析。将芯片读取后的数据采用NimbleScan 2.5软件进行分析,比较诱导的ELCs组与ESCs组DNA启动子区的甲基化差异。基因启动子甲基化情况的判断通过对启动子区设计的探针的甲基化富集峰值来体现,NimbleScan检测出的峰值必须至少2个探针P值的-log10值大于等于2。在500 bp范围内的峰值合并在一起来判断甲基化与否。
2.3.7数据分析处理 这里我们采用Bioconductor packages Ringo, Limma, and MEDME软件对芯片结果的原始信号值进行中位数中心化、位数标准化和线性化处理,用于后期的数据分析。用以上分析软件产生的log2标准化值被应用于检测固定长度的染色质中所在探针的富集信号强度。每个探针有相关的P值分数-log10,并设定一个样本中某个启动子区域内甲基化与否的阈值。
2.4芯片分析结果的PCR验证 将基因组DNA 超声打断成400~500 bp片段加热变性,并将变性后的单链DNA样品分为2份。其中一份单链DNA样品加入抗5-甲基胞嘧啶抗体。使用亲和层析分离共沉淀样品中的甲基化DNA片段的抗体复合物,样品中其余的非甲基化DNA片段被洗脱。纯化得到甲基化DNA 片段(MeDIP DNA)。另一份作为总对照样本DNA(total input DNA)样品。Real-time PCR检测。Total input DNA 样品作为模板,用待测基因特异性的引物进行real-time PCR,得到标准曲线的循环阈值(cycle threshold,Ct),即Ct(input);MeDNA-IP DNA样品作为模板进行检测,得到Ct (MeDNA-IP)。待测基因MeDIP的效率(MeDNA-IP/total input,%)= 2^[Ct(input)-Ct (MeDNA-IP)]× 100%。
结 果
1小鼠胚胎干细胞的培养及定向诱导鉴定
ESCs在分化前呈现巢式生长,细胞集落多呈圆或椭圆形,边界光滑,细胞排列紧密,无明显细胞边界,见图1A;诱导3 d 后,细胞形态出现明显变化,细胞大而扁平,呈多边形,排列紧密,细胞边界清晰,呈典型单层上皮样改变,见图1B。
2样本DNA质量检测
将ESCs组和ELCs组提取的基因组总DNA用ND-1000检测,A260/A280值均大于1.8,DNA纯度高,可用于后续实验。浓度为70.27 mg/L和103.76 mg/L,每管样本共100 μL的量,样本起始量均超过5 μg,足够实验所需。琼脂糖凝胶电泳见图2A, 总DNA条带清晰,DNA完整度好,未有降解,可用于后续实验。
上芯片的DNA样本需要进行超声片段化处理,用Nanodrop ND-1000检测,分析片段化的DNA大小的分布区域,通常标准的分布范围应该在200~1 000 bp。本次实验中,片段化的DNA基本符合上芯片的范围标准,见图2B。

Figure 1. Morphological changes of ESCs before and after differentiation (×200). A: undifferentiated ESCs; B: single layer of ELCs differentiated from ESCs.
图1ESCs分化前后的形态学变化

Figure 2. DNA quality tested by agarose gel electrophoresis. A: DNA integrity test; B: the size of DNA fragmentated by ultrasound. M: DNA marker; Lane 1: ESCs; Lane 2: ELCs.
图2样本DNA质量检测
3甲基化芯片及数据差异分析

Figure 3. Methylation chip images.A, B, C: ESCs; D, E, F: ELCs.
图3甲基化芯片扫描图
采用NimbleGen 1×2.1M小鼠高密度甲基化芯片进行杂交,样本采用Cy3绿色荧光,Cy5红色荧光双色通道标记,见图3,Cy3标记input对照组份,Cy5标记免疫共沉淀组份。从芯片扫描图上可见每张芯片信号均匀,清晰没有边缘化效应、刮伤等影响信号值,阳性定位点和阴性对照区域结果明显。利用小鼠高密度甲基化芯片对ESCs及诱导后的ELCs进行检测发现,在总共22 425个启动子中, 有17 500个启动子区发生甲基化,其中未分化前ESCs组检测有15 501个启动子发生甲基化事件,占总启动子的69.1%,诱导分化后ELCs组检测到15 973个启动子发生甲基化事件,占总启动子的71.2%;应用软件GenePix Pro 6.0,对结果进行差异分析,在差异甲基化谱中显示ESCs与诱导后的ELCs多达3 435个启动子发生甲基化差异变化,占总启动子的15.3%,富含CpG岛的启动子区(high CpG-containing promoter,HCP)有894个发生差异甲基化的启动子,含有CpG岛中等程度的启动子区(intermediate CpG-containing promoter,ICP)有974个发生差异甲基化的启动子,CpG岛含量微少的启动子区(low CpG-containing promoter,LCP)有1 567个发生差异甲基化的启动子。在HCP中,ESCs组基因启动子有357个发生了甲基化而ELCs中不存在甲基化,ELCs组中有537个基因启动子发生甲基化而ESCs组中没有检测到甲基化存在;在ICP中,ESCs组基因启动子有412个发生了甲基化而ELCs中不存在甲基化,ELCs组中有562个基因启动子发生甲基化而ESCs组中没有检测到甲基化存在;在LCP中,ESCs组基因启动子有714个发生了甲基化而ELCs中不存在甲基化,ELCs组中有853个基因启动子发生甲基化而ESCs组中没有检测到甲基化存在。
4基因启动子甲基化差异的生物信息学分析
通过GeneSpring 11.0软件对小鼠的高密度甲基化芯片中发生启动子甲基化差异的基因进行基因本体(gene ontology,GO)分析和信号通路(Pathway)分析,探讨这种表观遗传上的差异现象在生物学上的意义。甲基化基因的GO分析按照检验方法为Fisher’s exact test、P<0.01筛选,分析结果显示在GO三大类中,即生物学过程(biological process,BP)、分子功能(molecular function,MF)及细胞组分(cellular component,CC)。其中3 435个启动子甲基化差异的基因在BP中含有2 994个,在MF中含有3 032个,在CC中含有3 080个。按照可信值由高到底排列各挑选前10位的注释情况见表1。甲基化基因的信号通路(Pathway)分析按照检验方法为Fisher’s exact test、P<0.05筛选,在Pathway分析结果中显示共有1 102个差异甲基化的基因,并挑选可信值高的前10位进行分析,见表2。应用软件GenePix Pro 6.0,对在ESCs甲基化高ELCs中没有检测到的前10个基因,及在ELCs中甲基化高而在ESCs中没有检测到的前10个基因进行分析和注释,见表3、4。
5MeDIP-PCR实验对结果进行验证分析
表1小鼠ESCs诱导分化成ELCs差异甲基化基因的GO分析结果
Table 1. The GO result of the differentially methylated genes of the mouse ESCs induced to be ELCs

GOIDTermCountPopHitsBiologicalprocess GO:0009987Cellularprocess164011097 GO:0008152Metabolicprocess10597156 GO:0044238Primarymetabolicprocess9146294 GO:0065007Biologicalregulation9056258 GO:0044237Cellularmetabolicprocess8516046 GO:0050789Regulationofbiologicalprocess8315922 GO:0050794Regulationofcellularprocess7875624 GO:0043170Macromoleculemetabolicprocess7335283 GO:0023052Signaling5553836 GO:0032501Multicellularorganismalprocess6194435Molecularfunction GO:0005488Binding157910643 GO:0003824Catalyticactivity7814895 GO:0043167Ionbinding5853760 GO:0043169Cationbinding5743712 GO:0046872Metalionbinding5683683 GO:0004871Signaltransduceractivity4532789 GO:0060089Moleculartransduceractivity4532789 GO:0004872Receptoractivity4232559 GO:0005515Proteinbinding7475293 GO:0004888Transmembranereceptoractivity3161956Cellularcomponent GO:0005623Cell219914268 GO:0044464Cellpart219814267 GO:0005622Intracellular14429624 GO:0044424Intracellularpart14119377 GO:0016020Membrane11557341 GO:0031224Intrinsictomembrane9305710 GO:0043229Intracellularorganelle11957973 GO:0043226Organelle11957977 GO:0044425Membranepart9896244 GO:0016021Integraltomembrane9095591
GO ID:the ID of gene ontology term;term:the name of gene ontology term;count:the number of DE genes associated with the listed GO ID;Pop Hits:the number of background population genes associated with the listed GO ID.

表2 小鼠ESCs诱导分化成ELCs差异甲基化基因在Pathway中分析结果
Definition: the definition of the Pathway ID;selectioncounts:the count of the DE gene entities directly associated with the listed Pathway ID;count:the count of the chosen background population gene entities associated with the listed Pathway ID;enrichment score:the enrichment score value of the Pathway ID, which equals to “-log10(Pvalue)”。

表3 ESCs高甲基化水平的基因
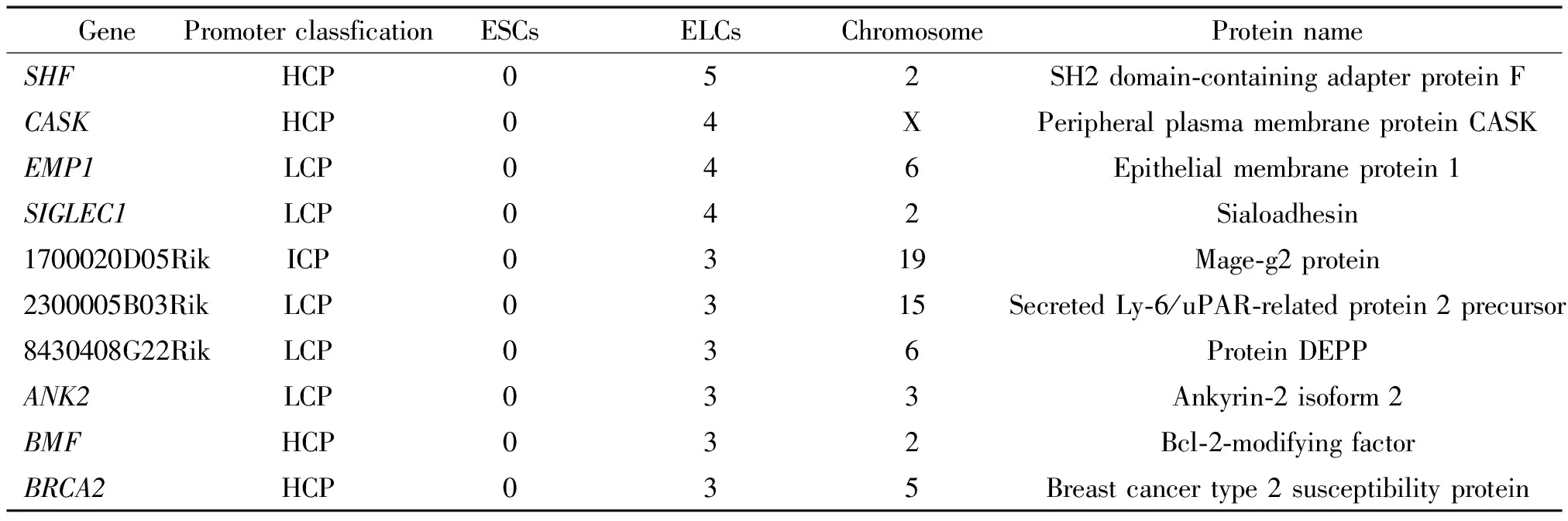
表4 ELCs高甲基化水平的基因
结合甲基化芯片的差异甲基化基因与GO分类分析挑选出可信值高,并可能与小鼠ESCs分化为ELCs相关的基因做进一步的芯片验证实验,以证实芯片的结果的可靠性。根据GO和Pathway分析结果,我们挑选参与细胞黏附和细胞周期的5个基因做验证,这5个基因分别为:细胞周期蛋白依赖性激酶7(cyclin-dependent kinase 7,CDK7)、钙黏蛋白17 (cadherin 17,CDH17)、原钙黏蛋白12 (protocadherin 12,PCDH12)、内皮细胞特异性黏附分子(endothelial cell-specific adhesion molecule,ESAM)和接触蛋白1 (contactin 1,CNTN1)。验证显示挑选的差异甲基化基因的启动子甲基化差异结果和RT-PCR结果趋势基本一致,见表5,表明芯片数据真实可靠,可为后续的功能研究提供依据。

表5 差异甲基化基因启动子芯片的甲基化情况与MeDIP-PCR对应情况
Number 0 or 1:a fragment of the gene promoter methylation number;IP/neg(MeDIP-PCR):the ratio of immunoprecipitation to negative control PCR result.
讨 论
表观遗传在正常的发育和细胞生长过程中起作用,并且在基因、环境与疾病的关系之间也可能起到至关重要的作用[3-4]。细胞分化是一个从全能到多能,最后到专能,外延不断缩小,内函不断扩大的过程。在这个过程中,基因组并未发生明显的序列变化,而是通过表观遗传有序的时空变化实现的,即基因启动子区甲基化水平的变化。基因启动子区的DNA甲基化的变化有可能直接调控了基因的表达,进而控制细胞功能和表型,因此,基因启动子区的甲基化有序的时空变化是个体发育过程中非常重要的调控环节,其中隐藏着大量与生命发生有关的玄机。从对基因启动子区的甲基化谱入手,可望阐明细胞定向分化的关键调控机制,为细胞分化、个体发育研究进行理论和方法学探索。
DNA甲基化芯片技术作为基因组学研究的工具,经过多年的发展和检验,已是一种成熟,可靠的常用技术[5]。这种技术可以将某一空间和时间的样本基因启动子甲基化情况整体地展现出来。这里我们研究采用的是NimbleGen公司的高密度芯片1×2.1M的芯片,甲基化启动子区的设计源于NCBI MM9权威数据库,每张芯片含有2.1兆根探针含有22 425个小鼠基因启动子信息,探针覆盖基因启动子转录起始位点上游8.2 kb,转录起始位点下游3 kb,还包含CpG岛及miRNA启动子甲基化信息。探针覆盖区域为tilling设计,每个基因的长寡核苷酸探针的长度为60 mer,可以提供更高的信噪比、灵敏度、专一性和辨别能力。使用该芯片检测出小鼠ESCs及诱导后的ELCs的差异甲基化,得到关于影响小鼠ESCs诱导为ELCs的大量宝贵的基础信息,为进一步研究其中的发生机制提供了依据。
跟据CpG岛的密集程度,基因启动子分为3类: HCP、ICP和LCP。CpG通常被认为是甲基化高发区段,在众多疾病研究领域中起着关键的作用[6-7],而在细胞分化过程中,我们通过数据统计可以看到ICP的甲基化占多数。
我们这里应用软件GenePix Pro 6.0,对甲基化芯片中基因启动子区进行了一种表达调控极端模式的筛选,即ESCs中存在基因启动子甲基化事件但ELCs中相应的基因却没有检测到甲基化信号的基因作为备选,进行后期研究,同样的ELCs中存在基因启动子甲基化事件,但是ESCs中相应的基因没有信号值的基因也作为备选后期研究的基因。这样总共筛选到了3 435个基因,有些基因在启动子区有多条探针信息显示发生了甲基化,也有微量发生甲基化。将这些基因按照GO和Pathway进行归类[8],并进行讨论其可能在ESCs诱导分化为ELCs的机理机制。
本文的GO分析是对发生了甲基化差异的基因的生物学功能进行注释。之前在全基因表达的研究成果中对GO分析做了非常详尽的介绍[9]。同样选择基因分类错误率(false discovery rate of the GOID, FDR)最小、且P值也是最小的前10位进行一个探讨,在生物学过程中,主要是以一系列的细胞基础代谢及生物分子调控为主,提示在小鼠ESCs定向分化为ELCs的过程正如其在胚胎发育的程中的表现情况类似,开启或关闭细胞中代谢过程的基因表达,调控与成熟分化后细胞所行使生物学功能相关的蛋白组分合成与降解,由量变上升为质变,改变生物学表型。在生物学过程中,某些基因收到相关DNA甲基化表观遗传的调控,使得表达上升或者下降,引导合成分化后细胞的功能相关蛋白,抑制影响分化合成的功能性蛋白的合成。其中我们会选取具有代表性意义的基因,进行下一步的功能分析,看此基因的表达情况能否影响其表型的发生。在分子功能的GO条目中,我们可以看出小鼠ESCs在诱导分化为ELCs的过程中DNA甲基化基因启动子调控是处于一种高度活跃的状态,激活或抑制某些跟受体激活、蛋白结合等调控相关的因子基因的表达,这些基因的表达调控可能与组织的特化相关。在细胞成分GO分类中,我们可以看出DNA甲基化的调控对细胞膜相关的基因占主导地位。本身表皮是属于生物体的一种不同于其它细胞器样的膜基质的细胞成分,故细胞膜上的基因表达明显受到DNA甲基化的表观调控。这一系列的DNA甲基化的变化对于ESCs分化为ELCs均可能是必要的。
ESCs分化为ELCs,主要是研究表皮方面的分化,在本研究中,我们着重对跟细胞黏附相关的基因的启动子区的DNA甲基化的情况做了筛选和进一步验证,从细胞黏附相关的基因中我们发现钙黏蛋白基因CDH17的DNA甲基化情况在ESCs分化为ELCs过程中是升高的,定量验证实验也证实了这个趋势。CDH17的主要功能是需要钙离子依赖活性、转运各种寡肽在膜上的一个功能团,它起源于脊椎动物发育早期的染色体复制[10]。CDH17基因的启动子属于LCP,是一种低CpG岛含量的启动子区,用芯片检测到一个甲基化区域内发生了甲基化事件,定量检测的趋势很明显,在本研究中定向诱导分化为ELCs的发育研究的结果是符合生长发育的基本规律,CDH17基因启动子区的甲基化水平升高,这很有可能在定向分化的过程中还是起到某种重要的表观调控作用,跟之前的基因谱检测结果也相一致,相应的基因也发生了下调[8];PCDH12也是一种钙依赖性跨膜蛋白,有研究报道它确实会影响到组织形态的形成,血管生成,细胞-基质的黏附和迁移,免疫反应和染色质重塑[11]。在本研究中PCDH12的启动子区的甲基化也属于LCP,芯片检测到有存在甲基化的可能,但在相应的位置上用定量检测,未发现有甲基化的片段存在,这可能说明PCDH12基因的DNA甲基化表观不是直接调控PCDH12的基因表达发送差异变化的主要因素,研究中检测到PCDH12基因表达差异很有可能与其它的表观调控模式有关,才会导致PCDH12的表达差异,例如组蛋白的修饰,有可能羟甲基化这种表观遗传修饰也会导致PCDH12的表达差异,这些都有待我们进一步的研究和证实。CNTN1 参与DNA甲基化、细胞黏附、Notch 信号通路生物学过程,也是一种膜上蛋白,具有与DNA结合、与蛋白结合、N端转移酶活性、GPI锚定的分子功能。DNA甲基化在定向分化的过程中起着非常重要的作用,某些基因的甲基化和去甲基化,直接决定分化后的生物表型和功能,在生长发育和疾病发生中有指导作用[12-13],CNTN1基因的启动子区属于ICP,是一种中度甲基化启动子区,在芯片检测中我们发现它从ESCs分化为ELCs的过程是甲基化程度升高的,并且在定量检测中我们也证实这一明显的趋势。ICP的基因启动子的定量检测数据中也发现,确实是比LCP要更高些。这个甲基化的趋势和之前基因表达的情况对比也是负相关的,CNTN1的表达也是随着分化而下降的[8]。这对今后更深入地研究胚胎干细胞分化为ELCs的发生机制给予提示和奠定基础。在细胞黏附过程中,我们还发现了一个ICP的ESAM基因启动子,定量检测发现它的甲基化变化差异特别大,我们对应之前的研究发现,它在表达谱中的表达也是明显下调的。ESAM基因位于小鼠第9号染色体,翻译的蛋白属于钙依赖的细胞黏附因子,主要在膜基质中起一定作用,具有受体激活和蛋白结合一类的分子功能[14],这提示我们这个基因在ESCs分化为ELCs的过程中可能存在比较重要的作用。
在信号通路的分析结果中,按通路中的基因启动子甲基化的富集分值大小,选择富集程度高的前10个同路的情况进行分析,发现其中在诱导成为ELCs过程中有一个很典型的信号通路,PPAR信号通路,这个信号通路研究发现与哺乳动物的生殖有很大关系,也符合我们的研究标准。并且在通路分析中,我们同样看到跟合成和代谢相关的通路也发生了比较大的变化。这里我们着重挑选一个我们感兴趣基因进行了讨论,CDK7基因启动子属于HCP,即它的启动子区富含CpG岛。CDK7参与转录过程、DNA依赖的转录调控、蛋白氨基酸的磷酸化、细胞周期和细胞分裂过程,是核内的一种分子结构,具有核苷酸结合、蛋白激酶活性、蛋白丝氨酸苏氨酸激酶活性、细胞周期依赖的蛋白激酶活性、ATP结合、转移酶活性等功能。本实验中,此基因的启动子DNA甲基化是一个去甲基化的过程,则按照调控负相关的原理,它对应的基因表达上也会有所升高,前期的研究也证实了这一点。CDK7基因无任是芯片检测结果还是定量检测中都是上升的趋势,提示CDK7在ESCs定向分化为ELCs后在维持ELCs细胞形态和功能上有一定作用。
综合以上分析,在ESCs体外定向分化为ELCs的过程中,基因的启动子区的DNA甲基化发生了巨大的变化,包括众多信号分子、酶和细胞结构蛋白基因的启动子区的甲基化变化,提示细胞分化不是一简单的分子事件,而是由一系列分子通过网络关联事件共同协作完成。该定向分化的关键分子调控机制在DNA甲基化水平已得到初步数据的展示,尚需要进一步大量功能实验研究阐明。我们的后续实验将从这些筛选出的结果中,挑选感兴趣的基因,深入功能研究讨探其在分化过程中表观调控所起作用及作用机制。
[1] 张仁礼,李海标. 人羊膜体外诱导胚胎干细胞分化为表皮样干细胞机理的初步研究[J]. 中国病理生理杂志, 2004, 20(7): 1163-1166.
[2] 张仁礼, 李海标. ES细胞源的表皮样干细胞分化潜能的初步研究[J]. 解剖学报, 2004, 35(1): 69-73.
[3] Weber M, Hellmann I, Stadler MB, et al. Distribution, silencing potential and evolutionary impact of promoter DNA methylation in the human genome[J]. Nat Genet, 2007, 39(4):457-466.
[4] Weber M, Davies JJ, Wittig D, et al. Chromosome-wide and promoter-specific analyses identify sites of differential DNA methylation in normal and transformed human cells[J]. Nat Genet, 2005, 37(8):853-862.
[5] 齐素文,涂植光,戴 勇,等.IgA肾病组蛋白H3K4三甲基化和DNA甲基化基因修饰的相关性[J].中国病理生理杂志,2012,28(2):314-319.
[6] Luo Z, Shi H, Zhang H, et al. Plasmid DNA containing multiple CpG motifs triggers a strong immune response to hepatitis B surface antigen when combined with incomplete Freund's adjuvant but not aluminum hydroxide[J]. Mol Med Report, 2012 , 6(6):1309-1314.
[7] Lu ZM, Zhou J, Wang X, et al. Nucleosomes correlate withinvivoprogression pattern ofdenovomethylation of p16 CpG islands in human gastric carcinogenesis[J]. PLoS One, 2012, 7(4): e35928.
[8] 张仁礼,刘彩霞,孟锦绣. 胚胎干细胞分化为表皮样细胞的基因表达谱差异研究[J].中山医科大学学报, 2012,33(2): 149-155.
[9] Robertson KD, Keyomarsi K, Gonzales FA, et al. Differential mRNA expression of the human DNA methyltransferases (DNMTs) 1, 3a and 3b during the G0/G1to S phase transition in normal and tumor cells[J]. Nucleic Acids Res, 2000, 28(10): 2108-2113.
[10] Wendeler MW, Jung R, Himmelbauer H, et al. Unique gene structure and paralogy define the 7D-cadherin family[J]. Cell Mol Life Sci,2006, 63(13): 1564-1573.
[11] Rampon C, Bouillot S, Climescu-Haulica A, et al. Protocadherin 12 deficiency alters morphogenesis and transcriptional profile of the placenta[J]. Physiol Genomics, 2008, 34(2):193-204.
[12] Davisson MT, Bronson RT, Tadenev AL, et al. A spontaneous mutation in contactin 1 in the mouse[J]. PLoS One, 2011,6(12): e29538.
[13] Liu P, Zhou J, Zhu H, et al. VEGF-C promotes the development of esophageal cancer via regulating CNTN-1 expression[J]. Cytokine, 2011, 55(1): 8-17.
[14] Sudo T, Yokota T, Oritani K, et al. The endothelial antigen ESAM monitors hematopoietic stem cell status between quiescence and self-renewal[J]. J Immunol, 2012, 189(1): 200-210.
Genome-widescreeningofpromotermethylationinembryonicstemcellsandembryonicstemcell-derivedepidermal-likecells
ZHANG Ren-li1, LIU Cai-xia1, MENG Jin-xiu2, ZHANG Li-li1, HAN Dong1, CAI Jia-jie1, WEN An-min1
(1ReproductiveMedicineCenter,2MedicalResearchCenter,GuangdongAcademyofMedicalScience/GuangdongGeneralHospital,Guangzhou510080,China.E-mail:renlizhangm@yahoo.com.cn)
AIM: To investigate the differential methylation of the promoters of the genes involved in the process of differentiation from embryonic stem cells (ESCs) into epidermal-like cells (ELCs).METHODSMouse ESCs were induced to differentiate into ELCsinvitroby using human amnion. Whole-genome DNA differential methylation between undifferentiated ESCs and ELCs was analyzed by DNA methylation chip. Immunoprecipitation of methylated DNA was performed using BiomagTMmagnetic beads coupled with mouse monoclonal antibody against 5-methylcytidine. The total input and immunoprecipitated DNA were labeled with Cy3- and Cy5-labeled random 9-mers, respectively, and hybridized to NimbleGen MM9 CpG promoter arrays (a 2.1M format array containing all known CpG islands annotated by UCSC and all well-characterized RefSeq promoter regions, from about -8 200 to +3 000 bp of the transcriptional start sites,totally covering 22 425 promoters). DNA methylation of promoter regions was analyzed to discover the potentially related genes.RESULTSAmong all the 17 500 DNA mathylation regions of the gene promoters in both ESCs and ELCs, 3 435 gene promoters showed to be differentially methylated, with 894 in high CpG-containing promoter,974 in intermediate CpG-containing promoter and 1 567 in low CpG-containing promoter.CONCLUSIONGreat changes of methylation in a large quantity of gene promoters occur during the differentiation of ESCs to ELCs, indicating that the differentiation is a complex process regulated by many genes, and how these genes work and co-work to regulate the differentiation procedure needs further investigation.
DNA methylation; Embryonic stem cells; Epidermal-like cells; Promoter; Immunoprecipitation of methylated DNA
R329.2+8
A
10.3969/j.issn.1000- 4718.2013.05.001
1000- 4718(2013)05- 0769- 09
2012- 11- 07
2013- 02- 05
国家自然科学基金资助项目(No. 30700327);广东省医学科学基金资助项目(No. WSTJJ201112062107021969052-10211);广东省自然科学基金资助项目(No. 9151009101000011)
△通讯作者 Tel: 020-83827812-60613; E-mail: renlizhangm@yahoo.com.cn
