栉孔扇贝EST-SNP标记开发及多态性分析*
2013-10-16李纪勤包振民胡晓丽
李纪勤,包振民,李 玲,胡晓丽
(中国海洋大学海洋生物遗传育种教育部重点实验室,山东 青岛266003)
栉孔扇贝(Chlamys farreri)是我国主要海水养殖种类之一,优良品种培育是产业健康可持续发展的关键。规模开发栉孔扇贝分子标记是发掘其种质资源,进行生产性状的遗传解析、建立分子育种技术和培育高产抗逆优良品种的基础工作之一。
在现有的分子标记技术中,单核苷酸多态性(Single Nucleotide Polymorphism,SNP)以其二态易于分型、遗传稳定性高、密度高、易于实现自动化检测分析等优点成为继扩增片段长度多态性(AFLP)、微卫星(SSR)之后新一代的分子标记,广泛应用于高密度遗传图谱的构建、基因定位、群体遗传学等研究[1]。表达序列标签(EST)数据库发掘是目前SNP筛选的重要途径。相比基因组来源的标记,EST-SNP均来自转录的基因。EST-SNP用于数量性状位点(QTL)研究可直接将目标性状定位到相关基因,有利于性状的遗传解析。
水产动物中EST-SNP标记开发已经有若干报道,如大西洋 鳕 鱼[2]、大 菱 鲆[3]、对 虾[4]、蟹[5]、太 平 洋 牡蛎[6]、太平洋鲍[7]、海湾扇贝[8]、虾夷扇贝[9]等。在栉孔扇贝中也开发了少量SNP标记[10-12],但数量远未达到高密度连锁图谱构建和QTL精细定位的要求。
目前已报导的SNP检测和标记开发技术有许多种[13],如基于构象改变的SSCP、DGGE、DHPLC等,基于杂交的TaqMan、microarray等,以及基于引物延伸的DNA测序如焦磷酸测序等。尽管方法众多,但分别存在开发通量低、操作繁琐、费用高、适用范围有限等问题,限制了SNP标记在非模式生物中的开发和利用。近年来,高分辨率熔解曲线(High resolution melting,HRM)被证明是一种通量大、效率高的SNP检测技术[14],其原理是基于不同等位形式对退火温度Tm的影响,通过熔解曲线的变化确定基因型。另外,可应用非标记探针进行HRM检测,从而大幅降低SNP标记开发成本[15]。本研究对栉孔扇贝转录组测序数据进行EST-SNP筛查,利用改进的HRM非标记探针技术进行SNP验证分型,开发了33个EST-SNP标记,可用于栉孔扇贝的遗传和育种研究。
1 材料与方法
1.1 模板DNA的制备
实验所用栉孔扇贝样品分别取自4个野生群体(长岛、荣成、青岛、日照),其中长岛、荣成、日照群体各12个个体,青岛群体48个个体。分别取其闭壳肌液氮速冻后转入-80℃冰箱中保存。利用酚-氯仿法提取冷冻样品的DNA[16],紫外分光光度计定量,-20℃保存备用。
1.2 EST-SNP的查找和引物、探针设计
本课题组之前对栉孔扇贝转录组进行了454测序,EST序列经过拼接后,利用QualitySNP程序对含有4条以上EST的拼接序列(contig)进行比对分析,查找候选SNP位点。选择2种等位形式均至少在2条EST序列中重复出现的单碱基突变作为候选SNP,进行后续验证分型。
针对每一候选位点设计2条引物,并对每个获得预期扩增产物的位点设计一条非标记非修饰探针[15]。利用在线程序Primer3V4.0进行引物的设计,引物长度至少19nt,Tm在59~61℃之间,预期PCR产物大小不超过150bp。预期扩增产物中只能包含一个候选SNP位点。探针设计采取如下标准:1)目标位点尽量位于探针序列的中间位置;2)Tm介于59~61℃,Tm可利用在线程序 Oligo Calculator V3.25(http://www.basic.northwestern.edu/biotools/oligocalc.htm)进行估计;3)探针长度20~35nt;4)探针设计完成后,在3′端添加2个错配碱基,进行3′端封闭,防止探针作为引物延伸;5)探针和引物序列不能有重叠。引物和探针由上海生工生物工程公司合成。
1.3 引物验证和位点多态性检测
以4个群体每个群体12个个体DNA等量混合作为模板DNA,进行引物的检测。实验采用10μL体系的不对称PCR(15ng模板DNA,0.1μmol/L正向引物,0.5μmol/L反向引物,1.5mmol/L MgCl2,2.0 mmol/L dNTP,1UrTaq DNA聚合酶,1XLCGreen Plus染料),95℃变性5min之后,进行60个循环的扩增(95℃40s,60℃40s,72℃40s),最后在72℃延伸5min。利用8%的聚丙烯酰胺凝胶电泳检测PCR产物。
根据电泳结果,选择产生大小符合预期、单一条带PCR产物的位点进行探针的检测并对4个群体的48个个体进行分型,PCR反应体系如前所述。扩增反应结束后,向每个反应添加相应的探针至终浓度达到2.0 μmol/L,95℃变性5min后25℃复性30s。HRM分析利用LightScanner仪器进行,以0.1℃/s的速率缓慢从40℃升温至95℃,以0.1℃的分辨率采集荧光信号,利用LightScanner软件进行荧光信号的分析处理。
1.4 遗传参数估计
选择在4个群体中检测为多态性的位点,对青岛群体48个个体进行分型,并进行相关遗传参数的统计。利用PopGen32计算低频等位基因(Minor allele)频率、观测杂合度(Observed heterozygosity,Ho)、期望杂合度(Expected heterozygosity,He)。利用在线软件 GENPOPE(http:∥genepop.curtin.edu.au/)进行 Hardy-Weinberg平衡(Hardy-Weinberg equilibrium,HWE)和连锁不平衡分析。
1.5 多态SNP功能注释
对验证为多态SNP,提取其所在的contig序列,利用NCBI的BLASTX程序进行功能注释。P<1×e-5作为同源序列判定的阈值标准。利用ORF finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)查找EST中的开放阅读框,确定SNP在遗传密码子的位置,与标准密码子比对确定该突变是否引起编码氨基酸的改变。
2 结果
2.1 栉孔扇贝EST序列中的SNP筛查
本课题组前期对栉孔扇贝转录组进行了454高通量测序,EST序列经过拼接后共获得38 944条contig。在其中18 780条含4条以上EST的contig中共检测到21 813个候选SNP位点,SNP平均频率为426.5 bp。SNP位点中,转换14 641个(A/G 7 408、C/T 7 233)、颠换7 172个(A/C 2 000、A/T 2 500、G/T 1 708、G/C 914)。其中,A/G 突变最 多,占 总 数的34.0%;G/C突变最少,占总数的4.2%。
2.2 候选SNP位点的分型
根据SNP位点侧翼序列的保守性和位点分布频率,选取90个位点设计引物。其中,66个位点产生符合预期大小的单一条带,24个位点(26.7%)扩增产物大于预期。对符合预期的66个位点设计合成探针,并利用4个群体共48个个体进行多态性检测。结果显示,其中18个位点(20.0%)无法准确分型,另有15个位点(16.7%)在检测的个体中无多态性,因此,能明显区分出2种不同基因型的位点为33个(36.7%)。图1所示为多态位点C15117S745_AG的HRM分型结果。33个多态位点的信息已提交到GenBank。
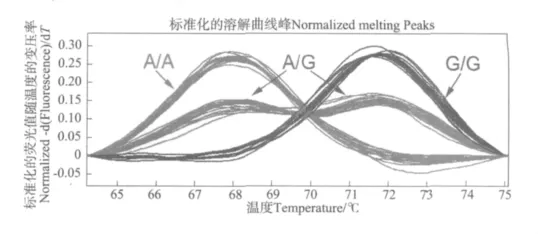
图1 SNP位点C15117S745_AG的非标记探针HRM分型检测结果Fig.1 Genotyping result of SNP C15117S745_AG using HRM assay with unlabeled probe
利用青岛野生群体对33个多态位点作进一步的验证分型。低频等位基因频率介于0.052 1~0.479 6,观测杂合度Ho和期望杂合度He的分布范围分别为0.062 5~0.744 7和0.099 8~0.504 6(见表1)。所有位点都处于连锁平衡状态,其中5个位点偏离HWE。
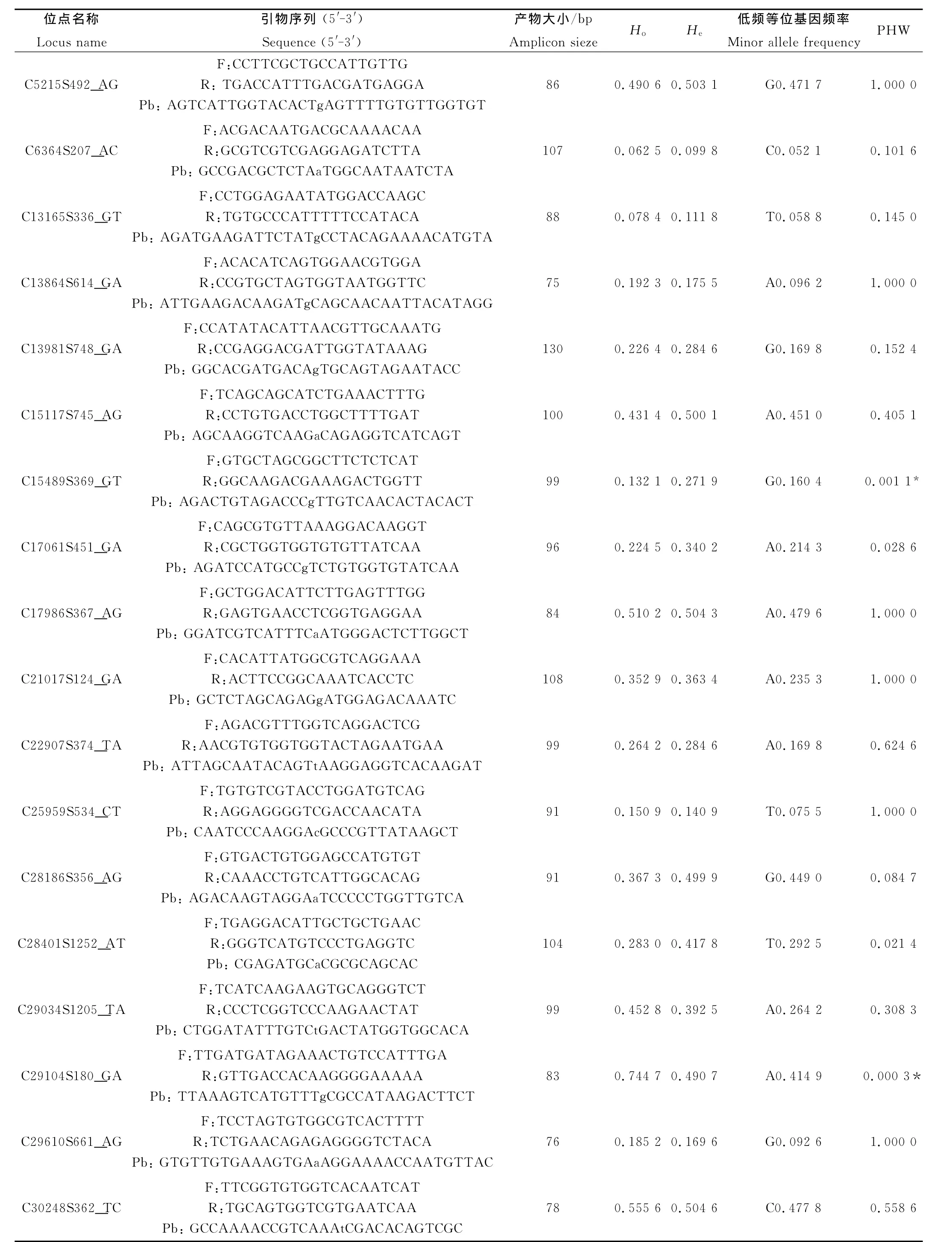
表1 栉孔扇贝33个多态SNP位点信息Table 1 Characterization of 33polymorphic SNP loci for Zhikong scallop(Chlamys farreri)
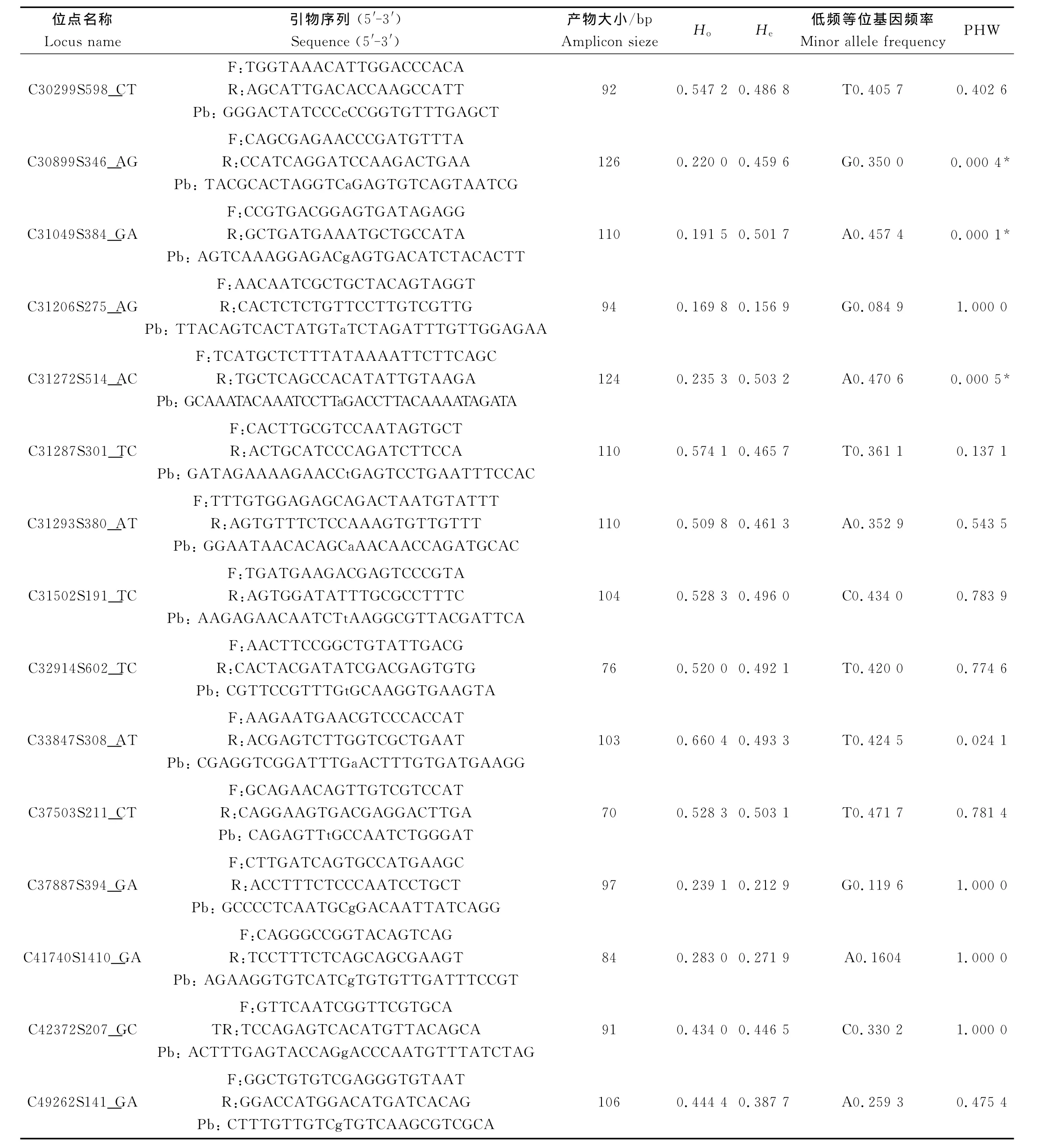
续表1
2.3 栉孔扇贝EST-contig的功能注释
利用NCBI的BLASTX程序,对33个多态SNP对应的contig序列进行了功能注释(见表2)。其中,6个SNP所在的contig没有找到其有效匹配;26个SNP对应可以注释的contig,涉及的基因包括转甲基酶(Putative methyltransferase NSUN5)、tRNA 特异腺苷脱氨酶(tRNA-specific adenosine deaminase)、ATP依 赖 的 RNA 解 旋 酶 (ATP-dependent RNA helicase DHX8)、跨膜蛋白(Transmembrane protein 186)等。
利用ORF finder推测出EST正确的开发阅读框,确定SNP在遗传密码子的位置,与标准密码子比对发现C29104S180_GA位于5′-UTR区;而 C21017S124_GA、C28186S356_AG、C29610S661_AG、C30899S346_AG、C32914S602_TC、C33847S308_AT、C49262S141_GA位于密码子的首位或第二位,C13864S614_GA位于密码子第三位,均造成了编码氨基酸的改变,因此为错义突变。位点C6364S207_AC位于起始密码子第一位,可能造成基因表达的沉默。剩余23个位点均位于密码子第三位,与标准密码子比对后确认为同义突变(见表2)。
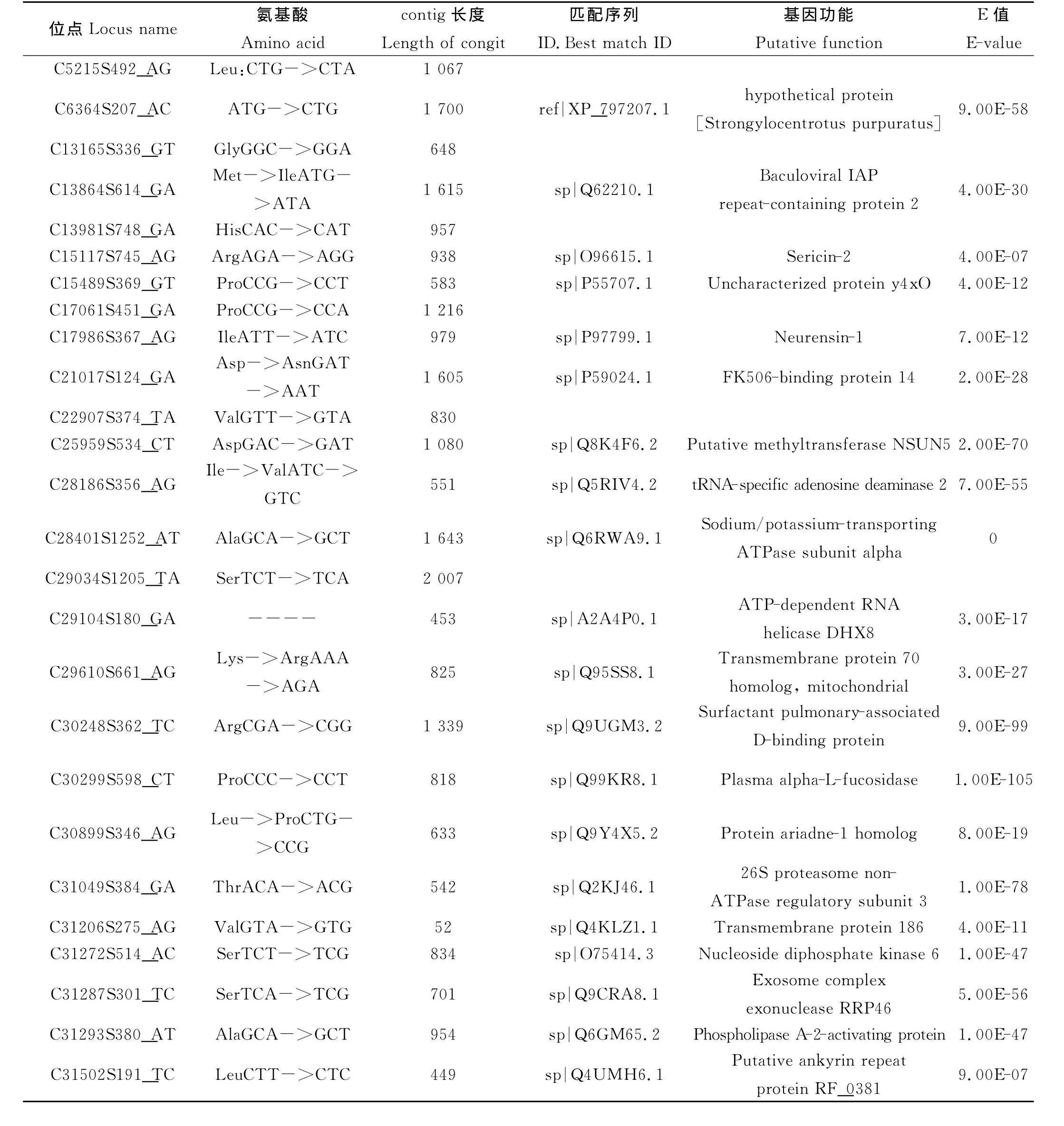
表2 栉孔扇贝33个多态SNP位点的功能注释Table 2 Functional annotation of 33polymorphic SNPs in Zhikong scallop(Chlamys farreri)
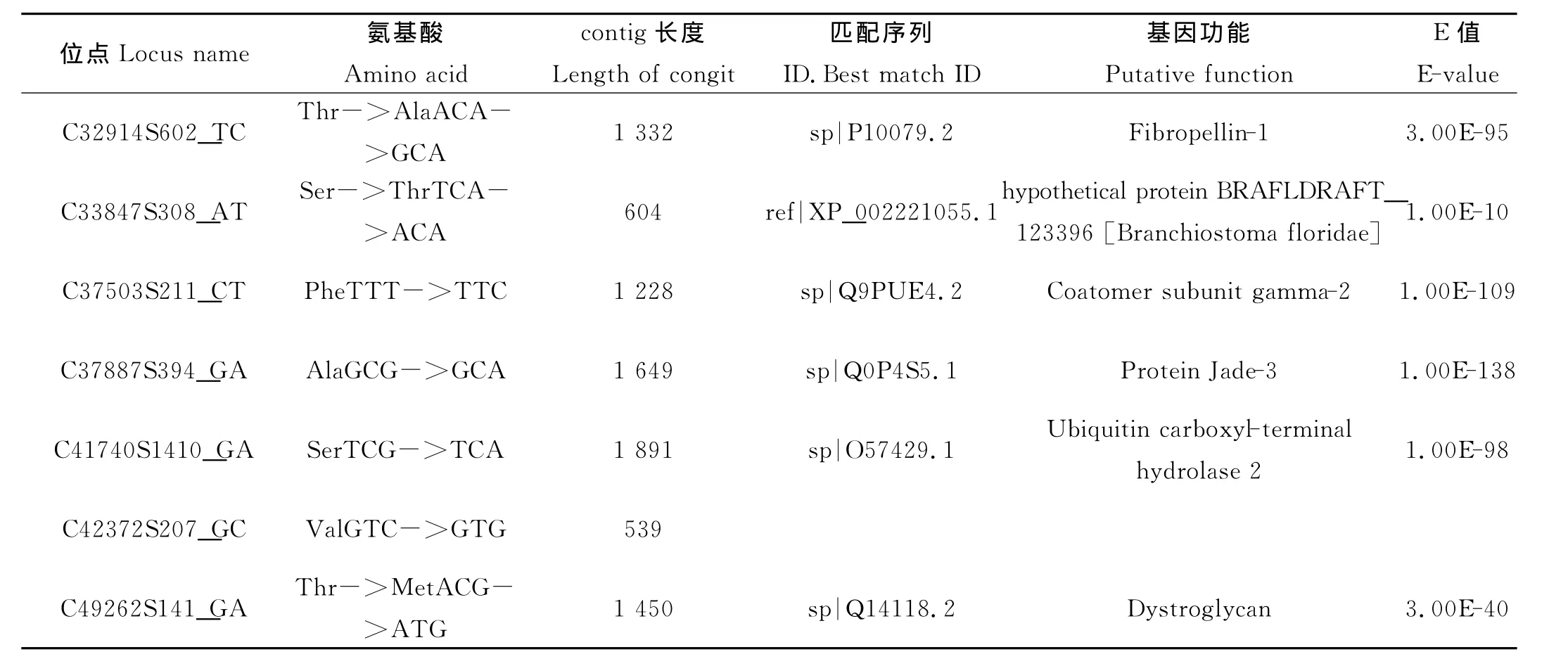
续表2
3 讨论
SNP是基因组中冗余度低而覆盖度最高的变异,在动植物性状的精细遗传解析及人类疾病和健康的遗传机制研究中具有广泛的应用前景。与模式生物相比,非模式生物的SNP研究,无论从SNP数量还是研究方法上,都相对滞后。栉孔扇贝作为我国重要经济海水养殖种类,就目前研究来看,其分子标记也以微卫星标记为主,只有少数SNP标记被报道[10-12]。本研究中,利用转录组测序得到栉孔扇贝大量EST序列,从中获得候选SNP位点21 813个,EST序列中平均426.5bp含有一个SNP,明显低于其他水产动物EST中的SNP位点平均间距(如鲍 100bp[3-4]、海湾扇贝118bp[5]、虾夷扇贝159.2bp[6])。除了不同物种间可能存在SNP密度差异外,另一个可能的原因是本研究采用了较为严谨的EST拼接条件(重叠区≥100bp,相似度≥95%)进行候选SNP位点的查找。
由于功能基因转录后,mRNA存在剪切编辑,因此通过生物信息技术筛查的候选SNP位点应在基因组中进行验证以保证其可靠性。本研究选取的90个候选EST-SNP中,24个位点(26.7%)扩增产物大于预期,推测是由于扩增产物跨越内含子造成的,部分位点经克隆测序确认了内含子的存在。为了降低扩增产物跨内含子的可能性,引物设计时应使预期扩增产物尽可能短。成功扩增的66个位点中,15个位点在4个群体中表现为单态,说明部分候选SNP位点可能存在假阳性;原因可能是由于EST序列通常较短,在聚类分析过程中错误地将同源性比较高的非等位基因聚为一簇[17],增加了假阳性的概率。Wang等[18]认为contig包含的EST数目和低频基因频率是影响EST-SNP开发成功率的2个最重要因素,contig包含的EST数目越多、低频等位基因频率越高,标记开发的成功率越高,因此,开发EST-SNP标记时应该尽量选择重叠群大和等位基因频率分布均匀的位点。另有18个位点没有得到准确的HRM分型结果,部分原因可能是单一位点的扩增产物中有2个或更多的SNP位点,或者探针存在非特异性结合。
对获得的33个SNP标记,在青岛野生群体中进行了验证和群体遗传多样性分析。所有位点均显示为多态,观测杂合度Ho和期望杂合度He的分布范围分别为0.062 5~0.744 7和0.099 8~0.504 6。对比显示,SNP标记揭示的群体多样性指数,如观测杂合度Ho和期望杂合度 He要低于 AFLP[19]和SSR[20]标记分析的结果,这是因为SNP标记为双等位形式,每个位点所携带的多态信息较少。本研究所使用的SNP来源于EST序列,可能受到更高的选择压力,因而在群体中保持较低的多态性水平。Hardy-Weinberg平衡检验结果显示,33个位点中有5个偏离Hardy-Weinberg平衡状态,表明青岛群体可能经历了遗传漂变、个体迁徙等过程。
从EST中开发SNP标记的1个显著优点,就是标记直接与功能基因相关,为将来的遗传性状功能解析和功能基因克隆提供了便利。本研究开发得到的33个SNP位点,其中有8个造成了编码氨基酸的改变。此类突变可能会进而造成蛋白质高级结构的变化,对表型产生影响。对比其它海洋贝类,如海湾扇贝、鲍、长牡蛎中,已报道的EST-SNP多产生同义突变,鲜有非同义突变,可能与物种基因组特征或者这些物种中已有的SNP位点数目较少有关。本研究还发现一个SNP位点位于5′-UTR区,有可能与相关基因的表达调控有关,另有1个SNP引起起始密码子的改变,可能会影响蛋白质的完整性和功能。对上述基因和位点的进一步研究正在进行中。
[1] Liu Z J,Cordes J F.DNA marker technologies and their applications in aquaculture genetics[J].Aquaculture,2004,238:1-37.
[2] Moen T,Hayes B,Nilsen F,et al.Identification and characterization of novel SNP markers in Atlantic cod:Evidence for directional selection[J].BMC Genet,2008,9:18.
[3] Vera M,Alvarez-Dios J A,Millan A,et al.Validation of single nucleotide polymorphism (SNP)markers from an immune expressed sequence tag(EST)turbot,Scophthalmus maximus,database[J].Aquaculture,2011,313:31-41.
[4] Yu M,Cheng Y,Fothschild M F.SNP analysis of Molting related genes in Penaeus monodon and Litopenaeus vannamei shrimp(Brief report)[J].Archiv Fur Tierzucht,2006,4:411-412.
[5] Ma H,Ma Q,Ma C,et al.Isolation and characterization of genederived single nucleotide polymorphism (SNP)markers in Scylla paramamosain [J].Biochem Syst Ecol,2011,39:419-424.
[6] Sauvage C,Bierne N,Lap gue S,et al.Single Nucleotide polymorphisms and their relationship to codon usage bias in the Pacific oyster Crassostrea gigas[J].Gene,2007,406(1-2):13-22.
[7] Qi H,Liu X,Zhang G.Characterization of 12single nucleotide polymorphisms(SNPs)in Paci?c abalone Haliotis discus hannai[J].Mol Ecol Resour,2008,8(5):974-976.
[8] Li R,Li Q,Kong L.Characterization of expressed sequence tagderived single-nucleotide polymorphisms in the bay scallop Argopecten irradians irradians [J].Fish Sci,2009,75(6):1389-1400.
[9] Liu W,Li H,Bao X,et al.The first set of EST-derived single nucleotide polymorphism markers for Japanese Scallop,Patinopecten yessonsis [J].J World Aquacult Soc,2011,42(3):456-461.
[10] Huan P,Zhang X,Li F,et al.Chromosomal localization and molecular marker development of the lipopolysaccharide and beta-1,3-glucan binding protein gene in the Zhikong scallop Chlamys farreri (Jones et Preston)(Pectinoida,Pectinidae)[J].Genet Mol Biol,2010,33(1):36-43.
[11] Wang X,Meng X,Song B,et al.SNPs in the myostatin gene of the mullusk Chalmys farreri:Association with growth traits[J].Comp Biochem Phys B,2010,155:327-330.
[12] Jiang G,Li J,Li L,et al.Development of 44gene-based SNP markers in Zhikong scallop,Chlamys farreri [J].Cons Genet Resour,2011,3(4):659-663.
[13] Garvin M R,Saitoh K,Gharrett A J.Application of single nucleotide polymorphisms to non-model species:a technical review[J].Mol Ecol Resour,2010,10(6):915-934.
[14] Li F,Niu B,Huang Y,et al.Application of high-resolution DNA melting for genotyping in Lepidopteran non-model species:Ostrinia furnacalis(Crambidae)[J].PloS ONE,2012 7(1):29664.
[15] Wang S,Zhang L,Meyer E,et al.Construction of a high-resolution genetic linkage map and comparative genome analysis for a reef-building coral Acropora millepora[J].Genome Biol,2009,10(11):126.
[16] Sambrook J,Fritsch E F,Maniatis T.Molecular Cloning:A Laboratory Manual[M].New York:Cold Spring Harbor Laboratory Press,1989.
[17] 王绍宗,李莉,亓海刚,等.长牡蛎(Crassostrea gigas)17个EST-SNP标记的开发[J].海洋与湖沼,2010,41(2):274-281.
[18] Wang S,Sha Z,Sonstegard T,et al.Quality assessment parameters for EST-derived SNPs from catfish [J].BMC Genomics,2008,9:450.
[19] 赵翠.栉孔扇贝与西施舌的群体遗传学研究[D].青岛:中国海洋大学,2008.
[20] 战爱斌.栉孔扇贝(Chlamys farreri)微卫星标记的筛选及应用[D].青岛:中国海洋大学,2007.
