大蒜果聚糖水解酶全长cDNA的克隆
2013-09-04何林乾黄雪松
何林乾,黄雪松
(暨南大学理工学院食品系,广东广州510000)
果聚糖(fructan)广泛存在于植物中,是由蔗糖与一个或多个果糖分子聚合而成的多聚体。研究表明,果聚糖是高等植物中一类重要的贮存物质或渗透调节物质,可以提高植物的抗逆性。目前已经发现的高等植物中的果聚糖的类型包括:菊糖型果聚糖,由β-2,1糖苷键连接;梯牧草型果聚糖,由β-2,6 糖苷键连接[1-2];混合型果聚糖,是前两种果聚糖的混合体,含分支。根据经典模型[3],果聚糖水解酶(FEH)主要负责果聚糖水解。大蒜(Allium sativum L.)中含有多种形式的果聚糖,这种果聚糖形式的多样性预示着大蒜中存在果聚糖的合成和分解两种相反的生物代谢过程[4-5]。1-果聚糖水解酶(1-FEH)主要负责菊糖型果聚糖(inulin)的水解,已经在大蒜的鳞茎中分离出具有这种活性的1-FEH[6]。不同植物物种中的FEHs已被克隆和分析:菊科如Vernonia herbacea(一种生于巴西的菊科植物)[7]、H.tuberosus(菊 芋)、Cichorium intybus(菊 苣)[8];禾 本 科 如Triticum aestivum(小 麦)[9-10]、Lolium perenne(黑 麦草)[11]、Bromus pictus(雀麦属)[12]、Beta vulgaris L.(甜菜)[13]、Arctium lappa(牛蒡)[14]。这几种植物 FEH 纯品都是通过cDNA克隆与测序的方法获得FEH基因后,再于毕赤酵母等微生物中表达,经过分离纯化获得的。而对于大蒜果聚糖酶的研究,仅有关于大蒜果聚糖外切酶的一些酶学特征的研究。而利用基因工程的方法克隆并表达大蒜FEH的基因的报道,国内外尚未见到。研究大蒜水解酶,及大蒜中果聚糖[EC3.2]的含量,有重要的商业价值和产业应用价值。本实验的目的在于克隆出大蒜中 FEH的cDNA,并分析其功能,以为进一步研究FEH基因在果聚糖代谢中的表达情况、FEH在贮存保鲜或加工的应用奠定基础。
1 材料与方法
1.1 材料与仪器
大蒜(Allium sativum L.)山东金乡产,购于广州市石牌农贸市场;感受态大肠杆菌DH5α、PMD-19 T Vector 均购自 TaKaRa公司;RNAiso Plus、PrimeScriptⅡ 1st Strand cDNA Synthesis Kit、Ex Taq、DNA Marker、3'-Full RACE Core Set Ver.2.0、5'-Full RACE Kit 宝生物工程(大连)有限公司产品;苯酚、氯仿、异丙醇、乙醇等 为国产分析纯试剂;液氮。
PCR仪 德国Eppendorf AG;核酸蛋白测定仪德国Eppendorf AG;JY600+电泳仪 北京君意东方电泳设备有限公司;SHA-B水浴恒温振荡器 江苏金坛市宏华仪器厂。
1.2 实验方法
1.2.1 大蒜总RNA的提取 切取15~30mg超低温冻结的大蒜鳞茎组织,迅速转移至-80℃预冷的研钵中研磨,其间不断加入液氮,直至研磨成粉末状后,加入适量RNAiso Plus,再依次使用氯仿、异丙醇抽提,RNA沉淀用75%的乙醇清洗后溶于RNase-free水中,保存于-80℃。
检测RNA的纯度和完整性。测定所获RNA样品的OD260和OD280以及2.0%凝胶电泳凝胶判断RNA的纯度和完整性。
1.2.2 合成cDNA第一条链 根据试剂盒PrimeScriptⅡ1st Strand c DNA Synthesis Kit的方法,以获得的 RNA为模板,以 oligo(dT)为引物,合成cDNA第一条链。
1.2.3 大蒜FEH基因的中间片段的扩增 根据已知的高等植物FEH基因的保守氨基酸序列NWMNDPNGPM和WECPDF设计引物 Gl-FEHa、Gl-FEHb(表1),以cDNA为模板进行PCR反应。PCR条件为:94℃预变性 5min;94℃ 30s,50℃ 30s,72℃1min,35个循环;72℃延伸7min。用Ex Tag DNA合成酶。
将PCR产物克隆到PMD-19 T载体上,并利用感受态大肠杆菌DH5α细胞进行阳性筛选。提取质粒纯化。对插入的DNA序列送上海生物工程公司测序,测序序列命名为DegeFEH-16。
1.2.4 c DNA全长扩增 利用c DNA末端扩增技术(RACE)获取大蒜果聚糖的cDNA全长。5'RACE扩增:根据已获得的DegeFEH-16的 DNA序列,设计引物 CTF333 R2、CTF333 R3(表1)。按照TaKaRa 5'-Full RACE Kit试剂盒说明,将处理好的Total RNA与 5'Adaptor连接后,反转录合成cDNA,同时设立M-MLV(-)对照。用CTF333 R2与5'RACE Outer Primer进行 Outer PCR,用 CTF333 R3与5'RACE Inner Primer进行Inner PCR。PCR条件为:94℃预变性 3min;94℃ 30s,55℃ 30s,72℃ 2min,30个循环;72℃延伸10min。
3'RACE扩增:根据已经获得的DegeFEH-16的DNA序列,设计引物 CTF333 F1、CTF333 F3(表1)。Total RNA 与 3'Adaptor后,,使用 TaKaRa 3'-Full RACE Core Set Ver.2.0将RNA反转录成cDNA,同时设立M-MLV(-)对照。用 CTF333 F1与3'RACE Outer Primer进行 Outer PCR,用 CTF333 F3与 3'RACE Inner Primer进行 Inner PCR。PCR条件为:94℃预变性 3min;94℃ 30s,55℃ 30s,72℃ 2min,30个循环;72℃延伸10min。
经RACE扩增后的PCR产物均进行切胶回收,并使用TaKaRa DNA Ligation Kit Ver.2.0中的连接酶,将回收产物与pMD20-T Vector连接,热转化至E.coli Competent Cells JM109中,涂布平板,37℃过夜培养。挑选阳性菌落,提取质粒进行测序。
1.2.5 序列拼接、RACE序列验证 将获得的ORF部分序列、5'序列和3'序列用DNAstar分析软件进行全长拼接。根据得到的全长序列,设计并合成引物CTF333 F5、CTF333 R8(表 1),用反转录产物进行PCR。将PCR产物进行琼脂糖凝胶电泳检测。
1.2.6 序列结构分析及同源性分析 应用SignalP 3.0Server和ExPASy等对cDNA全长序列进行序列分析,包括寻找ORF,推导编码氨基酸序列、预测信号肽及其保守结构域。
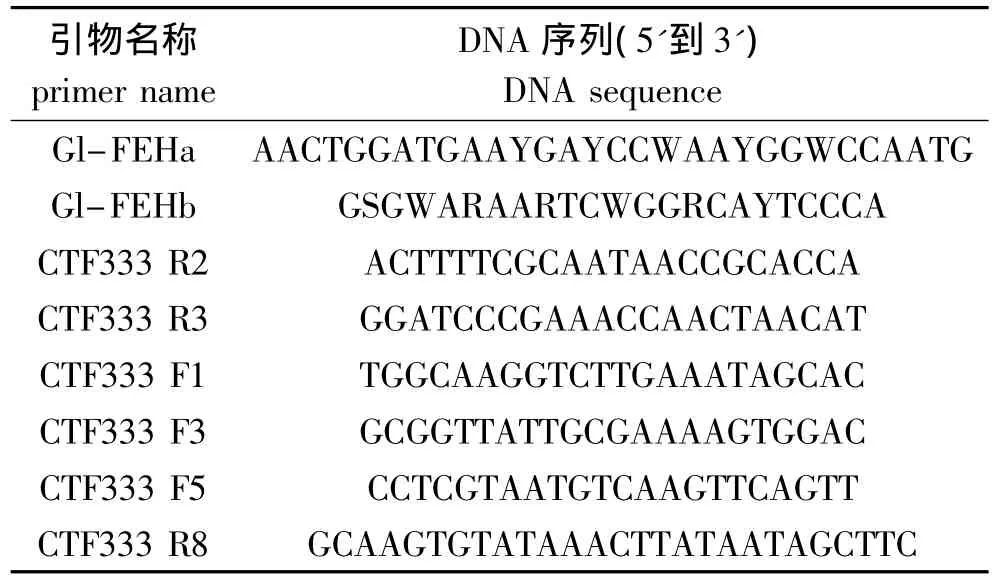
表1 引物序列Table 1 Primer sequence
将所获得的大蒜FEH基因的cDNA序列及编码的氨基酸序列放入NCBI的GenBank中进行BLAST,分析其和已收录植物FEH的同源性。用软件Clustal X和MEGA 4.0程序中的邻接法(Neighbor-Joining,NJ)构建系统发育树。
2 结果与分析
2.1 总RNA提取结果检测
提取大蒜总RNA进行2%琼脂糖凝胶电泳,结果如图1所示。从电泳结果看,总RNA的28s和18s两条带较为清晰,表明RNA降解不严重,完整性较好。检测总RNA样品A260/A280为2.04,纯度符合实验要求。综合以上检测结果,提取的RNA可以用于反转录。

图1 总RNA的琼脂糖电泳凝胶Fig.1 Agarose gel electrophoresis of total RNA
2.2 中间片段DegeFEH-16的PCR结果
经过PCR得到大小在500~750bp之间的中间片段(图2),经质粒测序,得到569bp的基因序列。将序列在NCBI中 BLAST检索,该序列与已收录的FEH序列有高度同源性。
2.3 3'RACE结果
经反转录和PCR反应后,将反应液进行琼脂糖凝胶电泳(图3A)。经质粒测序得到1134bp的3'端未知序列。
2.4 5'RACE结果
经反转录和PCR反应后,将反应液进行琼脂糖凝胶电泳(图3B)。经质粒测序得到185bp的5'端未知序列。

图2 DegeFEH-16的琼脂糖凝胶电泳Fig.2 Agarose gel electrophoresis of DegeFEH-16

图3 RACE产物的琼脂糖凝胶电泳Fig.3 Agarose gel electrophoresis of product of RACE
2.5 cDNA全长拼接和RACE序列验证
将利用PCR得到的中间基因序列和通过RACE PCR方法获得的3'和5'端序列,用DNAStar软件拼接,获得了全长1900bp的序列,序列为(阴影部分是CDS区):


用引物CTF333 F5和CTF333 R8进行PCR验证RACE的结果,产物进行凝胶电泳(图3C)。经验证,得到的5'和3'序列是根据已知序列得到的。
2.6 cDNA全长序列分析、同源性比对和系统进化树构建
用DNAstar将获得的全长基因序列翻译成氨基酸序列为:

经ExPASy软件在线分析计算,所获序列全长1900bp,编码区(CDS 区)位于 33~1700bp,共编码555个氨基酸,信号肽的长度是27个氨基酸。其编码蛋白分子量和等电点pI分别为63453u和5.79。
将已获得的氨基酸序列与GenBank中已收录的其它高等植物FEH氨基酸序列相比对,该序列与禾本科植物的有较高的同源性,与小麦 FEH(BAE44509.1)同源性为53%,与黑麦(AAZ29514.1)的同源性为 52%。另外,与石刁柏(Asparagus officinalis)的一种细胞壁转化酶(BAF93492.1)同源性为64%,与石刁柏的6-酮糖水解酶(BAF93491.1)同源性为62%。
用软件Clustal X比对所获的氨基酸序列与其他植物的FEH氨基酸序列,所获序列含有保守氨基酸序列WMNDP、RDP和WECVD。(图4)

图4 大蒜和其他已知高等植物的FEH的保守氨基酸序列比对结果Fig.4 Comparison of deduced amino-acid sequence of FEH from Allium sativum L.with FEHs reported from other plants
用MEGA 4.0程序中的邻接法(Neighbor-Joining,NJ)构建系统发育树如图5所示。

图5 基于大蒜和其他已知高等植物的FEH的氨基酸序列的系统进化树Fig.5 Phylogenetic analysis ofAllium sativum L.FEH with related FEH
3 结论
3.1 如上所述,我们获得的cDNA序列全长1900bp,含1668bp的CDS区,编码555个氨基酸的多肽链,经ExPASy软件分析预测其蛋白分子量为63453u,该分子量与已报道的、经SDS-PAGE测得大蒜1-FEH的分子质量[6]、即61700u基本一致。
3.2 本文所获得的氨基酸序列中,包含WMNDP、RDP和WECVD三个保守氨基酸序列,与已知的菊苣、牛蒡、黑麦及小麦等植物的FEH中的保守序列基本吻合。这也表明所获上述基因应当是FEH。我们注意到:扩增中间片段时,利用的是已知保守氨基酸序列NWMNDPNGPM和WECPDF,与最终获得的序列中相应序列不能完全对应,但这既可能是因为大蒜中三个功能区的氨基酸序列与已知的FEH保守序列不完全一致,也可能是因为利用氨基酸序列设计引物时有兼并碱基、或基因测序时有个别碱基的突变。其具体原因有待进一步研究证实。
3.3 本实验获得的氨基酸序列与小麦、黑麦的FEH同源性为53%。根据高等植物中果聚糖的研究[2],菊粉型果聚糖(1-FEH)主要存在于双子叶植物中,如菊芋、菊苣,而梯牧草型果聚糖(6-FEH)常见于单子叶植物中,如禾本科的小麦。一般同属一科的植物,FEH同源性较高,如同属菊科的牛蒡,与菊苣的同源性为82%,与斑鸠菊的同源性为80%,但与菊苣中的另一FEH同源性仅为53%[14]。属不同科的植物,一般FEH同源性较低,如藜科的甜菜与菊科的菊苣的同源性只要55%。所以只能对不同物种间的FEH同源性高低做相对的比较。大蒜属于百合科,单子叶植物,与禾本科同源性相对较近,但仍属于不同的科。本实验获得的氨基酸序列与小麦、黑麦的FEH同源性(53%)高于属双子叶的菊科植物及甜菜。
3.4 本实验获得酶的序列与属百合科的石刁柏中的转化酶(invertase,蔗糖酶)有较高同源性,且高于其与FEH的同源性。根据已有研究,FEH不能降解蔗糖分子,需要通过细胞壁上的转化酶(invertase)彻底水解,FEH可能是从转化酶进化而来。分析已收录的FEH的氨基酸个数约为560~580。而石刁柏中的转化酶的氨基酸个数为750、739。可以排除本实验所获的555个氨基酸序列不是转化酶。
3.5 基于以上已有大蒜1-FEH的研究结果、保守氨基酸序列、同源性等分析与讨论,可以初步推断本实验得到的是大蒜中的FEH。但仍需要进一步通过异源表达、测定酶活性、反应物分析等进行验证。
[1]唐建平,王正鹏,张树珍,等.高等植物果聚糖分类和功能的研究进展[J].海南大学学报:自然科学版,2009(27):308-312.
[2]谢柳.成善汉.高等植物果聚糖研究进展[J].植物学通报,2002(19):280-289.
[3]Van den Ende W,De Coninck B,Van Laere A,et al.Plant fructan exohydrolases:a role in signaling and defense[J].Trends in Plant Science,2004(9):523-528.
[4]王志敏.高等植物的果聚糖代谢[J].植物生理学通讯,2000(36):71-76.
[5]李慧娟,孙建全,李俊峰,等.高等植物果聚糖合成途径、相关基因和功能的研究进展[J].中国农学通报,2007(9):186-191.
[6]Kit Leong Cheong,Fengchao Yan,Xuesong Huang,et al.Enymogic Characterization of Garlic Fructan Exohydrolase[J].Journal of Food Chemistry,2012,36(2):248-253.
[7]Asega AF,do Nascimento JR,Schroeven L,et al.Cloning,characterization and functional analysis of a 1-FEH cDNA from Vernonia herbacea(Vell.)Rusby [J].Plant and Cell Physiology,2008,49:1185-1195.
[8]Van den Ende W,Michiels A,De Roover J,et al.Cloning and functionalanalysis ofchicory rootfructan1-exohydrolase I(1-FEH I):a vacuolarenzyme derivedfrom a cell- wall invertase ancestor?Mass fingerprint of the 1-FEH I enzyme[J].Plant Journal,2000(24):447-456.
[9]Kawakami A,Yoshida M,Van den Ende W,et al.Molecular cloning and functional analysis of a novel 6&1-FEH from wheat(Triticum aestivum L.)preferentially degrading small graminans like bifurcose[J].Gene,2005,358:93-101.
[10]Jingjuan Zhang,Shaobai Huang,John Fosu- Nyarko,et al.The genome structure of the 1-FEH genes in wheat(Triticum aestivum L.):new markers to track stem carbohydrates and grain filling QTLs in breeding[J].Molecular Breeding,2008(22):339-351.
[11]Jeremy L,Bertrand L,Katrien L R,et al.Cloning,gene mapping,and functional analysis of a fructan 1-exohydrolase(1-FEH)from Lolium perenne implicated in fructan synthesis rather than in fructan mobilization[J].Journal of Experimental Botany,2007(8):1969-1983.
[12]Florencia del Viso,Andrea F Puebla,H EstebanHopp,et al.Cloning and functionalcharacterization ofa fructan 1-exohydrolase(1-FEH)in the cold tolerant Patagonian species Bromus pictus[J].Planta,2009(1):13-25.
[13]Wim Van den Ende,Barbara De Coninck,Stefan Clerens,et al.Unexpected presence of fructan 6-exohydrolases(6-FEHs)in non-fructan plants:characterization,cloning,mass mapping and functional analysis of a novel‘cell-wall invertase-like’specific 6-FEH from sugar beet(Beta vulgaris L.)[J].Plant Journal,2003(36):697-710.
[14]Keiji Ueno,Yojiro Ishiguro,Midori Yoshida,et al.Cloning and functional characterization of a fructan 1-exohydrolase(1-FEH)in edible burdock(Arctium lappa L.)[J].Chemistry Central Journal,2011(5):697-710.
