絮凝基因内衔接重复序列与酵母菌絮凝特性多样性及遗传稳定性
2013-09-04岳峰郭雪娜何秀萍张博润
岳峰,郭雪娜,何秀萍,张博润
1 中国科学院微生物研究所 酵母菌分子遗传与育种实验室,北京 100101
2 中国科学院大学,北京 100049
高度重复序列广泛存在于真核生物和一些原核生物的基因组中,衔接重复的DNA序列是基因组中高度动态化 (非常活跃) 的成分。多数重复序列位于基因之间,但也有一些位于基因内的编码序列或假基因中。在高等真核生物中,这些基因内重复序列数量的改变常常与某些疾病的发生密切相关,如Huntington’s舞蹈症、X型脆弱综合症等。作为单细胞真核生物,酵母菌基因组中也含有大量的高度重复序列,在酿酒酵母Saccharomyces cerevisiae基因组中有 44个开放阅读框 (Open reading frame,ORF) 存在基因内重复序列 (http://www.sequence.standorf.edu/group/yeast),它们多数编码的是细胞表面蛋白。编码絮凝蛋白的基因统称为FLO基因家族,是酵母菌基因组中典型的含基因内重复序列的基因,它们编码的细胞表面糖蛋白通过和其他细胞表面的寡聚甘露糖链之间相互作用,使酵母细胞之间相互聚集形成絮状或颗粒状细胞团,并迅速沉降到发酵液底部或漂浮到发酵液顶部,即产生絮凝表型[1-2]。不同的絮凝蛋白具有相似的模块式结构,即疏水的氨基端,主要参与配体 (糖分子或寡糖链) 的识别和相互作用;羧基端与 GPI锚定蛋白有很高的同源性,末端包括一个 GPI锚钩;蛋白的中间区域是富含丝氨酸和苏氨酸的高度重复序列,是发生 N-和 O-糖基化的主要区域[3-4](图 1a)。编码絮凝蛋白的基因中间存在大量衔接重复序列,它们驱动基因内或基因间的滑移和重组,在产生絮凝特性多样性的同时导致絮凝特性的遗传不稳定性,从而影响絮凝特性的可控应用。
1 絮凝基因及基因内重复序列
絮凝的发生是酵母细胞表面絮凝蛋白和其他细胞表面寡聚甘露糖链之间相互作用的结果,因此絮凝表型会受到游离糖分子的竞争性抑制[5]。根据对不同糖的敏感性,酵母菌的絮凝表型通常被分为絮凝型 (Flo1-type) 和新絮凝型(NewFlo-type) 两种,其中前者只受甘露糖的抑制,而新絮凝型则受多种糖 (甘露糖、葡萄糖、麦芽糖、蔗糖等) 的抑制。絮凝表型的差异取决于细胞内编码絮凝蛋白的基因序列。野生型酿酒酵母中编码絮凝蛋白的基因主要有FLO1、FLO5、FLO9、FLO10、FLO11和Lg-FLO1,其中Lg-FLO1编码的絮凝蛋白赋予酵母菌新絮凝表型,FLO11表达使酵母菌产生 Flo1型絮凝或类似生物膜结构,其他基因的表达使酵母菌只产生 Flo1型絮凝,这些基因的共同特征是存在大量的基因内重复序列 (图1b)。FLO1位于染色体Ⅰ的右臂上,1994年日本的 Watari等报道了完整的FLO1基因序列,它含有一个 4 614 bp的ORF,编码1 537个氨基酸组成的、富含丝氨酸和苏氨酸的蛋白,ORF中66.97%的序列为衔接重复序列,根据其编码氨基酸序列的一致性,这些重复序列可以划分为4个重复单元,其中重复单元A是135 bp的核苷酸序列重复18次,重复单元B是60 bp的核苷酸序列重复2次,重复单元C是153 bp的核苷酸序列重复3次,重复单元 D是 27 bp的核苷酸序列重复 3次[6];FLO5是FLO1的同源异型基因,位于Ⅷ号染色体上,其编码产物与Flo1的氨基酸序列相似性为96%,FLO5含有与FLO1相同的重复单元B和C,但重复单元 A中重复序列的个数比FLO1少了 10个,重复单元D的重复序列仅剩1个完整拷贝[7];FLO9位于染色体Ⅰ的左臂,其编码产物与Flo1的氨基酸序列相似性为 94%,FLO9也含有与FLO1相同的重复单元B和C,但重复单元A中重复序列的个数比FLO1少了5个,重复单元D中重复序列比FLO1少了1个完整拷贝[7];FLO10位于染色体Ⅺ上,其编码产物与 Flo1的氨基酸序列相似性为58%,基因内有两个拷贝的重复序列与FLO1中重复单元C的重复序列类似,另有两类重复序列,分别是78 bp的A1重复6次和105 bp的A2重复4次,并且存在间隔排列,它们与FLO1中的重复序列没有明显的同源性[7];位于染色体Ⅸ上的FLO11与上述基因间有很大的差异,其编码产物与 Flo1的氨基酸序列相似性仅为37%,重复序列占ORF的21.12%,可分为5种类型,其中30 bp的重复序列A3重复了6次,45 bp的重复序列A4重复了 26次,36 bp的重复序列A5重复了18次,135 bp的重复序列C1重复了4次,99 bp的重复序列C2重复了2次[8-9];Lg-FLO1位于染色体Ⅷ上,ORF为3 903 bp,其编码产物与Flo1的氨基酸序列相似性约为60%,重复序列占ORF的44.09%,其中与FLO1重复单元A中135 bp重复序列同源的有8个拷贝,与重复单元C中重复序列同源的有2个拷贝,并保留了FLO1中重复单元D中的一个重复序列拷贝,此外还有Lg-FLO1特有的重复序列,即4个拷贝的42 bp重复序列A6,14个拷贝的48 bp重复序列A7和4个拷贝的21 bp的重复序列C3[5,10]。
2 基因内重复序列变化引发的絮凝多样性
重复序列是基因内高度活跃的成分,通过驱动基因内或基因间的滑移和重组,改变基因内重复序列的数量或排列方式,从而影响絮凝蛋白的结构和功能,使细胞表现出絮凝多样性。
2.1 絮凝基因内重复序列与酵母细胞絮凝能力
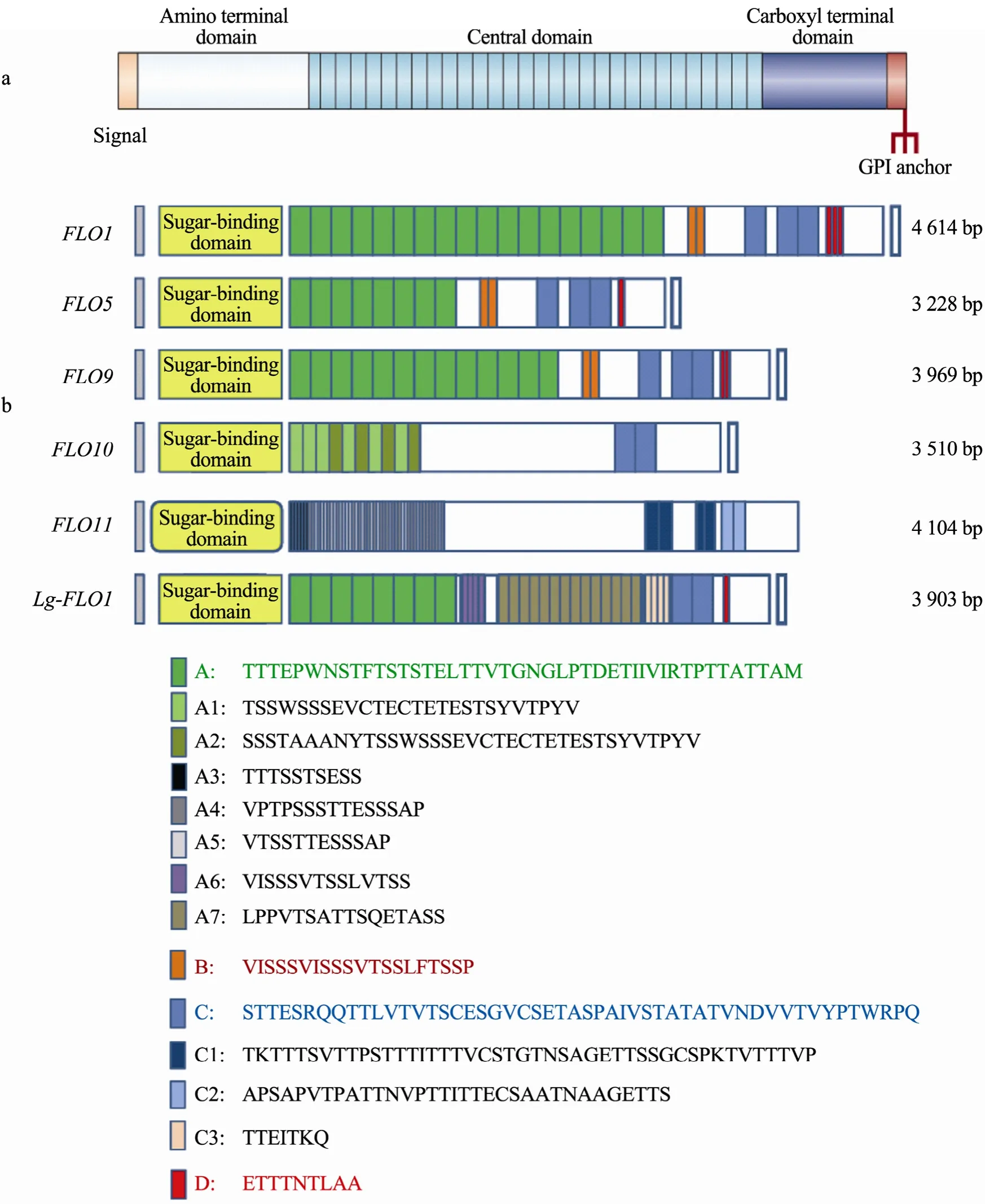
图1 絮凝蛋白模块式结构 (a)[3]及絮凝基因内重复序列 (b) 示意图Fig. 1 Schematic representation of the module structure of Flo protein (a)[3] and the intragenic repeats in FLO genes(b). Different color represents a kind of repetitive sequence, and the corresponding amino acid sequences are indicated.Analysis were conducted on the base of nucleotide sequences of FLO1 (AY949848), FLO5 (NM001179342), FLO9(NM001178205), FLO10 (NM001179892), FLO11 (NM001179541) and Lg-FLO1 (AB288349) deposited in GenBank.A, B, C and D in Figure B indicate the different repeat units in FLO.
完整絮凝基因FLO1及其重复序列数量发生变化的衍生基因FLO1M和FLO1S在非絮凝酵母中表达,带有完整絮凝基因FLO1的酵母细胞絮凝能力明显高于带有衍生基因的酵母细胞,其中重复序列数量最少的FLO1S基因赋予酵母细胞的絮凝能力最低[11-13],提示絮凝基因内重复序列数量与酵母细胞絮凝能力间有密切关系。Verstrepen等在美国哈佛大学和比利时鲁汶大学对FLO1基因内重复序列和细胞絮凝表型之间的关系进行了系统研究,他们通过基于URA3基因插入和敲除的5-氟乳清酸 (FOA) 抗性筛选模型获得基因内部重复序列个数发生改变的大小在2.9~5.4 kb的FLO1的衍生基因,这些基因间的差异主要是FLO1内重复单元A中重复序列数量的多少;对这些基因的表达分析证实重复序列的个数与细胞之间相互聚集产生絮凝的能力呈正相关,基因内重复序列越长,细胞所表现的絮凝能力越强[14]。2012年He等报道了一个来源于絮凝型工业酵母 SPSC01的FLO1类基因,即FLO1SPSC,该基因含有一个8 049 bp的ORF,编码2 682个氨基酸残基的蛋白质;序列比较发现该基因与完整FLO1相比,除了一些位点发生变异外,最明显的差异是重复单元A中重复序列的个数多了 25个;该基因在非絮凝型酵母中表达可以产生非常强的絮凝[15]。但由于没有不同基因表达的比较分析,因此重复序列的增多是否导致更强的絮凝还不清楚。对FLO10和FLO11的研究发现,基因内重复序列数量增加,增强了细胞表面疏水性,从而提高了细胞之间相互聚集的能力[16-17]。上述重复序列数量的变化主要发生在重复单元A区域,对FLO1中其他重复单元内重复序列进行不同程度的缺失,发现重复单元B、C、D分别全部缺失或部分缺失对细胞絮凝能力几乎没有影响[18-19]。因此,重复单元A所编码的多肽是絮凝蛋白中参与跨越细胞壁功能的主要结构域,该区域重复序列数量发生变化将影响絮凝蛋白跨越细胞壁在细胞表面的展现,同时影响细胞表面疏水性,从而影响邻近细胞之间的相互作用,使细胞表现出絮凝强度的差异。絮凝基因内重复序列的数量与细胞絮凝能力成正相关,但不是简单的线性关系[20]。
2.2 重复序列与絮凝蛋白和游离糖相互作用特异性和敏感性
存在于细胞表面的絮凝蛋白通过其氨基端与邻近细胞表面甘露糖链相互识别和作用使细胞发生聚集,产生絮凝表型;因此当环境中存在游离的糖分子时,可以通过与絮凝蛋白的竞争性识别和结合抑制细胞间的聚集和絮凝的发生。据此可以将絮凝表型划分为絮凝型 (Flo1-type) 和新絮凝型 (NewFlo-type)。通常认为不同糖对絮凝是否表现出抑制作用只与絮凝蛋白的氨基端糖结合域有关,只有此功能域发生突变才会导致糖识别和作用的特异性和敏感性发生变化[5,17,21]。但本实验室的研究发现,FLO1重复单元A内重复序列发生部分缺失或合并重复单元 B、C、D缺失导致酵母细胞絮凝表型发生转换[22-23]。我们从具有新絮凝 (NewFlo-type) 表型的酿酒酵母基因组文库中克隆到两个基因FLONS和FLONL,序列分析发现,絮凝蛋白 FloNS的1~302、303~615和616~1 132氨基酸序列与絮凝蛋白Flo1的1~302、527~839和1 021~1 537氨基酸序列完全相同,Flo1的两个内部重复序列303~526和840~1 020在FloNS中缺失;FLONS缺失的两段序列均位于FLO1基因重复单元 A中。推测FLONS可能是由于FLO1基因在染色体复制时基因内重复序列发生重组或重排产生的。而FLONL不但缺失了上述两个重复区域,还缺失了FLO1基因重复单元 B、C、D及其他部分3端编码序列,推测序列缺失发生在文库构建过程中由基因内重复序列引发的重组事件。FLONS和FLONL在非絮凝酵母细胞中表达产生的絮凝表型受多种糖 (甘露糖、葡萄糖、麦芽糖、蔗糖) 的抑制,是典型的新絮凝 (NewFlo-type)表型;更为有意思的是,国际上公认半乳糖对任何絮凝表型都没有抑制作用,但我们的研究表明FLONS或FLONL表达产生的絮凝受到半乳糖的抑制[22-24]。这是国内外首次报道FLO1基因内部重复序列数量变化不仅可以改变酵母细胞的絮凝能力,还可以改变絮凝蛋白与不同糖识别和相互作用的特异性。对FLO1中重复单元B、C、D的研究,发现这些重复单元分别发生缺失,不改变絮凝蛋白与游离糖分子相互作用的特异性,但降低了对甘露糖的敏感性[18-19]。可见絮凝基因内重复序列数量变化不仅影响细胞絮凝能力,同时对絮凝蛋白和糖分子之间相互识别和作用的特异性和敏感性也有重要的调控作用。这为通过培养液中糖种类和浓度变化对絮凝的发生进行时序控制奠定了理论基础。
2.3 絮凝基因内重复序列与絮凝的酸碱适应性
絮凝的发生除依赖于其遗传学基础外,还受到各种环境条件 (如温度、pH、金属离子种类和浓度、营养条件、细胞大小和世代等) 的影响。其中环境酸碱变化可以通过影响絮凝基因表达、细胞表面静电荷水平、絮凝蛋白活性等3个方面对酵母菌絮凝产生影响[1,25]。FLO1基因重复单元A中重复序列发生缺失,虽然使酵母细胞在最适pH条件下的絮凝能力降低,但使酵母细胞在偏酸或偏碱环境中的絮凝能力明显高于含有完整FLO1基因的酵母细胞,提高了絮凝对环境酸碱变化的适应性[20,23];FLONS与FLO1相比缺失了重复单元A中两个重复序列区域,在最适pH条件下,带有FLONS基因的酵母菌株絮凝能力是带有FLO1基因酵母菌絮凝能力的77.6%,但前者絮凝能力在pH 2.0~7.0范围内没有明显变化,而后者在pH 2.0、pH 7.0条件下的絮凝能力分别比在pH 4.0时降低了10%和30%[23]。絮凝基因FLO1a3与FLO1相比重复单元A中重复序列减少了12个拷贝,在最适pH条件下,带有FLO1a3基因的酵母菌絮凝能力是带有FLO1基因酵母菌絮凝能力的70.5%;与最适pH条件下相比,前者在pH 2.0和pH 9.0条件下的絮凝能力分别降低了47.8%和37.3%,而后者在pH 2.0、pH 9.0条件下的絮凝能力分别降低了58.9%和91.6%[20]。FLO1基因内重复单元B、C、D中重复序列发生缺失同样提高了酵母菌絮凝对环境酸碱变化的适应性[18-19];对重复单元C内重复序列的进一步分析,发现FLO1基因内衔接重复序列发生缺失增强了絮凝蛋白在环境酸碱变化中的构象稳定性,从而提高了酵母菌絮凝特性对环境酸碱变化的适应性[19]。这种基于重复序列变化产生的广泛的环境酸碱适应性对满足工业发酵过程和环境修复中的多样化需要具有重要意义。
3 絮凝基因的遗传不稳定性
Sato等在对底层发酵酿酒酵母的研究中发现经过连续传代培养后,细胞絮凝能力表现出逐渐降低的趋势,而细胞的其他生理生化特性却没有明显变化[13]。絮凝特性的这种遗传不稳定性是由絮凝基因家族的不稳定性引起的,该家族基因有着非常显著的分化能力[26-27],产生这一现象的原因一方面是由于絮凝基因家族 (除FLO11外)都位于染色体末端[10,28],因而有大量的重排事件发生,从而造成基因的重复、缺失以及转位[29];另一方面是由于絮凝家族基因内部普遍存在大量的重复序列[14,30],这些重复序列作为基因组中非常活跃的成分,导致了絮凝基因内部和基因之间的大量重组,而且重复序列区域的突变频率是常规突变的100倍以上[14]。因此絮凝特性存在遗传不稳定的严重问题。
目前对重复序列间如何相互作用而导致重组事件发生还没有非常清楚的了解,推测重组事件主要是由于 DNA复制过程中FLO基因内或基因间重复序列发生不对等的配对和交换的结果[3,14]。Verstrepen等利用URA3-FOA抗性筛选系统,分析比较了FLO1内衔接重复序列在DNA修复和重组突变体中发生重组的频率,发现重组事件的发生与由于 DNA复制障碍形成的双链断裂密切相关[14]。我们通过大肠杆菌和酿酒酵母双重筛选模型分析连续传代培养过程中絮凝基因序列、细胞絮凝能力及遗传稳定性变化,并结合相关的生物信息学分析,发现絮凝基因重复单元A中存在重组热点序列,缺失上述序列使絮凝特性的遗传稳定性明显提高[19,31]。
4 总结与展望
良好的絮凝特性既是酵母细胞抵抗环境胁迫的一种群体保护机制,同时为酵母菌参与的工业发酵过程提供了一种更加有效的、环境友好的、低廉的细胞分离和产品澄清途径。其在燃料乙醇、啤酒酿造等产业领域已有很好的研究和应用实例[24,32-36],同时在其他发酵工业中也凸显出非常重要的应用潜力[37]。近年来,利用酵母菌的絮凝特性去除工业废水中的 Cu2+、Ni2+、Cr3+、Cr6+、Zn2+、Cd2+等重金属离子也受到极大关注[37-38]。絮凝的发生是遗传因素和环境因素共同作用的结果,絮凝基因内高度重复序列与絮凝蛋白结构和功能,以及酵母菌絮凝特性有非常密切的关系,通过调控絮凝基因内重复序列种类和数量可以实现对酵母菌絮凝能力、环境酸碱变化适应性和遗传稳定性的相对定量控制,以及基于糖抑制特异性和敏感性的絮凝发生时序控制,从而为絮凝特性在发酵工业或环境修复过程中的可控应用提供新的解决策略。未来通过结构生物学等方法解析重复序列对絮凝蛋白结构及功能影响的具体机制,将为基于序列调控策略改造酵母菌絮凝特性提供更加准确的遗传修饰靶点。
[1]Verstrepen KJ, Derdelinckx G, Verachtert H, et al.Yeast flocculation: what brewers should know.Appl Microbiol Biotechnol, 2003, 61(3): 197−205.
[2]Bester MC, Pretorius IS, Bauer FF. The regulation ofSaccharomyces cerevisiaeFLOgene expression and Ca2+dependent flocculation by Flo8p and Mss11p. Curr Genet, 2006, 49(6): 375−383.
[3]Verstrepen KJ, Reynolds TB, Fink GR. Origins of variation in the fungal cell surface. Nat Rev Microbiol, 2004, 2(7), 533−540.
[4]Goossens KVY, Willaert RG. Flocculation protein structure and cell-cell adhesion mechanism inSaccharomyces cerevisiae. Biotechnol Lett, 2010,32(11): 1571−1585.
[5]Kobayashi O, Hayashi N, Kuroki R, et al. Region ofFLO1proteins responsible for sugar recognition.J Bacteriol, 1998, 180(24): 6503−6510.
[6]Watari J, Takata Y, Ogawa M, et al. Molecular cloning and analysis of the yeast flocculation geneFLO1. Yeast, 1994, 10(2): 211−225.
[7]Teunissen AW, Steensma HY. The dominant flocculation genes ofSaccharomyces cerevisiaeconstitute a new subtelomeric gene family. Yeast,1995, 11(11): 1001−1013.
[8]Lo WS, Dranginis AM.FLO11, a yeast gene related to theSTAgenes, encodes a novel cell surface flocculin. J Bacteriol, 1996, 178(24):7144−7151.
[9]Guo B, Styles CA, Feng Q, et al. ASaccharomycesgene family involved in invasive growth, cell-cell adhesion, and mating. Proc Natl Acad Sci USA,2000, 97(22): 12158−12163.
[10]Ogata T, Izumikawa M, Kohno K, et al.Chromosomal location ofLg-FLO1in bottom-fermenting yeast and theFLO5locus of industrial yeast. J Appl Microbiol, 2008, 105(4):1186−1198.
[11]Sieiro C, Reboredo NM, Blanco P, et al. Cloning of a newFLOgene from the flocculatingSaccharomyces cerevisiaeIM1-8b strain. FEMS Microbiol Lett, 1997, 146(1): 109−115.
[12]Watari J, Sato M, Ogawa M, et al. Genetic and physiological instability of brewing yeast. Euro Brew Convent Monographs, 1999, 28: 148−160.
[13]Sato M, Watari J, Shinotsuka K. Genetic instability in flocculation of bottom-fermenting yeast. J Am Soc Brew Chem, 2001, 59(3): 130−134.
[14]Verstrepen KJ, Jansen A, Lewitter F, et al.Intragenic tandem repeats generate functional variability. Nat Genet, 2005, 37(9): 986−990.
[15]He LY, Zhao XQ, Bai FW. Engineering industrialSaccharomycescerevisiaestrain with theFLO1-derivative gene isolated from the flocculating yeast SPSC01 for constitutive flocculation and fuel ethanol production. Appl Energ, 2012, 100(1): 33−40.
[16]Fidalgo M, Barrales RR, Ibeas JI, et al. Adaptive evolution by mutations in theFLO11gene. Proc Natl Acad Sci USA, 2006, 103(30): 11228−11233.
[17]Van Mulders SE, Christianen E, Saerens SMG, et al. Phenotypic diversity of Flo protein family-mediated adhesion inSaccharomyces cerevisiae. FEMS Yeast Res, 2009, 9(2): 178−190.
[18]Li EE, Chang Q, Guo XN, et al. Regulation of tandem repeats on the function of flocculation protein inSaccharomycescerevisiae. Acta Microbiol Sin, 2012, 52(1): 69−76 (in Chinese).
李娥娥, 常琦, 郭雪娜, 等. 衔接重复序列对酵母菌絮凝蛋白功能的调控作用. 微生物学报,2012, 52(1): 69−76.
[19]Li EE, Yue F, Chang Q, et al. Deletion of intragenic tandem repeats in unit C ofFLO1ofSaccharomycescerevisiaeincreases the conformational stability of flocculin under acidic and alkaline conditions. PLoS ONE, 2013, 8(1):e53428.
[20]Chang Q, Yue F, Guo XN, et al. Regulatory effect ofFLO1tandem repeats on the flocculation characteristics and genetic stability inSaccharomyces cerevisiae. Acta Microbiol Sin,2012, 52(11): 1360−1368 (in Chinese).
常琦, 岳峰, 郭雪娜, 等. 酿酒酵母FLO1基因内重复序列引发的絮凝能力稳定性研究. 微生物学报, 2012, 52(11): 1360−1368.
[21]Zupancic ML, Frieman M, Smith D, et al. Glycan microarray analysis ofCandida glabrataadhesin ligand specificity. Mol Microbiol, 2008, 68(3):547−559.
[22]Liu N, Wang DL, He XP, et al. Genetic basis of flocculation phenotype conversion inSaccharomyces cerevisiae. FEMS Yeast Res, 2007,7(8): 1362−1370.
[23]Liu N, Wang DL, He XP, et al. Deletion of tandem repeats causes flocculation phenotype conversion from Flo- to NewFlo-type inSaccharomyces cerevisiae. J Mol Microbiol Biotechnol, 2009,16(3-4): 137−145.
[24]Wang DL, Wang ZY, Liu N, et al. Genetic modification of industrial yeast strains to obtain controllable Newflo flocculation property and lower diacetyl production. Biotechnol Lett, 2008,30(11): 2013−2018.
[25]Jin YL, Speers RA. Effect of environmental conditions on the flocculation ofSaccharomyces cerevisiae. J Am Soc Brew Chem, 2000, 58(3):108−116.
[26]Halme A, Bumgarner S, Styles C, et al. Genetic and epigenetic regulation of theFLOgene family generates cell-surface variation in yeast. Cell, 2004,116(3): 405−415.
[27]Van Mulders, Ghequire SE, Daenen M, et al.Flocculation gene variability in industrial brewer’s yeast strains. Appl Microbiol Biotechnol, 2010,88(6): 1321−1331.
[28]Bhattacharyya MK, Lustig AJ. Telomere dynamics in genome stability. Trends Biochem Sci, 2006,31(2): 114−122.
[29]Miller T, Krogan NJ, Dover J, et al. COMPASS: A complex of proteins associated with a trithorax-related SET domain protein. Proc Natl Acad Sci USA, 2001, 98(23): 12902−12907.
[30]Rando OJ, Verstrepen K. Timescales of genetic and epigenetic inheritance. Cell, 2007, 128(4):655−668.
[31]Chang Q. Regulatory effect of tandem repeats inFLO1on the flocculation characteristics and genetic stability ofSaccharomyces cerevisiae[D].Beijing: Institute of Microbiology, Chinese Academy of Sciences, 2012 (in Chinese).
常琦.FLO1内重复序列对酿酒酵母絮凝特性及遗传稳定性的影响[D]. 北京: 中国科学院微生物研究所, 2012.
[32]Ishida-Fujii K, Goto S, Sugiyama H, et al. Breeding of flocculent industrial alcohol yeast strains by self-cloning of the flocculation geneFLO1and repeated-batch fermentation by transformants. J Gen Appl Microbiol, 1998, 44(5): 347−353.
[33]Cuncha AF, Missawa SK, Gomes LH, et al. Control by sugar ofSaccharomyces cerevisiaeflocculation for industrial ethanol production. FEMS Yeast Res,2006, 6(2): 280−287.
[34]Govender P, Domingo JL, Bester MC, et al.Controlled expression of the dominant flocculation genesFLO1,FLO5, andFLO11inSaccharomyces cerevisiae. Appl Environ Microbiol, 2008, 74(19):6041−6052.
[35]Zhao XQ, Bai FW. Yeast flocculation: new story in fuel ethanol production. Biotechnol Adv, 2009,27(6): 845−856.
[36]Li Q, Zhao XQ, Chang AK, et al. Ethanol-induced yeast flocculation directed by the promoter ofTPS1encoding trehalose-6-phosphate synthase 1 for efficient ethanol production. Metab Eng, 2012,14(1): 1−8.
[37]Bauer FF, Govender P, Bester MC. Yeast flocculation and its biotechnological relevance.Appl Microbiol Biotechnol, 2010, 88(1): 31−39.
[38]Machado MD, Santos MS, Gouveia C, et al.Removal of heavy metals using a brewer’s yeast strain ofSaccharomycescerevisiae: the flocculation as a separation process. Bioresour Technol, 2008, 99(7): 2107−2115.
