同一患者不同部位的4株肺炎克雷伯菌对碳青霉烯类抗生素耐药机制研究和同源性分析
2013-08-23沈立松
刘 瑛,俞 静,张 良,沈立松
肺炎克雷伯菌广泛分布于自然界,存在于土壤和水中,人和动物肠道内更为常见。肺炎克雷伯菌可引起典型的原发性肺炎,也可引起各种肺外感染,常见有婴儿肠炎、脑膜炎,成人泌尿系感染、外伤感染和血流感染等。碳青霉烯类抗生素是目前治疗肠杆菌科细菌包括肺炎克雷伯菌,尤其是产超广谱β内酰胺酶(ESBLs)和AmpC酶等多重耐药菌引起的严重感染最有效的抗菌药物之一。近年来,随着这类抗生素的广泛应用,临床逐渐出现了耐碳青霉烯类抗生素的肠杆菌科细菌,特别是耐碳青霉烯类抗生素肺炎克雷伯菌的报道日见增多,所涉及的国家和区域越来越广,成为临床抗该菌感染治疗的棘手问题。
本研究收集我院临床微生物室2012年3月从1例男性膀胱癌术后患者的血、尿、痰、盆腔引流液4种标本中分离得到的不重复的4株耐碳青霉烯类抗生素肺炎克雷伯菌,并对其耐药机制和同源性进行了分析,现将结果报道如下。
材料与方法
一、材料
(一)菌株来源 我院临床微生物实验室从1例男性膀胱癌术后患者的血、尿、痰和盆腔引流液中先后分离到4株对包括厄他培南和亚胺培南在内的所有临床常用抗菌药物均耐药的肺炎克雷伯菌,分别命名为Kpsck1、Kpsck2、Kpsck3和Kpsck4。质控株为大肠埃希菌ATCC 25922、铜绿假单胞菌ATCC27853、肺炎克雷伯菌ATCC700603,由上海市临床检验中心提供。产KPC-2酶肺炎克雷伯菌(LYKPC)由上海第六人民医院检验科惠赠。IMP和VIM阳性菌株由上海瑞金医院微生物科惠赠。
(二)仪器与试剂 VITEK2 Compact全自动微生物分析系统及革兰阴性杆菌鉴定卡(GNI)、革兰阴性杆菌药敏卡(GN13)购自法国Bio Mérieux公司,Mastercycler EP Gradient Thermal Cycler PCR仪购自德国Eppendorf公司,ABI Prism310 Genetic Analyzer DNA测序仪购自美国 Applied Biosystems公司,SDS-PAGE电泳装置及电泳仪购自美国Bio-Rad公司,PCR扩增试剂盒及DNA胶回收试剂盒购自上海生工生物工程公司,基因测序扩增试剂盒购自美国Applied Biosystems公司,厄他培南纸片(10μg)购自英国OXOID公司。
二、方法
(一)菌株鉴定和药敏试验 所有菌株均由VITEK-2 Compact全自动微生物分析系统进行鉴定及药敏试验,药敏判断标准及质控遵循2011年版CLSI[1]标准执行。
(二)改良Hodge试验 根据2009年版CLSI[2]推荐,将0.5麦氏单位的大肠埃希菌 ATCC 25922菌液稀释10倍后均匀涂布在MH平皿上,中间贴10μg/片厄他培南的纸片,将受试菌株以厄他培南纸片为起点,沿离心方向划线,35℃培养过夜,抑菌圈内呈现矢状者为阳性。肺炎克雷伯菌ATCC 700603为阴性对照株,肺炎克雷伯菌LYKPC作为阳性对照株。
(三)PCR扩增及测序 按文献[3]报道采用水煮法提取菌株DNA。采用PCR方法扩增碳青霉烯酶基因,包括blaKPC-2、blaIMP-1、blaIMP-2、blaVIM-1、blaVIM-2、blaNDM-1和blaOXA-487种常见基因。引物及扩增条件见参考文献(表1)。将PCR扩增产物进行琼脂糖凝胶电泳,于紫外凝胶成像仪观察并摄片。

表1 7种碳青霉烯酶基因的引物Table 1 Primers of 7 carbapenemase genes
(四)基因测序 待测序的PCR扩增阳性产物经过琼脂糖凝胶电泳分离后,割取对应位置的含清晰DNA条带的琼脂凝胶块,用DNA胶回收试剂盒回收得到测序单向PCR反应的模板,单向PCR反应的具体体系与扩增程序参考BigDye Terminator v3.1测序试剂盒操作手册,单向PCR反应产物利用ABI Prism310 Genetic Analyzer DNA测序仪测序。
(五)外膜蛋白SDS-PAGE电泳分析 通过超声破壁法制备肺炎克雷伯菌外膜蛋白[9],从细菌细胞膜蛋白成分中分离得到月桂酰肌氨酸钠不溶的蛋白组分作为外膜蛋白样品,对外膜蛋白样品进行SDS-PAGE电泳分析,电泳结束后采用考马斯亮蓝R250染色。
(六)ERIC-PCR DNA 指纹图谱分析 ERICPCR方法对4株耐碳青霉烯类肺炎克雷伯菌和另1株同期分离自同一科室其他患者的尿标本中肺炎克雷伯菌(Kpsck5)进行同源性分析。引物序列为5-AAGTAAGTGACTGGGGTGAGCG-3[3],PCR 反应体系和反应条件参见文献[10]。
结 果
一、菌株鉴定和药敏试验结果
4株分离自血、尿、痰和盆腔引流液标本的菌株经VITEK2 Compact全自动微生物分析系统鉴定均为肺炎克雷伯菌,生化编码均为:6607734653564010。药敏结果显示,4株细菌除了对厄他培南(MIC≥8 mg/L)和亚胺培南(MIC≥16 mg/L)耐药外,还对头孢唑林、头孢他啶、头孢曲松、头孢吡肟、氨苄西林-舒巴坦、哌拉西林-他唑巴坦、头孢替坦、氨曲南、阿卡米星、庆大霉素、环丙沙星、左氧氟沙星和甲氧苄啶-磺胺甲口恶唑耐药(表2)。
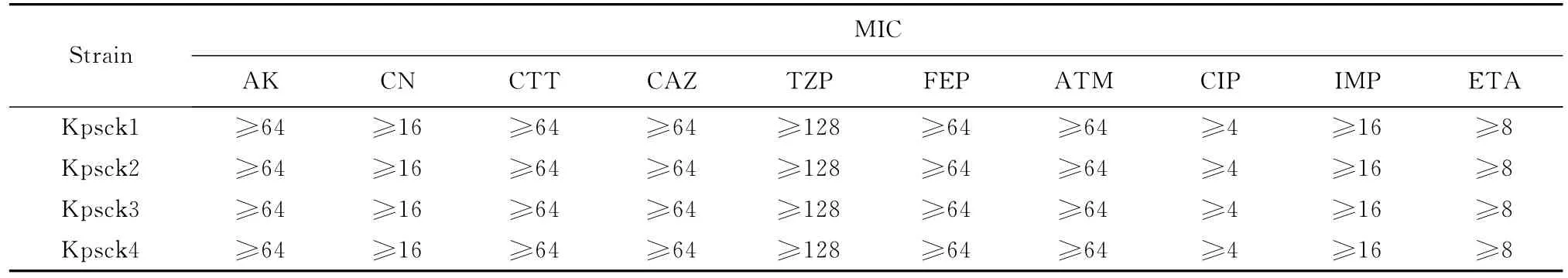
表2 4株肺炎克雷伯菌的药敏试验结果(mg/L)Table 2 The antimicrobial susceptibility of the 4 K.pneumoniae strains(mg/L)
二、改良Hodge试验
4株肺炎克雷伯菌改良Hodge试验结果均呈阳性(图1)。
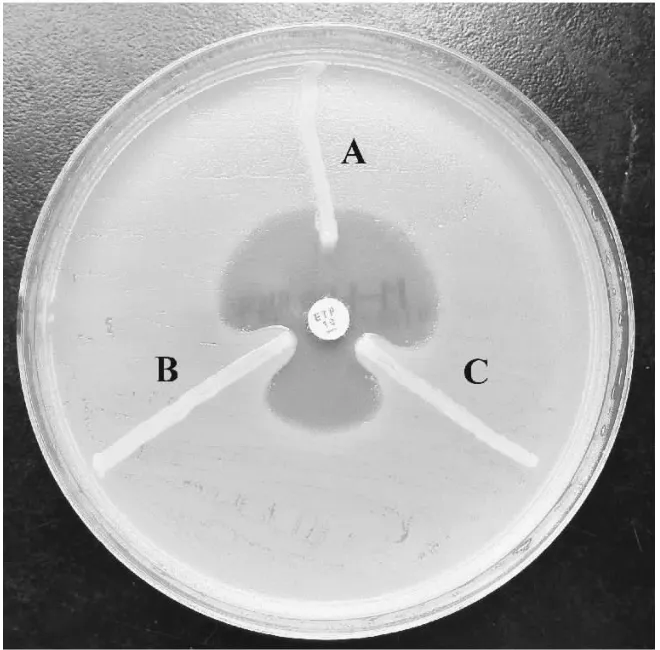
图1 肺炎克雷伯菌(Kpsck1)的改良Hodge试验结果Figure 1 Modified Hodge test result of Klebsiella pneumoniae(Kpsck1)
三、耐药基因检测及基因测序
对7种耐药基因进行PCR扩增,4株肺炎克雷伯菌均在1 000 bp左右,有KPC-2基因的阳性扩增条带(图2)。PCR产物测序后于美国NCBI网站Blast软件比对,证明PCR产物为KPC-2基因。4株 细 菌 均 未 检 测 到 blaIMP-1、blaIMP-2、blaVIM-1、blaVIM-2、blaNDM-1和blaOXA-48等耐药基因。
四、外膜蛋白组分分析
外膜蛋白SDS-PAGE电泳分析结果显示,Kpsck1、Kpsck2、Kpsck3和Kpsck4的外膜蛋白图谱完全相同,但与肺炎克雷伯菌ATCC700603的外膜蛋白图谱不同。Kpsck1、Kpsck2、Kpsck3、Kpsck4及肺炎克雷伯菌ATCC700603的外膜蛋白中均存在35 ku左右的蛋白条带。肺炎克雷伯菌ATCC700603表达的外膜蛋白中存在约36 ku的蛋白条带,Kpsck1、Kpsck2、Kpsck3和Kpsck4的外膜蛋白图谱中缺失36 ku大小的蛋白条带,而出现了一条分子量大于36 ku的异常蛋白条带(图3)。
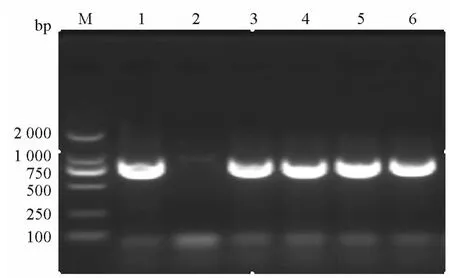
图2 KPC-2基因PCR扩增电泳图谱Figure 2 Electrophoresis of KPC-2 PCR amplification products
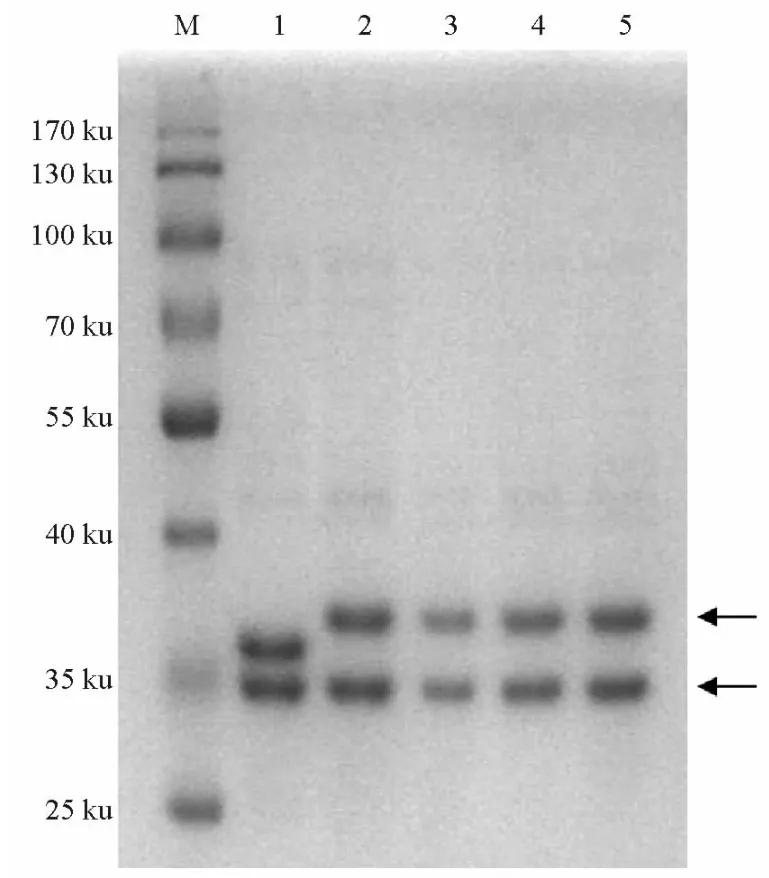
图3 肺炎克雷伯菌的外膜蛋白SDS-PAGE分析Figure 3 SDS-PAGE analysis of Klebsiella pneumoniae outer membrane porin
五、ERIC-PCR指纹图谱
ERIC-PCR试验的结果显示,Kpsck1、Kpsck2、Kpsck3和Kpsck4具有完全相同的DNA指纹图谱(图4),表明4株耐碳青霉烯类肺炎克雷伯菌是同一克隆株。
讨 论
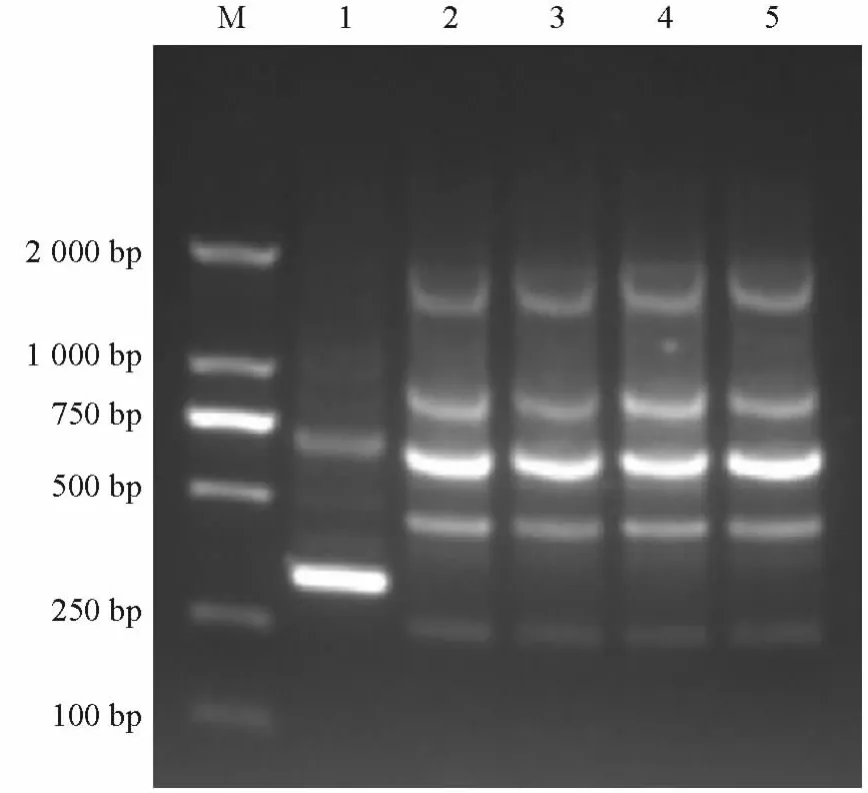
图4 5株肺炎克雷伯菌的ERIC-PCR指纹图谱Figure 4 ERIC-PCR fingerprinting profiles of Klebsiella pneumoniae
肠杆菌科细菌对碳青霉烯类抗生素的主要耐药机制是产碳青霉烯酶,且以肺炎克雷伯菌碳青霉烯酶(KPC)为多见。KPC酶属于Ambler分类的A类丝氨酸β内酰胺酶,2001年在美国北卡罗来纳州报道发现第1株产KPC酶的肺炎克雷伯临床分离株[11]。自此之后KPC型β内酰胺酶在诸多肠杆菌科细菌中被发现,迄今为止,已发现的KPC型酶达13种[12]。本研究中的4株肺炎克雷伯菌均检出KPC-2基因,而其他 Ambler B类酶包括IMP、VIM、NDM-1等金属酶及D类OXA-48型酶均未检出。因此,产KPC-2型碳青霉烯酶是这4株细菌对碳青霉烯类抗生素耐药最主要的耐药机制。KPC基因通常存在于可传播的含有转座子的质粒中,这可使细菌耐药性在不同种的细菌之间水平传播[13],增加了细菌获得耐药性的机会,引起医院内耐药菌的播散和流行,对控制医院感染和临床治疗是巨大的挑战。
外膜蛋白是细菌与外界进行物质交换的重要通道,改变通道蛋白性质和数量而降低细菌的膜通透性可以使细菌产生耐药性。肺炎克雷伯菌产生的主要外膜蛋白是OmpK35和OmpK36。已有文献报道,碳青霉烯类耐药的肺炎克雷伯菌中缺失36 ku大小的外膜蛋白组分而引起细菌耐药性增强[14-15]。本研究中Kpsck1、Kpsck2、Kpsck3和Kpsck4的外膜蛋白图谱中均缺失36 ku大小的蛋白条带,而出现了一条分子量大于36 ku的异常蛋白条带,异常外膜蛋白组分引起的细胞膜渗透性的改变与其碳青霉烯类耐药性相关,也可能导致此4株细菌的多重耐药性。因此,本研究中的4株耐碳青霉烯类抗生素肺炎克雷伯菌的主要耐药机制为产KPC-2酶合并外膜蛋白异常引起的外膜蛋白通透性改变。
ERIC-PCR主要是以肠杆菌科细菌基因间非编码区的重复共有序列为基础进行的PCR扩增,是一种简单、灵敏度高、可重复性和可靠性较好的分子生物学分型方法,可对肠杆菌科细菌的同源性进行分析。本研究中该例患者4个部位标本中均检出耐碳青霉烯类抗生素肺炎克雷伯菌,且结果显示为同一克隆型。表明该例患者是多脏器的全身泛耐药肺炎克雷伯菌感染。
Hussein等[16]和 Gasink等[17]通过临床资料对比研究,发现了感染碳青霉烯类抗生素耐药肺炎克雷伯菌的6种独立危险因素。回顾此患者的临床资料:年龄79岁,因膀胱癌行根治性膀胱切除术,4周后首次从尿标本中分离到泛耐药肺炎克雷伯菌,期间曾先后进行了帕尼培南、左氧氟沙星、亚胺培南和万古霉素等抗菌药物治疗,具备感染碳青霉烯类抗生素耐药肺炎克雷伯菌的危险因素。值得一提的是,该患者的临床标本分离到耐碳青霉烯类肺炎克雷伯菌后,临床选用了阿莫西林-克拉维酸、磷霉素和多西环素治疗,而后对患者血、尿、痰及盆腔引流液标本进行细菌培养,结果均为阴性。此案例说明,尽管药敏试验结果显示分离自患者的肺炎克雷伯菌为泛耐药株,但通过合适的抗菌药物治疗,尤其联合用药后细菌感染得到控制,这也给类似严重感染患者的治疗方案提供了参考依据。
[1] Clinical and Laboratory Standards Institute.Performance standards for antimicrobial susceptibility testing[S].Sixteenth Informational Supplement,2011,M100-S21.
[2] Clinical and Laboratory Standards Institute.Performance standards for antimicrobial susceptibility testing[S].Sixteenth Informational Supplement,2009,M100-S19.
[3] Grundmann HJ,Tower KJ,Kijkshoorn L,et al.Multicenter study using standardized protocols and reagents for evaluation of reproducibility of PCR-based fingerprinting of Acinetobacter spp[J].J Clin Microbiol,1997,35(12):3071-3077.
[4] Smith Moland E,Hanson ND,Herrera VL,et al.Plasmidmediated,carbapenem-hydrolysingβ-lactamase,KPC-2,in Klebsiella pneumoniae isolates[J].J Antimicrob Chemother,2003,51(3):711-714.
[5] Senda K,Arakawa Y,Ichiyama S,et al.PCR detection of metallo-β-lactamase gene blaIMPin gram-negative rods resist-ant to broad-spectrumβ-lactams[J].J Clin Microbiol,1996,34(12):2909-2913.
[6] Tsakris A,Pournaras S,Woodford N,et al.Outbreak of infections caused by Pseudomonas aeruginosa producing VIM-1 carbapenemase in Greece[J].J Clin Microbiol,2000,38(3):1290-1292.
[7] Krüttgen A,Razavi S,Imöhl M,et al.Real-time PCR assay and a synthetic positive control for the rapid and sensitive detection of the emerging resistance gene New Delhi Metallo-βlactamase-1(blaNDM-1)[J].Med Microbiol Immunol,2011,200(2):137-141.
[8] Poirel L,Héritier C,Tolün V,et al.Emergence of oxacillinase-mediated resistance to imipenem in Klebsiella pneumoniae[J].Antimicrob Agents Chemother,2004,48(1):15-22.
[9] Martínez-Martínez L,Pascual A,Hernández-Allés S,et al.Roles ofβ-lactamases and porins in activities of carbapenems and cephalosporins against Klebsiella pneumoniae[J].Antimicrob Agents Chemother,1999,43(7):1669-1673.
[10] 刘瑛,费奇力,陈峰,等.产碳青霉烯酶产气肠杆菌的ERICPCR图谱分析[J].中华医院感染学杂志,2011,21(11):2176-2178.
[11] Yigit H,Queenan AM,Anderson GJ,et al.Novel carbapenem-hydrolyzingβ-lactamase,KPC-1,from a carbapenem-resistant strain of Klebsiella pneumoniae [J]. Antimicrob Agents Chemother,2001,45(4):1151-1161.
[12] Lahey Clinic.β-lactamase classification and amino acid sequences for TEM,SHV and OXA extended-spectrum and inhibitor resistant enzymes.Available from:URL[EB/OL].http://www.lahey.org/studies/webt.asp.
[13] Munoz-Price LS,Quinn JP.The spread of Klebsiella pneumoniae carbapenemases:a tale of strains,plasmids,and transposons[J].Clin Infect Dis,2009,49(11):1739-1741.
[14] Bidet P,Burghoffer B,Gautier V,et al.In vivo transfer of plasmid-encoded ACC-1 AmpC from Klebsiella pneumoniae to Escherichia coli in an infant and selection of impermeability to imipenem in K.pneumoniae[J].Antimicrob Agents Chemother,2005,49(8):3562-3565.
[15] Leavitt A,Chmelnitsky I,Colodner R,et al.Ertapenem resistance among extended-spectrum-β-lactamase-producing Klebsiella pneumoniae isolates[J].J Clin Microbiol,2009,47(4):969-974.
[16] Hussein K,Sprecher H,Mashiach T,et al.Carbapenem resistance among Klebsiella pneumoniae isolates:risk factors,molecular characteristics,and susceptibility patterns[J].Infect Control Hosp Epidemiol,2009,30(7):666-671.
[17] Gasink LB,Edelstein PH,Lautenbac HE,et al.Risk factors and clinical impact of Klebsiella pneumoniae carbapenemasesproducing K.pneumoniae[J].Infect Control Hosp Epidemiol,2009,30(12):1180-1185.
