大豆分子标记辅助育种研究进展
2013-08-11长江大学园艺园林学院湖北荆州434025浙江省农业科学院作物与核技术利用研究所浙江杭州310021
高 莎 (长江大学园艺园林学院,湖北 荆州434025;浙江省农业科学院作物与 核技术利用研究所,浙江 杭州310021)
董德坤 (浙江省农业科学院作物与核技术利用研究所,浙江 杭州310021)
刘乐承 (长江大学园艺园林学院,湖北 荆州434025)
大豆 [Glycine max L.Merr.]起源于中国,是重要的植物蛋白质和食用油脂来源[1]。为克服传统育种方法的局限性,以增产、改善品质、增强抗性等为目标的分子育种技术也登上了大豆育种的舞台。实际上,从1994年抗除草剂转基因大豆推广以来,转基因大豆所占的市场份额越来越大,分子育种技术的研究和应用是世界大豆生产发展的重要动力[2]。近几年,随着转基因技术的日渐成熟和深入,美国等大豆生产先进国家扩大了领先优势[3],中国也加快了研究的步伐,并获得了长足的发展。2010年大豆基因组测序的完成并公布,使得大豆基因组研究有了突飞猛进的发展[4]。分子标记技术有助于在分子水平上对大豆相关性状进行遗传改良,但前提是要发现与重要性状紧密相关的QTL位点,通过遗传图谱进行QTL定位是近年来发展的研究数量性状遗传的主要手段[5-6]。
1 大豆分子遗传图谱与基因测序
遗传图谱的建立对近等基因系群体的分析、QTL定位和提高大豆单产都具有重要的意义[7]。中国第一个大豆分子遗传图谱是以限制性片段长度多态性 (Restriction Fragment Length Polymorphism,RFLP)为基础的,材料是长农4号×新民6号的F2代,随后用该组合的F8重组自交系 (RIL)在原有基础上添加了更多的遗传标记[5,8-9]。然后我国大豆遗传图谱的构建就变得频繁 (表1)。
早在20世纪80年代末期,国外就有了第一个分子遗传图谱[29]。它是由Cregan等[30]构建的含有1423个标记的遗传图谱,后来Song等[31]增加了426个标记,使其图谱长度为2523.6cM,这是迄今为止标记数目最多、密度最高、应用最为广泛的大豆 “公共图谱”。为更进一步明确遗传距离和物理距离的关系,也为了使分子遗传群体与染色体相对应,研究者们已将遗传图谱、物理图谱和染色体图谱联系到一起[32-33]。Hwang等[34]公布了以SSR标记为主高密度地大豆整合遗传连锁图谱,包含20个连锁群,用3个RIL群体作为材料。上述遗传图谱的建立,使大豆分子辅助选择育种研究及其重要性状的定位更有说服力,也为该领域的研究提供了可借鉴的信息。
通过全基因组鸟枪测序法,大豆1.1GB的基因组序列也已测通,科学家们在序列中整理定位了5500个遗传标记,是鸟枪法测序的植物中基因组最大的物种之一[35]。精准的大豆基因组序列图谱极大方便了目的基因的图位克隆,分子标记开发等工作,同时加快了大豆品种改良的节奏。
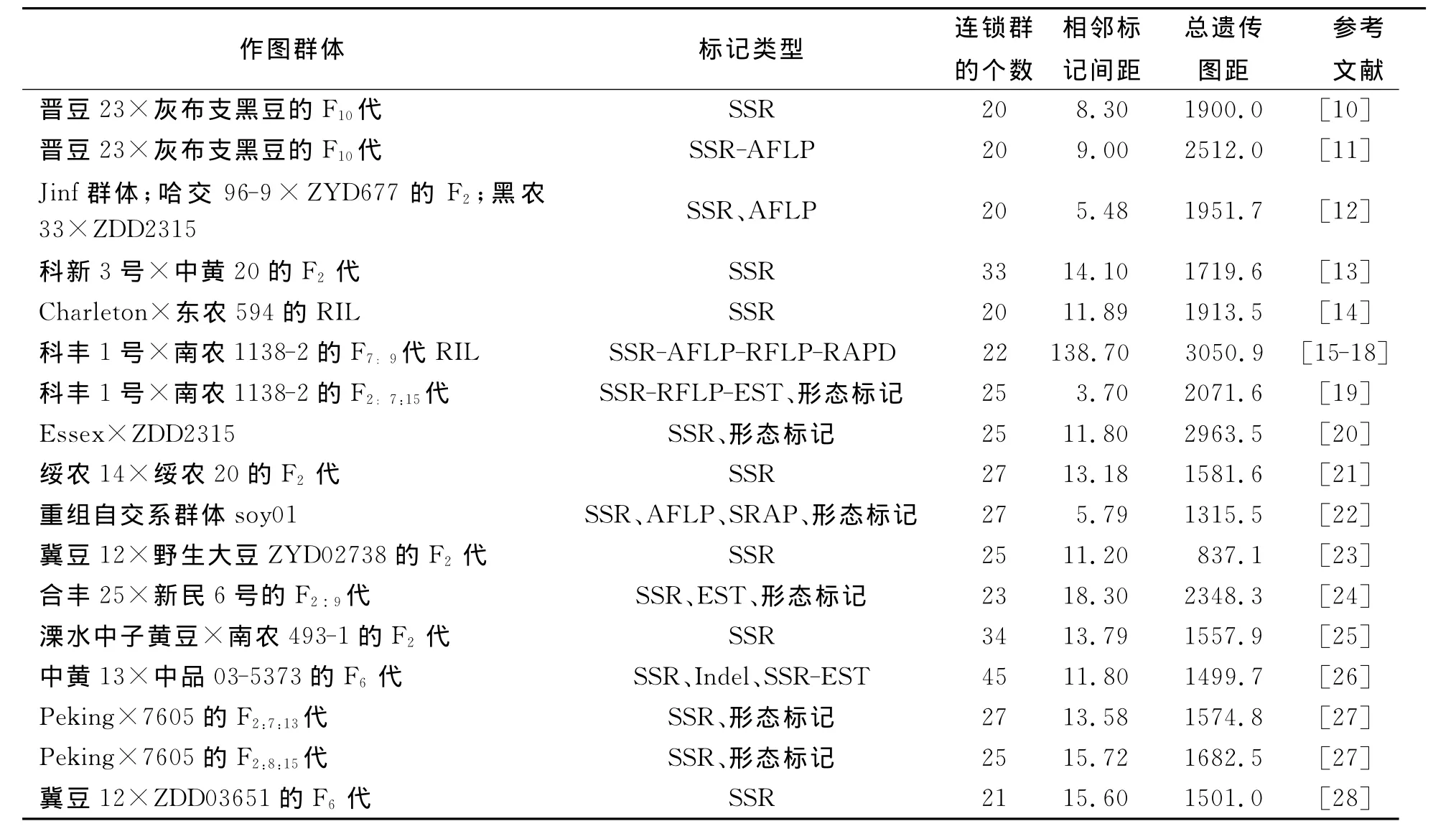
表1 2001年以来国内大豆遗传图谱的构建
2 大豆重要性状基因的标记和定位
2.1 产量及与产量相关QTL
大豆产量性状是典型的受多基因控制的数量性状。农艺性状QTL与产量性状是连锁的,大豆株高、成熟期、百粒重、节间长、收获指数等性状都与产量性状存在显著上位性效应[36-37],产量与地上部生物量、根重、冠层宽和高等均有极显著正相关,但叶面积指数与产量呈正或负相关,结果不一致[38]。
关于大豆产量及与其相关性状的QTL检测,据公布的数据已定位产量QTL 154个,株高QTL 172个,分枝数QTL 13个,节数和荚数各为33个 (http://soybase.org)。种子大小及形状相关的QTL也有报道[39]。对叶柄夹角QTL的研究也取得了可喜的成果[40-41]。蒋春志等[42]发现C2连锁群上聚集了与产量相关QTL位点。牛远等[43]研究表明,Glyma10g35240和Glyma10g34980可能是控制粒形性状发育的候选基因。孙亚男等[44-45]从已有的报道中收集到65个与百粒重相关QTL,随后又检测出13个与之相关QTL。
2.2 品质性状
蛋白质含量和油分含量的高低是评判大豆品质好坏的一个标准。Li等[46]用KF1与NN1138-2作材料研究种子含油量,检测到与其QTL相连的位点Satt685。梁慧珍等[47]、单大鹏等[48]都检测出与大豆蛋白质含量相关QTL。Pandurangan等[49]研究认为大豆种子蛋白质含量与天冬氨酸浓度呈正相关。魏崃等[50]研究认为,DNA片段长度为270bp时,Satt193可作为油分的分子筛选标记,而在DNA片段长度为220bp时,可用来筛选蛋白质材料。据soybase数据库公布的数据,已检测到165个与种子含油量相关QTL,与种子蛋白质含量相关QTL 137个。
大豆高异黄酮品种也受到育种者的重视。用中豆27×九农20的F5︰7代RIL群体为材料,定位了可用于高异黄酮品种的分子标记辅助选育的位点[51]。研究表明,A1染色体上与异黄酮含量相关的一个主效QTL能在多年多环境下稳定出现[52]。也有报道表明,F连锁群上的中等效应QTL与I连锁群的QTL存在上位性互作[53]。在Essex和PI437654为亲本的RIL群体中,检测到26个存在加性主效的QTL[54]。
2.3 抗病性
大豆包囊线虫 (SCN)由于致病力不同,生理小种亦不同,目前我国鉴定出有1、2、3、4、5、6、7、9和14号生理小种[55]。Rhg4位点是抗大豆孢囊线虫3号生理小种最主要的QTL之一[56]。Rhg1位点会减缓植株根系的生长,并推断Rhg1是显性多基因抗性位点[57]。盖钧镒等[58]研究表明,大豆的抗性是由包括1显3隐的相对独立的主基因决定的。以Magellan×PI438489B为材料,检测到5个与抗SCN相关QTL[59]。对大豆种质资源进行进行遗传多样性和关联性的分析,检测到抗SCN、抗大豆花叶病毒病 (SMV)、高油分含量、高蛋白质含量呈显著相关的SSR标记共有16个[60]。
大豆灰斑病菌 (FLS)是引起大豆灰斑病的主要原因。张文慧等[61]、陈立君等[62]研究认为,FLS 1号生理小种位于C1连锁群上。研究表明大豆对FLS的抗性由单个显性基因控制,同时筛选到与抗FLS基因连锁的3个RAPD标记[63]。对Blackhawk×Davis的F2:3代抗FLS分析,筛选到2个SSR标记[64],姜翠兰等[65]也检测到与抗病基因Rcs15紧密连锁的5个SSR标记。已有的研究报道了大量针对不同品种的抗性基因,但对病原遗传多样性知之甚少[66]。
大豆花叶病 (SMV)是大豆的主要病害之一。因SMV有不同的生理小种,所以研究的结果各不相同。Ma等[67]以齐黄1号×南农1138-2的RIL为材料,开发了一个SNP标记MY525。王大刚等[68]对国内外关于SMV抗性基因的分子标记及定位的研究以表格的形式进行综述,认为不同SMV株系的抗性基因是成簇出现的。郑翠明等[69-70]研究表明,品系95-5383对SMV3号株系的抗性受1对隐性基因控制,随后以HB1×ICGR95-5383为材料,发现大豆对SMV的抗性受单个隐性基因控制。Buss等[71]则认为SMV的抗性是受单显性基因控制,其所使用的研究材料是Lee68×PI96986的F2︰3。Jeong等[72]和Hayes等[73]的研究同样表明,大豆对SMV的抗性受单个基因控制。杨小凤等[74]研究认为Q0926对SC6、中豆35对SC17的抗病性分别由1对显性基因控制。
3 大豆分子标记选择
3.1 目标基因选择
国内关于大豆性状QTL分子标记的研究信息虽不丰富,然而研究者的努力有目共睹。一批对大豆细菌斑点病抗性较高的资源和品种得到初步鉴定75],现行大豆种质资源具有比较丰富的抗灰斑病资源[76],抗锈病的鉴定也有一定的进展[77],对大豆胞囊线虫病具有抗性的大豆种质资源区域也逐年增多[78-79]。相比之下,国外关于这方面的知识则更全面,最经典的研究成果是将外源大豆种质资源鉴定出的高产QTL定位于B2连锁群上,并将其转入到不同遗传背景高品质大豆中,发现该QTL在6个中的2个大豆遗传背景中有增产效果;虽然产量QTL在大豆遗传背景中的适应力有限,但同时也表明外源的种质资源具有提高大豆产量的可能性[80]。Kim等[81]的研究也表明,外来种质是新等位基因的来源,可以提高大豆产量。近年来关于抗蚜[82]、耐盐碱[83]等方面也有报道。
3.2 遗传背景选择
为提高轮回亲本的恢复率,在回交育种中需要进行遗传背景选择。大豆分子标记辅助背景选择的最初要明确适宜的标记数目和选择方式。首先用少数标记淘汰不符合选择要求 (要求遗传背景回复率高)的材料,其次是用较多标记对遗传背景回复率高的材料作精确地筛选,伴随着世代的增加,不再分析已经恢复为轮回亲本的标记位点,然后供选择标记的数目就会随着世代的递增而减少[84]。抗草甘膦商业品种BeningRR的育成,就是利用了SSR标记恢复轮回亲本Bening的基因组[2]。越南大豆锈病也利用此技术,使BC4F1代保留了抗锈病基因Rpp5[85]。回交世代在表型选择的基础上,利用分子标记辅助选择,可以加速育种进程、提高选择效率[80]。
4 展望
为丰富遗传图谱的内容,应加大遗传图谱的密度以及遗传图谱与染色体图谱、物理图谱的整合[86]。其次,在重要性状QTL定位方面,虽然分子标记的开发取得了长足的进展,仍应特别重视不同材料的相关分子标记比较鉴定,以找出具有共同特征的分子标记,因为QTL位置的准确度和稳定性仍受限制[69-70,87],目前也没有针对其精准度的QTL定位软件出现。再次,QTL定位的关键是有良好的作图群体,如近等基因系群体,作图群体个体间遗传背景差异程度以及作图群体对QTL检测的分辨率都会影响分子标记的定位准确度,可由于大豆进行人工杂交困难,想要获得回交种子需花费大量的时间和精力[41]。
[1]Miles M R,Frederick R D,Hartman G.Soybean rust:Is the US soybean crop at risk?[R].American Phytopathological Society,2003.
[2]James C.Global Status of Commercialized Biotech/GM Crops[M].Ithaca:The International Service for the Acquisition of Agri-biotech Applications(ISAAA),2011.
[3]蒋炳军,岳岩磊,王彩洁,等.大豆分子育种研究进展 [J].大豆科学,2012,31(4):662-667.
[4]张 乐,郭政阳,刘玉林,等.大豆分子标记遗传图谱研究进展 [J].科技信息,2012,(17):135.
[5]万志兵,戴晓港,尹佟明.林木遗传育种基础研究热点述评 [J].林业科学,2012,48(2):150-154.
[6]邱丽娟,王昌陵,周国安,等.大豆分子育种研究进展 [J].中国农业科学,2007,40(11):2418-2436.
[7]张 芳,谢 皓,陈学珍.大豆分子标记辅助育种研究进展 [J].北京农学院学报,2010,25(2):73-77.
[8]张德水,董 伟,惠东威,等.用栽培大豆与半野生大豆间的杂种F2群体构建基因组分子标记连锁框架图 [J].科学通报,1997,42 (12):1326-1330.
[9]刘 峰,陈受宜,庄炳昌.大豆基因组F连锁群较高密度图谱的构建和基因定位 [J].自然科学进展,2000,10(11):1012-1017.
[10]宛煜嵩,王 珍,肖英华,等.一张含有227个SSR标记的大豆遗传连锁图 [J].分子植物育种,2005,3(1):15-20.
[11]吕 蓓,张丽霞,宛煜嵩,等.用AFLP标记饱和大豆SSR遗传连锁图 [J].分子植物育种,2005,3(2):163-172.
[12]巩鹏涛,木金贵,赵金荣,等.一张含有315个SSR和40个AFLP标记的大豆分子遗传图的整合 [J].分子植物育种,2006,4(3):309-316.
[13]杨 喆,关荣霞,王跃强,等.大豆遗传图谱的构建和若干农艺性状的QTL定位分析 [J].植物遗传资源学报,2004,5(4):309-314.
[14]陈庆山,张忠臣,刘春燕,等.应用Charleston×东农594重组自交系群体构建SSR大豆遗传图谱 [J].中国农业科学,2005,38(7):1312-1316.
[15]Zhang W K,Wang Y J,Luo G Z,et al.QTL mapping of ten agronomic traits on the soybean(Glycine max L.Merr.)genetic mapand their association with EST markers[J].Theor Appl Genet,2004,108:1131-1139.
[16]吴晓雷,贺超英,王永军,等.大豆遗传图谱的构建和分析 [J].遗传学报,2001,28(11):1051-1061.
[17]贺超英,张志永,王永军,等.利用微卫星标记评估大豆重组近交系NJRIKY [J].遗传学报,2001,28(2):171-181.
[18]王永军,吴晓雷,贺超英,等.大豆作图群体检验与调整后构建的遗传图谱 [J].中国农业科学,2003,36(11):1254-1260.
[19]周 斌,邢 邯,陈受宜,等.大豆重组自交系群体NJRIKY遗传图谱的加密及其应用效果 [J].作物学报,2010,36(1):36-46.
[20]卢为国,盖钧镒,郑永战,等.大豆遗传图谱的构建和抗胞囊线虫 (Heterodera glycines Ichinohe)的QTL分析 [J].作物学报,2006,32 (9):1272-1279.
[21]朱晓丽.大豆遗传图谱构建及在两个群体重要农艺性状的QTL定位 [D].哈尔滨:东北农业大学,2006.
[22]王贤智,周 蓉,张晓娟,等.大豆遗传图谱的构建和含油量的QTL分析 [J].中国油料作物学报,2008,30(3):272-278.
[23]张 颖,刘兵强,刘立伟,等.冀豆12遗传图谱初步构建 [J].华北农学报,2009,24(2):60-63.
[24]吕祝章,丁立孝,田纪春,等.大豆重组自交系群体 (ZKS-HX)遗传图谱的构建和形态标记的定位 [J].植物生理学通讯,2009,45 (4):345-350.
[25]汪 霞.大豆遗传图谱构建和百粒重等七个农艺性状的QTL定位 [D].南京:南京农业大学,2010.
[26]刘玉林.大豆遗传图谱构建及重要农艺性状的QTL定位 [D].北京:中国农业大学,2011.
[27]洪雪娟,侯金锋,丁 卉,等.大豆异地衍生重组自交系群体遗传图谱的构建及比较 [J].作物学报,2012,38(4):614-623.
[28]雷雅坤,闫 龙,杨春燕,等.大豆公共遗传图谱C1连锁群SSR标记空白区段的填补 [J].华北农学报,2012,27(6):5-10.
[29]Apuya N R,Frazier B L,Keim P,et al.Restriction fragment length polymorphisms as genetic markers in soybean(Glycine max L.Merrill)[J].Theor Appl Genet,1988,75:889-901.
[30]Cregan P B,Jarvik T,Bush A L,et al.An integrated genetic linkage map of the soybean genome[J].Crop Science,1999,39:1464-1490.
[31]Song Q J,Marek L F,Shoemaker R C,et al.A new integrated genetic linkage map of the soybean[J].Theor Appl Genet,2004,109:122-128.
[32]Cregan P B,Kollipara K P,Xu S J,et al.Primary trisomics and SSR markers as tools to associate chromosomes with linkage groups in soybean[J].Crop Science,2001,41:1262-1267.
[33]Zou J J,Singh R J,Hymowit Z T.Association of the yellow leaf(y10)mutant to soybean chromosome3[J].Journal of Heredity,2003,94:352-355.
[34]Hwang T Y,Sayama T,Takahashi M,et al.High-density integrated linkage map based on SSR markers in soybean[J].DNA Re-search,2009,16:213-225.
[35]Wilcox C H.美科学家完成大豆基因组测序 [J].农业生物技术学报,2010,18(1):191.
[36]Palomeque L,Liu L J,Li W B,et al.QTL in mega-environments:II.Agronomic trait QTL co-localized with seed yield QTL detected in a population derived from a cross of high-yielding adapted × high yielding exotic soybean lines[J].Theor Appl Genet,2009,119:429-436.
[37]Martin S K,Xie F T,Zhang H J,et al.Epistasis for quantitative traits in crosses between soybean lines from China and the United States[J].Crop Sci,2009,49:20-28.
[38]黄中文,赵团结,喻德跃,等.大豆产量有关性状QTL的检测 [J].中国农业科学,2009,42(12):4155-4165.
[39]Xu Y,Li H N,Li G J,et al.Mapping quantitative trait loci for seed size traits in soybean(Glycine max L.Merr.)[J].Theor Appl Genet,2011,122:581-594.
[40]仕相林,孙亚男,王家麟,等.大豆叶片性状QTL的定位及Meta分析 [J].作物学报,2012,38(2):256-263.
[41]王吴彬,何庆元,杨红燕,等.大豆分枝数和叶柄夹角的相关野生片段分析 [J].中国农业科学,2012,45(23):4749-4758.
[42]蒋春志,裴翠娟,荆慧贤,等.大豆品质及农艺性状的QTL分析 [J].华北农学报,2011,26(5):127-130.
[43]牛 远,谢芳腾,布素红,等.大豆粒形性状QTL的精细定位 [J].作物学报,2013,39(4):609-616.
[44]孙亚男,仕相林,蒋洪蔚,等.大豆百粒重QTL的上位效应和基因型×环境互作效应 [J].中国油料作物学报,2012,34(6):598-603.
[45]Qi Z M,Sun Y N,Wang J L,et al.Meta-Analysis of 100-Seed Weight QTLs in Soybean[J].Agricultural Sciences in China,2011,10:327-334.
[46]Li H,Zhao T,Wang Y,et al.Genetic structure composed of additive QTL,epistatic QTL pairs and collective unmapped minor QTL conferring oil content and fatty acid components of soybeans[J].Euphytica,2011,182:117-132
[47]梁慧珍,王树峰,余永亮,等.大豆异黄酮与脂肪、蛋白质含量基因定位分析 [J].中国农业科学,2009,42(8):2652-2660.
[48]单大鹏,朱荣胜,陈立君,等.大豆蛋白质含量相关QTL间的上位效应和QE互作效应 [J].作物学报,2009,35(1):41-47.
[49]Pandurangan S,Pajak A,Molnar S,et al.Relationship between asparagine metabolism and protein concentration in soybean seed[J].J Exp Bot,2012,63:3173-3184.
[50]魏 崃,孙鸿雁,唐晓飞,等.大豆遗传群体选择与品质QTL的获得 [J].分子植物育种,2009,7(4):727-735.
[51]Zeng G L,Li D M,Han Y P,et al.Identification of QTL underlying isoflavone contents in soybean seeds among multiple environments[J].Theor Appl Genet,2009,118:1455-1463.
[52]李 侠,常 玮,韩英鹏,等.大豆种子脂肪酸含量的遗传分析 [J].大豆科学,2009,28(3):402-408.
[53]Bachlava E,Dewey R E,Burton J W,et al.Mapping and comparison of quantitative trait loci for oleic acid seed content in two segregating soybean populations[J].Crop Sci,2009,49:433-442.
[54]Gonzalez J J,Wu X L,Zhang J,et al.Genetic control of soybean seed isoflavone content:importance of statistical model and epistasis in complex traits[J].Theor Appl Genet,2009,119:1069-1083.
[55]张姗姗,李英慧,李金英,等.优良品系中品03-5373系谱的遗传解析及抗大豆胞囊线虫病相关标记鉴定 [J].作物学报,2013,39 (10):1746-1753.
[56]Liu X,Liu S,Jamai A,et al.Soybean cyst nematode resistance in soybean is independent of the Rhg4locus LRR-RLK gene[J].Functional &integrative genomics,2011,11:539-549.
[57]Afzal A J,Srour A,Saini N,et al.Recombination suppression at the dominant Rhg1/Rfs2locus underlying soybean resistance to the cyst nematode[J].Theor Appl Genet,2012,124:1027-1039.
[58]王永军,盖钧镒,邢 邯,等.大豆抗感SCN基因的一个RAPD标记 [J].大豆科学,2000,19(4):93-98.
[59]Vuong T D,Sleper D A,Shannon J G,et al.Confirmation of quantitative trait loci for resistance to multiple-HG types of soybean cyst nematode(Heterodera glycines Ichinohe)[J].Euphytica,2011,181:101-113.
[60]Li Y,Smulders M J M,Chang R,et al.Genetic diversity and association mapping in a collection of selected Chinese soybean accessions based on SSR marker analysis[J].Conservation Genetics,2011,12:1145-1157.
[61]张文慧,陈庆山,杨庆凯,等.大豆灰斑病1号生理小种抗性基因的SSR标记分析 [J].大豆科学,2004,23(3):169-173.
[62]陈立君,郭 强,刘迎雪,等.大豆灰斑病1号生理小种抗性基因的SSR标记 [J].中国农学通报,2009,(9):43-46.
[63]邹继军,董 伟.大豆对灰斑病菌7号小种抗性的遗传分析及抗病基因的RAPD标记 [J].科学通报,1998,43(21):2302-2307.
[64]Mian M A R,Wang T,Phillips D V,et al.Molecular mapping of the Rcs3gene for resistance to frogeye leaf spot in soybean[J].Crop Science,1999,39:1687-1691.
[65]Jiang C L,Ding J J,Wen J Z,et al.Identification and mapping of the Cercospora sojinarace 15resistance gene in soybean[J].Acta Phytophylacica Sinica,2011,38:116-120.
[66]Bradley C A,Wood A,Zhang G R,et al.Genetic diversity of Cercospora sojina revealed by amplified fragment length polymorphism markers[J].Canadian Journal of Plant Pathology,2012,34:410-416.
[67]Ma Y,Wang D,Li H,et al.Fine mapping of the RSC14Qlocus for resistance to soybean mosaic virus in soybean[J].Euphytica,2011,181:127-135.
[68]王大刚,智海剑,张 磊.大豆抗大豆花叶病毒研究进展 [J].中国油料作物学报,2013,35(3):341-348.
[69]郑翠明,常汝镇,邱丽娟.大豆对SMV3号株系的抗性遗传分析及抗病基因的RAPD标记研究 [J].中国农业科学,2001,34(1):1-4.
[70]Zheng C M,Chang R Z,Qiu L J,et al.Identification and characterization of a RAPD/SCAR marker linked to a resistance gene for soybean mosaic virus in soybean[J].Euphytica,2003,132:199-210.
[71]Yu Y G,Maroof M A S,Buss G R,et al.RFLP and microsatellite mapping of a gene for soybean mosaic virus resistance[J].Phytopathology,1994,84:60-64.
[72]Jeong S C,Kristipati S,Hayes A J,et al.Genetic and sequence analysis of marker slightly linked to the soybean mosaic virus resistance gene,Rsv3[J].Crop Science,2002,42:265-270.
[73]Hayes A J,Ma G,Buss G R,et al.Molecular marker mapping of Rsv4,agene conferring resistance to all known strains of soy bean mosaic virus[J].Crop Science,2000,40:1434-1437.
[74]阳小凤,杨永庆,郑桂杰,等.大豆对大豆花叶病毒株系SC6和SC17抗病基因的精细定位 [J].作物学报,2013,39(2):216?221.
[75]丁俊杰.大豆细菌斑点病抗性鉴定的研究概况 [J].黑龙江农业科学,2013,(1):132-133.
[76]马淑梅.大豆种质资源对灰斑病抗性鉴定评价 [J].植物遗传资源学报,2011,12(5):820-824.
[77]龙华英.大豆种质资源抗锈鉴定 [J].农业工程,2012,2 (9):70-74.
[78]朱英波,史凤玉,李建英,等.抗大豆胞囊线虫病野生大豆种质资源的初步筛选 [J].大豆科学,2011,3(6):959-963.
[79]孔祥超,李红梅,耿 甜,等.大豆种质资源对大豆胞囊线虫3号和4号生理小虫的鉴定 [J].植物保护,2012,38(1):146-150.
[80]Concibido V,Vallee B L,Mclaird P,et al.Introgression of a quantitative trait locus for yield fromGlycine sojainto commercial soybean cultivars[J].Theor Appl Genet,2003,106:575-582.
[81]Kim K,Diers B,Hyten D,et al.Identification of positive yield QTL alleles from exotic soybean germplasm in two backcross populations[J].Theor Appl Genet,2012,125:1353-1369.
[82]Bhusal S J,Jiang G L,Tilmon K J,et al.Identification of soybean aphid resistance in early maturing genotypes of soybean[J].Crop science,2013,53:491-499.
[83]Tuyen D D,Zhang H M,Xu D H.Validation and high-resolution mapping of a major quantitative trait locus for alkaline salt tolerance in soybean using residual heterozygous line[J].Mol Breeding,2013,31:79-86.
[84]段红梅,王文秀,常汝镇,等.大豆SSR标记辅助遗传背景选择的效果分析 [J].植物遗传资源学报,2003,4(1):36-42.
[85]Khanh T D,Anh T Q,Buu B C,et al.Applying molecular breeding to improve soybean rust resistance in vietnamese elite soybean[J].American Journal of Plant Sciences,2013,4:1-6.
[86]李文滨,赵 雪.2009年大豆分子标记及辅助选择育种研究进展 [J].东北农业大学学报,2010,41(1):139-148.
[87]郭丹丹,陈海峰,杨中路,等.大豆对SMV SC-3株系的抗性遗传和QTL分析 [J].大豆科学,2012,31(4):511-516.
