基因芯片技术在常见肠道致病菌感染检测中的应用
2013-08-05戈海泽梁慧敏卢晋英刘树业
戈海泽,梁慧敏,卢晋英,刘树业
(1天津市第三中心医院,天津300170;2天津医科大学)
肠道致病菌是临床上引起以腹泻、呕吐为主要症状的首要病因之一,也是危害食品安全的主要因素。近年来由于天然食物源生产的全球化、新食品生产工艺的应用、人们饮食习惯的改变以及卫生环境恶化等各种因素在一定程度上降低了食品的安全性,增加了肠道致病菌感染的几率[1]。据世界卫生组织的统计报告,发达国家死于食物中毒的儿童中,70.0%是由微生物性食物中毒所致,而肠道致病菌是引起食源性疾病中最常见的生物致病因素之一[2~5]。因此,能够及时准确地检出腹泻病原菌,对于早期发现传染源、控制疾病流行及有效治疗腹泻病患者有非常重要意义。传统的细菌培养是目前细菌感染的诊断金标准,但是存在着培养周期长,不同的细菌对营养条件和培养环境的要求都不相同,很难在同一条件下鉴定出不同种属的细菌。本实验研究时间为2010年10~12日,我们利用细菌种属特异性基因、重要毒力基因以及目前已经可区分菌株亚型的基因作为靶基因探针制备基因芯片,检测常见肠道致病菌,为疾病预防控制和临床快速诊断提供一种新的技术手段。
1 材料与方法
1.1 实验材料 标准菌株选取引起肠道感染的常见肠道致病菌,均为真空冷冻干燥保存的细菌(来自中国医学菌种保藏管理中心),按照常规方法进行复苏和形态学鉴定,包括金黄色葡萄球菌、志贺菌、副溶血弧菌、大肠埃希菌 O157∶H7、伤寒沙门菌、小肠结肠炎耶尔森菌、单核细胞增生李斯特菌、蜡状芽孢杆菌、空肠弯曲菌、艰难梭菌。
1.2 主要试剂及仪器 VITEK-Jr全自动微生物鉴定系统,美国安玛西亚Lucidea Spotter基因芯片点样仪;MDS公司GENEPIX 4400A基因芯片扫描仪,美国Bio-Rad公司PTC200型PCR扩增仪,麦康凯培养基,汉克培基,庆大霉素—亚碲酸钾平板由天津市金章医用新技术研究所提供;DNA抽提试剂盒、链霉亲和生物素碱性磷酸酶,PCR试剂盒购自上海普洛麦格公司。
1.3 方法
1.3.1 引物、探针的设计与合成及基因组DNA扩增 从GenBank获取这些细菌16S rDNA、23S rDNA序列,利用上述病原菌的特异性基因、重要毒力基因以及目前区分菌株亚型的基因,设计并合成引物,寡核苷酸探针设计在每重病原菌基因组两对引物之间的可变区,在5'端标记氨基基团,反转录标记cDNA探针并纯化,Cy3-dUTP标记16S rDNA,Cy5-dUTP标记23S rDNA;挑取鉴定的单个菌落,研磨、振荡、混匀于100 μL裂解液震荡混匀,置沸水浴10 min,再经13 000 r/min离心10 min,取上清液作PCR模板,-20℃保存备用;基因组DNA扩增应用50 μL PCR反应体系,扩增条件为:94℃预变性5 min,94℃变性30 s、60℃退火30 s、72℃延伸30 s进行40个循环,最后72℃延伸5 min,反应结束后,2%琼脂糖凝胶上电泳检测PCR产物,为使PCR达到最高的扩增效率和灵敏度,对反应体系中上、下游引物的用量及比例,反应液成分及反应条件进行优化。
1.3.2 临床标本采集与DNA扩增 收集2008~2011年就诊于天津市第三中心医院肠道科100份腹泻患者粪便标本,平行采集两份,一份用于基因芯片检测,另一份用于常规培养鉴定。
1.3.3 基因芯片的制备和杂交 将制备的探针用点样液(6×SSC,0.1%SDS)稀释至终浓度为50 μmol/L,各取6 μL置于384孔板,用微阵列点样仪点到醛基化玻片上,每点体积约为0.5 nL,直径约200 μm,间距500 μm,重复2 次;同时设立不含寡核苷酸片段点样液的空白对照,点样时保持相对湿度为70%、温度为25℃。将点样后的芯片于室温放置至少20 h。使用时用0.2%的SDS洗去芯片上一些未共价结合的寡核苷酸,然后用双蒸水冲掉残余的SDS,自然晾干,待用。将PCR产物于PCR扩增仪上94℃变性5 min,迅速置于0℃的冰上5 min;取变性过的PCR扩增液1 μL与9 μL杂交液混匀,将10 μL混合液转移至芯片的杂交区域。将芯片置于杂交盒中,50℃水浴保温1 h。将杂交后的芯片依次于室温下在洗液A(1×SSC,0.2%SDS)、洗液B(0.2×SSC)和洗液C(0.1×SSC)中各洗涤1 min。用洗液进行洗膜3次后,加入POD作用10 min,然后加入TMB避光显色5 min。将洗涤完的芯片置于荧光扫描仪中扫描。
1.3.4 统计学方法 采用SPSS13.0软件进行统计分析,两组率的比较采用χ2检验。以P≤0.05为有统计学差异。
2 结果
2.1 PCR扩增与条件优化 分别对引物之间的浓度、模板量、退火温度和镁离子浓度进行了优化。同时,分别对目的基因上、下游引物比例进行了优化。PCR产物电泳检测结果见图1。
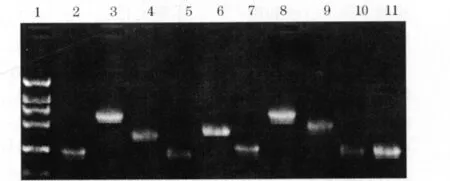
图1 PCR产物琼脂糖凝胶电泳图
2.2 基因芯片杂交检测结果 每株标准菌的基因组DNA进行PCR扩增后与膜芯片进行杂交结果见封三图1;蓝色斑点仅出现在其对应的位置,在其他位置未出现,表明该方法能够准确检测出这些肠道致病菌。
2.3 临床标本检测结果 采用基因芯片技术与细菌培养对100份粪便标本进行平行检测,基因芯片共检出50份(50%)携带肠道致病菌,其中检出金黄色葡萄球菌7株、志贺菌7株、副溶血弧菌17株、大肠埃希菌O157∶H72株、伤寒沙门菌2株、小肠结肠炎耶尔森菌4株、单核细胞增生李斯特菌3株、蜡状芽孢杆菌2株、空肠弯曲菌4株、艰难梭菌2株;细菌培养鉴定出47份(47%)携带肠道致病菌,其中金黄色葡萄球菌8株、志贺菌6株、副溶血弧菌16株、大肠埃希菌O157∶H73株、伤寒沙门菌2株、小肠结肠炎耶尔森菌3株、单核细胞增生李斯特菌2株、蜡状芽孢杆菌2株、空肠弯曲菌3株、艰难梭菌2株,其中42例基因芯片检测与细菌培养结果一致,准确率为79.25%,两种方法比较差异无统计学意义(χ2=5.28,P >0.05)。
3 讨论
基因芯片技术是近年发展起来的分子生物学技术平台,以其快速、准确、高通量、自动化等特点被广泛应用于生物学的各个领域,如细菌分子流行病学调查、细菌基因鉴定、基因突变及多态性分析、基因表达谱分析、DNA 测序等[6~8]。不同肠道致病菌感染的主要临床症状均是恶心、呕吐和腹泻,临床症状难以区分各种肠道致病菌,因此,鉴定肠道致病菌主要依赖临床实验室检测。尽管细菌培养是病原菌诊断鉴定的金标准,但是在相同条件下很难同时培养多种肠道致病菌,而且培养至少需要3~5 d才能确定致病菌类型,不仅耗时而且很难实现高通量平行检测多种肠道致病菌[9~11]。本研究依据细菌DNA生物信息学信息,以基因芯片为技术平台,成功实现了对细菌样本及临床标本的预处理、PCR扩增和基因芯片杂交等研究。我们针对细菌16S rDNA、23S rDNA序列建立的基因芯片检测系统,可以快速准确地鉴定临床常见的十余种肠道致病菌,整个检测过程只需要2~3 h,较传统的细菌培养检测技术明显缩短了检测时间,对于症状危急的感染者,可以做到及早诊断、对症用药。
基因芯片技术的特异性是本方法的关键,与所选取的靶基因和筛选的引物、探针密切相关。由于本实验所检测靶细菌中有很多属于同一种属内细菌,具有相近的遗传关系,因此在选择靶基因时需要严格筛选,要求相对应的引物与探针具有很高的特异性[12]。经过对PCR反应条件的优化以及大量寡核苷酸探针的筛选,我们将实验中的探针和引物在GenBank进行BLAST比对,每条引物和探针序列均具有很高的特异性,保证了检测方法的可靠性。基因芯片检测的灵敏度也是该鉴定方法的重要评价指标。本实验所建立的基因芯片检测技术对于每种靶细菌的灵敏度均可以达到103CFU/mL,而实验中的PCR电泳检测的灵敏度只有达到105CFU/mL,明显低于基因芯片技术,与目前报道的其他一些基因芯片检测方法的灵敏度一致[13,14]。我们采用基因芯片技术与细菌培养对100份粪便标本进行平行检测,两种方法比较差异无统计学意义。两种检测方法的差异可能与以下几方面原因有关:①基因芯片检测是在PCR的基础上进行的,死亡或破碎的细菌也同样可以扩增;②细菌数量不足103CFU/mL,并含有较多的杂菌;③革兰阳性细菌破壁较难,基因组DNA没有完全释放出来,所以PCR扩增会产生阴性结果;④细菌培养需要在很多杂菌中挑选出致病菌的克隆,在细菌量少的情况下就很难进行分离纯化[14,15]。
临床腹泻患者的快速检测与诊断是目前研究的一个热点,随着基因芯片技术应用范围的不断扩大,使其快速、高通量检测得以实现。针对目前肠道致病菌基因芯片检测的现状与不足,后续研究还需要在临床样本的基因组DNA提取方面做一些实验,以便使对于临床样本的处理更科学、合理、有效。我们下一步将增加探针的数目,争取涵盖更多的病原菌,不断扩大实验菌株的检测范围,并进一步评价芯片系统的特异性和敏感性。虽然目前基因芯片技术成本昂贵,检测的可靠性、实用性还有待于进一步验证,但基因芯片技术快速、高通量等优势将为肠道致病菌的复杂、多样性提供更为广阔的应用前景。
[1]蔡炯.食源性疾病的现状与防制[J].中国卫生检验杂志,2005,15(9):1150-1152.
[2]Koehler KM,Lasky T,Fein SB,et al.Population based incidence of infection with selected bacterial enteric pathogens in children younger than five years of age,1996-1998 [J].Pediatr Infect Dis J,2006,25(2):129-134.
[3]Hyytia-Trees EK,Cooper K,Ribot EM,et al.Recent developments and future prospects in subtyping of foodborne bacterial pathogens[J].Future Microbiol,2007,2(2):175-185.
[4]Mittal R,Bulgheresi S,Emami C,et al.Enterobacter sakazakii targets DC-SIGN to induce immunosuppressive responses in dendritic cells by modulating MAPKs [J].J Immunol,2009,183(10):6588-6599.
[5]Ashida H,Ogawa M,Mimuro H,et al.Shigella infection of intestional epithelium and circumvention of the host innate defense system[J].Curr Top Microbiol Immunol,2009,337:231-255.
[6]金大智,文思远,王升启.基因芯片技术在检测肠道致病菌方面的应用[J].微生物学报,2006,46(2):500.
[7]Niewold TA,Kerstens HH,van der Meulen J,et al.Development of a porcine small intestional cDNA micro-array:characterization and functional analysis of the response to enterotoxigenic E.coli[J].Vet Immunol Immunopathol,2005,105(3-4):317-329.
[8] Chang DE,Smalley DJ,Tucker DL,et al.Carbon nutrition of Escherichia coli in the mouse intestine[J].Proc Natl Acad Sci U S A,2004,101(19):7427-7432.
[9]Ramaswamy V,Cresence VM,Rejitha JS,et al.Listerial review of epidemiology and pathogenesis [J].J Microbiol Immunol Infect,2007,40(1):4-13.
[10]Schild S,Tamayo R,Nelson EJ,et al.Genes induced late in infection increase fitness of Vibrio cholerae after release into the environment[J].Cell Host Microbe,2007,2(4):264-277.
[11] Tao P,Zheng W,Wang Y,et al.Sensitive HPV Genotyping Based on the Flow-Through Hybridization and gene chip[J].J Biomed Biotechnol,2012,2012:938780.
[12]Jin DZ,Wen SY,Chen SH,et al.DNA microarrays assay for simultaneous detection and identification of multiple commonintestinal pathogens[J].Mol Cellular Probes,2006,(20):337-347.
[13]Call DR,Brockman FJ,Chandler DP.Detecting and genotyping Escherichia coli O157:H7 using multiplexed PCR and nucleic acid microarrays[J].Int J Food Microbiol,2001,67(1-2):71-80.
[14]Hagens S,Loessner MJ.Application of bacteriophages for detection and control of foodborne pathogens[J].Appl Microbiol Biotechnol,2007,76(3):513-519.
[15]Ballif M,Hii J,Marfurt J,et al.Monitoring of malaria parasite resistance to chloroquine and sulphadoxine-pyrimethamine in the Solomon Islands by DNA microarray technology[J].Malar J,2010,(9):270.
