微生物单宁酶研究现状
2013-07-23朱继新杨民和
朱继新,杨民和
(福建师范大学生命科学学院,福建福州 350108)
单宁酶,即单宁酰基水解酶(EC3.1.1.20),是一种具有水解催化能力的生物催化剂,可催化水解水合单宁如单宁酸中的酯键、缩酚键和糖苷键,水解产物为葡萄糖和五倍子酸,如图1所示[1]。单宁酶的主要作用底物是单宁类物质,主要由蛋白类、淀粉类、纤维素类等大分子物质复合组成,单宁被认为是自然界第二大多酚类物质。传统的单宁被分为浓缩单宁和水解单宁,目前公认的是分为没食子酸单宁、逆没食子酸单宁、浓缩单宁和复合单宁等4大类。单宁的生物降解主要通过微生物降解和酶降解,最有效的方法是利用生物转化把大分子单宁降解成为小分子化合物,但各种微生物对单宁的降解方式不径相同[2-5]。
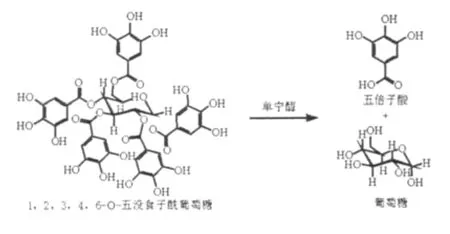
图1 单宁酶水解1,2,3,4,6-O-五没食子酰葡萄糖Fig.1 Hydrolysis of 1,2,3,4,6-pentagalloylglucose catalyzed by tannase
单宁酶是一种诱导酶,能由某些微生物在单宁酸等诱导物存在时诱导合成,它的水解能力和合成能力均需要在适合的环境中进行。单宁酶有着广泛的工业应用,例如饮料、食品、化妆品、医药、制革、饲料等方面。美国和日本等国家还申请了一些相关专利,美国食品与药品监督管理局(FDA)已经明确了单宁酶是安全可靠的产品[6],随着单宁酶的应用越来越广泛深入,也为单宁酶的开发和生产提供了强大的动力,在商业中越来越重要。然而,单宁酶的大规模生产存在2个限制因素,包括较高的生产成本和相关酶学性质研究的缺失[5]。近年来,微生物单宁酶越来越受到研究者的青睐,其商业化应用也越来越广泛。
1 单宁酶的来源
单宁酶存在于动物、植物及微生物中,目前主要依靠微生物在单宁酸等诱导物存在情况下发酵产生,国内外已报道的微生物有细菌、酵母和真菌等。这些微生物主要从各种植物的叶片、表皮、根等部位分离纯化获得,或者是来源于富含单宁的土壤或工业废水中。目前产单宁酶的微生物主要是真菌类的曲霉属和青霉属。表1总结了近几十年来国内外学者对产单宁酶菌株来源的相关研究[5]。
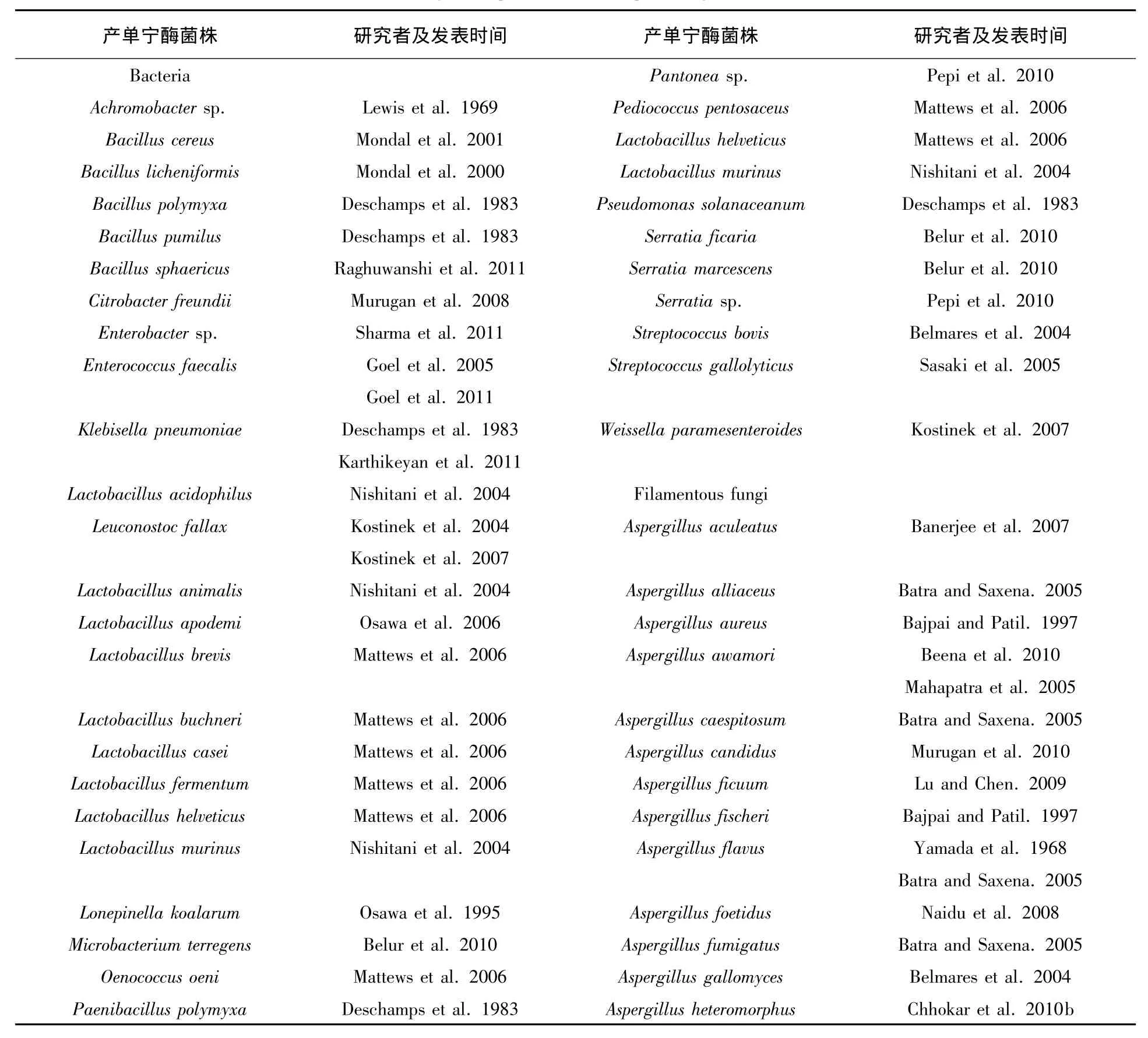
表1 产单宁酶菌株及其来源Table1 The origin and production of the producing strains of tannase
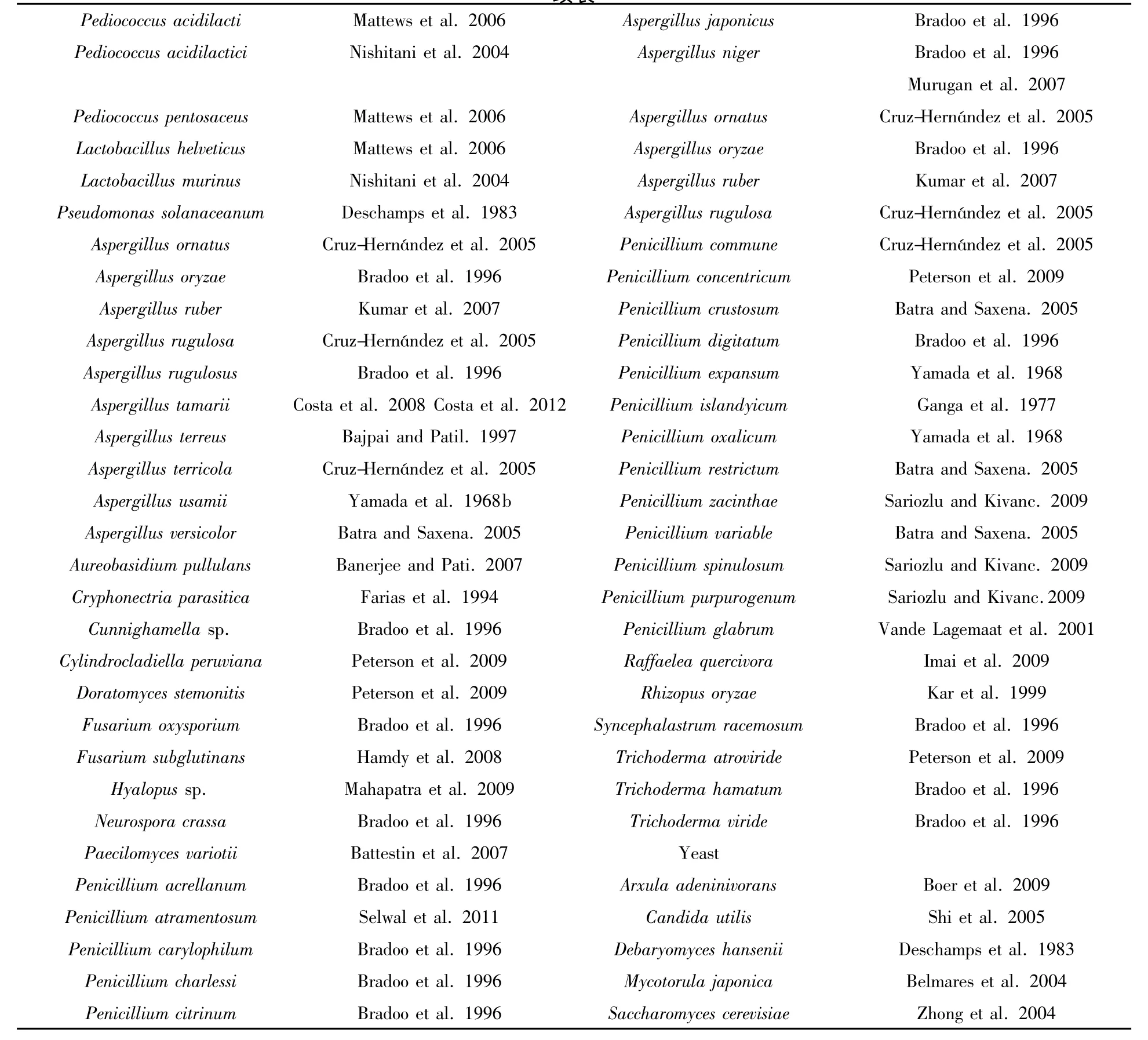
续表
由表1可知,产单宁酶的微生物以丝状真菌和细菌较多,酵母菌较少,这些工作为今后产单宁酶菌株的选择提供了参考。
2 单宁酶的生产
单宁酶的生产由菌株、诱导底物、发酵方式和发酵条件等4个条件决定。最早报道单宁酶仅仅是在单宁酸诱导下产生的,其终产物为没食子酸和葡萄糖。其后发现五倍子粉、没食子酸丙酯、没食子酸等也可作为单宁酶的诱导物。单宁酶的生产可采用固体发酵和液体发酵2种发酵方式,这2种方式可产生不同类型的单宁酶,分别是胞外酶和胞内酶。多年以来,工业生产单宁酶都是以液体发酵为主。近些年,利用固体发酵生产单宁酶也有许多的报道。利用微生物固体发酵生产单宁酶有以下几个特点:①是天然的胞外酶;②酶的产率较高;③酶活较高;④单宁酶的pH和温度稳定性较好。
早在1994年Lekha和Lonsane就对固体发酵、液体深层发酵、液体表层发酵产单宁酶做了比较,发现固体发酵产单宁酶酶活分别是其他2种液体发酵形式的2.5倍和4.8倍[7]。Cruz-Herandez等[8]利用黑曲霉GH1菌株,分别采用固体发酵和液体深层发酵方式,比较在不同温度下产单宁酶的情况。结果表明,固体发酵比液体深层发酵产单宁酶酶活力高4倍多,达到2 291U/L。Renovato等[9]对黑曲霉分别进行固体发酵和液体发酵,其产生的单宁酶在结构、稳定性、催化性能和等电点等方面有显著的差异,固体发酵比液体发酵产单宁酶的酶活高5.5倍;固体发酵和液体发酵产生的单宁酶在最适温度、最适pH、分子量大小和酶的糖基化水平等方面均有不同。这是第1次报道固体发酵和液体发酵产生单宁酶结构不同,为2种发酵方式产生的单宁酶有不同的稳定性和催化活性提供了依据。由上所述,固体发酵产单宁酶酶活力普遍比液体发酵要高,回收更加容易。由于发酵方式的不同,所产生单宁酶的性质和结构也有所差异。近些年来,以固体发酵方式生产单宁酶的研究逐年增多。
国内外有很多关于产单宁酶培养条件优化的报道。Bradoo等[10]发现用日本曲霉变种通过添加单一或复合糖类,当以单宁酸为唯一碳源进行液体摇瓶发酵时,单宁酶的产量提高2倍多。刘如石等[11]对黑曲霉No.3菌株产单宁酶液体发酵条件进行优化研究,使单宁酶比活力达到17.71 U/mg;同时还探索了菌丝生长与产酶的关系,发现黑曲霉No.3菌株产酶属于生长偶联型。Battestin等[12]利用响应面法对拟青霉产单宁酶固体发酵培养基优化,在培养温度为29~34℃、单宁酸添加量为8.5%~14%、稻壳∶麸皮比为50∶50的条件下,该最优培养条件发酵培养5 d后,单宁酶酶活提高了8.6倍。保玉心等[13]经紫外诱变筛选黑曲霉,以五倍子为诱导物进行固体发酵,使单宁酶酶活有很大的提高。在对培养基进行优化后,单宁酶酶活达到58.2 U/g。Costa等[14]分别以单宁酸、没食子酸和没食子酸甲酯为诱导物,进行黑曲霉液体发酵产单宁酶研究,结果表明以单宁酸为诱导物所产单宁酶酶活最高,达到 20.6 U/mL。Mnjit等[15]自制革厂排出的污水中分离筛选出产单宁酶菌株烟曲霉MA,首次以玫瑰果叶为固体基质,在25℃,pH 5.0,培养96 h酶活最高达174.32 U/g。培养基中添加单宁酸或没食子酸单宁等诱导物就可诱导微生物产生单宁酶。谷文等[16]从铁冬青植物组织中筛选分离得到高产单宁酶木霉属真菌,采用正交设计对培养基进行优化,发酵48 h后酶活力达到0.392 μmol/(min·mL)。
从上述研究看,影响单宁酶生产的主要培养条件是诱导物和固体基质的选择。目前这些菌株产单宁酶酶活力还不是很高,尚难于进行大规模产业化生产。各种微生物产单宁酶发酵条件的优化,将是今后进一步努力的方向。
3 单宁酶提取和纯化
酶的纯化是指将酶从细胞或含酶原料中分离出来,从而获得所要求的酶制品的过程,目的是为了提高其催化能力,增强其稳定性,防止不必要的反应。单宁酶是一种细胞结合酶,由于液体发酵产单宁酶主要是胞内酶,在单宁酶的分离纯化过程中微生物细胞的破壁相对比较困难,一般的破壁方法有超声波、石英研磨和渗透法等。这些方法所得单宁酶酶活损失较大,给后续纯化过程带来困难。常规酶的纯化步骤复杂、费时,而且酶活损失很大。纯化倍数、回收率和洗脱时间是纯化过程中典型的几个决定性因素。
王征等[17]采用40%~80%饱和度的硫酸铵分级沉淀,再经过透析浓缩,DEAE-纤维素阴离子交换层析,葡聚糖G-150凝胶层析,单宁酶的纯化倍数达到115。刘如石等[18]经过超滤浓缩、DEAE离子交换层析、Gephadex G-150凝胶层析和Sepharos-6B凝胶柱层析等四步纯化后,酶的回收率达到11%,纯化倍数达113.5。Ramirez-Coronel等[19]利用固体发酵生产胞外单宁酶,通过制备等电聚焦,FPLC阴离子交换层析和葡聚糖凝胶层析等步骤进行单宁酶纯化,SDS-PAGE后得到2种单体的单宁酶,分子量分别为90 ku和180 ku。Sharma等[20]利用两步快速纯化方法,超滤浓缩除盐,Sephadex-200凝胶层析后,纯化倍数135,回收率91%。Gaikai等[21]把离子表面活性剂用于反胶团对单宁酶的浓缩和纯化,因为单宁酶是高度吸水的糖蛋白,目前这种方法是Aspergillus allahabadi产单宁酶提取和纯化的最佳方法,纯化倍数12.7,回收率81.2%,单宁酶浓缩3倍;和常规的纯化方法相比,在回收率较低的情况下,却有很高的纯化倍数。在浓缩过程中表面活性剂、浓缩时间、pH、离子强度和有机溶剂比例都是主要的影响因素。由于产单宁酶菌株及培养方式的不同,导致所使用的纯化方法可能不尽相同,但总的目的是为了去除杂蛋白,提高酶的比活力,得到人们所需要的目的酶。
4 单宁酶酶学性质的相关研究
酶学性质和很多因素有关,如温度、pH值、有机溶剂和金属离子等。单宁酶的来源和培养条件是酶学性质不同的主要原因,例如来自酵母和真菌的单宁酶都是糖蛋白类,但是细菌单宁酶并没有翻译后修饰[9]。此外,已有的研究发现同种微生物利用不同的培养方式产生的单宁酶在糖基化上有显著性的差异。国内外学者对单宁酶酶学性质的研究见表2,酶学性质的研究为单宁酶的产业化打下了坚实的基础。
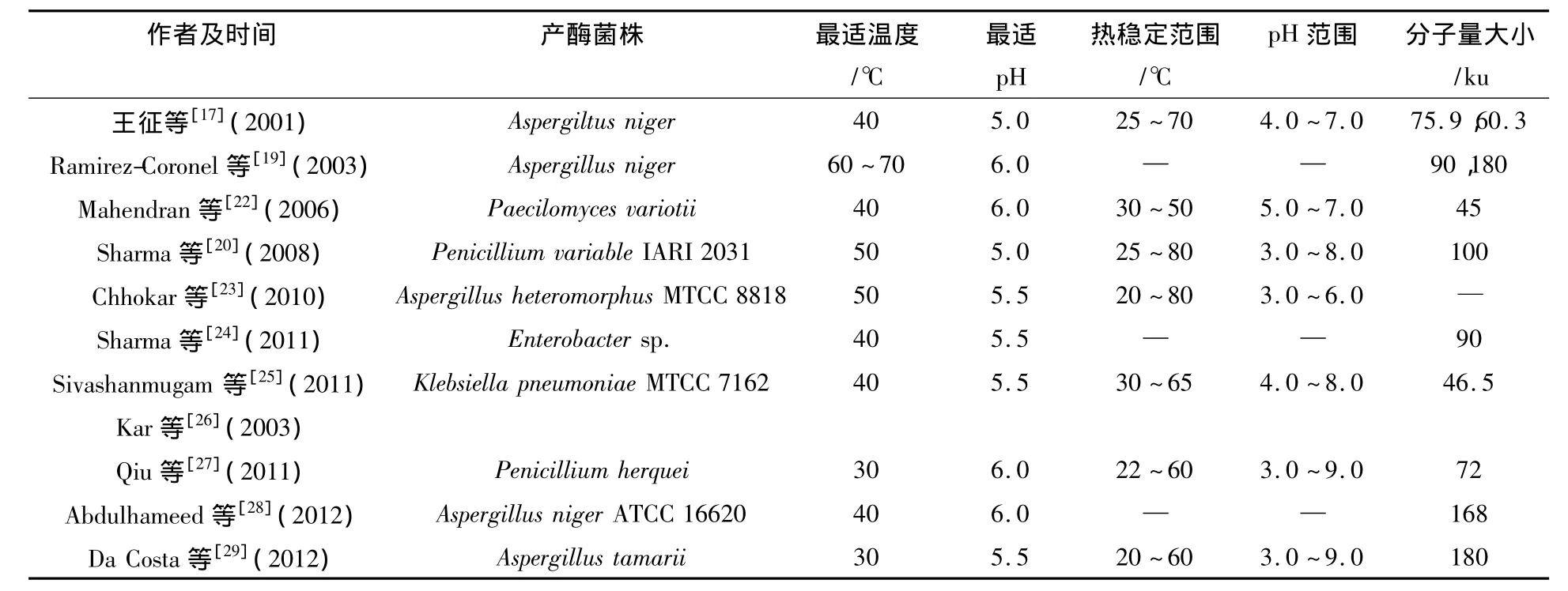
表2 单宁酶酶学性质Table2 Characterization of tannase
不同来源的单宁酶酶学性质有一定的差异,综合分析表2可知大多数菌株产单宁酶最适温度为30~50℃,Ramirez-Coronel等所研究的黑曲霉产单宁酶的最适反应温度是60~70℃,比其他研究者最适温度要高很多,初步说明此菌所产单宁酶耐热性很好。最适pH值为5.0~6.0,热稳定性范围在70~80℃以下,pH稳定范围在3.0~8.0。但是不同来源的单宁酶的分子量的大小差别很大,最小的为45 ku,最大达到180 ku,大多数在100 ku左右。
近些年研究金属离子、表面活性剂以及有机溶剂对单宁酶影响的相关报道也有很多。Chhokar等[23]研究表明 Ca2+、Fe2+、Cu2+、Cu1+对酶活有促进作用,然而Hg2+、Na+、K+、Zn2+、Ag+、Mg2+、Cd2+对酶活有抑制作用;异戊醇、甲醇、乙醇、苯、甲苯、丙三醇等有机溶剂对单宁酶有抑制作用,表面活性剂如吐温-80、吐温-20、EDTA也有抑制效果,但是SDS可增强单宁酶活力。Qiu等[27]从橡椀果子分离得到的郝克氏青霉菌产单宁酶,Fe3+、Zn2+、DTT、β-巯基乙醇、乙醇等对单宁酶有抑制作用,而K+、Mn2+、吐温-80、Triton-100等对其有促进作用。Kar等[26]研究金属离子对米根霉单宁酶的影响,发现Mg2+、Hg+对单宁酶活性有促进作用,但是Ba2+、Ca2+、Zn2+、Hg2+、Ag+有抑制作用,Fe3+和Co2+对单宁酶完全抑制。另外,Marco等[30]发现Fe3+对黑曲霉GH1单宁酶也有完全抑制作用,Cu2+和Zn2+抑制效果较小,Co2+有很强的促进作用。由上述研究可知,由于单宁酶的来源不同,蛋白质结构和功能存在差异,导致金属离子、表面活性剂以及有机溶剂对其的影响效果也不尽相同。
5 单宁酶分子生物学的相关研究
有关单宁酶分子生物学的相关研究近年来报道较少。Hatamoto等[31]早在1996年就对来自米曲霉的单宁酶基因进行了研究,发现该单宁酶基因没有内含子,可编码588个氨基酸序列,分子量为64 ku。把此单宁酶基因克隆到其他产单宁酶菌株,拷贝数和酶活力成正比。Albertse等[32]从黑曲霉克隆单宁酶基因,并在酿酒酵母中表达,重组体单宁酶与其他单宁酶的比较研究结果显示:重组体单宁酶表达量很低,可能在酵母菌中表现出过糖基化现象。Iwamoto等[33]第一次从除葡萄球菌之外的其他细菌—乳酸杆菌中克隆出单宁酶基因tanLpl。SDS-PAGE分析显示,纯化的tanLpl蛋白是个单体多肽,分子量约为50 ku。对于不同来源的单宁酶基因的差异和联系有待深入研究。
近些年,基因工程技术在产单宁酶微生物育种方面的应用也有报道,获得了一些遗传特性稳定、酶活力较高的高产菌株(表3)。
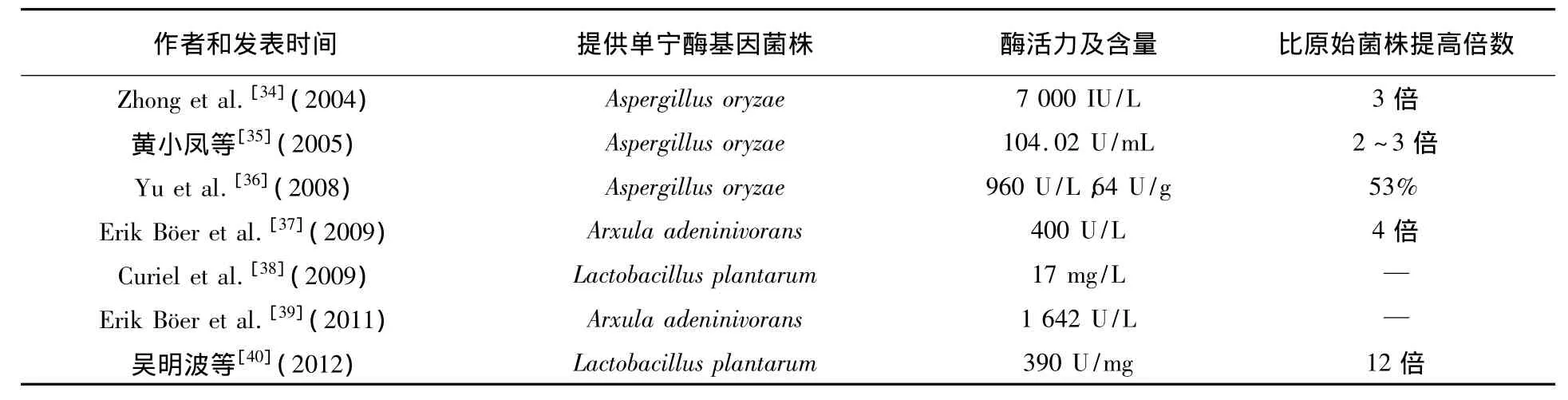
表3 单宁酶微生物育种相关研究Table3 The tannase microbial breeding research
由表3可看出,通过基因工程技术在单宁酶微生物育种方面有一定的进展,与原始菌株相比酶活力都有所提高,不同来源的产单宁酶菌株,其编码单宁酶的基因的特征也是有差异的。
6 总结和展望
近年来在单宁酶及其应用研究方面虽然有了一定的进展,但是受到生产、回收、纯化的高成本,以及生产工艺条件不成熟等因素的影响,单宁酶的生产和应用受到限制,因而还未能形成大规模的工业化生产。从表1可知,微生物单宁酶具有广泛的来源,进一步发掘微生物资源,开发微生物单宁酶,将为单宁酶的产业化创造条件。从已有的文献报道可知,固体发酵产单宁酶比液体发酵更有优势。但近年来由于新的生物反应器的出现,对2种发酵方式产单宁酶都有所提高,也为单宁酶的生产奠定基础。目前的趋势是应用分子生物学技术和工业发酵技术相结合,来提高单宁酶的产量和降低成本。需要做的是确立一个微生物表达载体,需要发展低成本的微生物发酵工艺,并提高单宁酶的固定化等。对单宁酶的蛋白结构和蛋白质工程方面的相关研究,也有助于增强单宁酶活力水平。单宁酶可以作用于某些特定的底物,根据不同需要研究单宁酶对这些底物的生物转化,可以为人类提供特殊的代谢产物。但是,单宁酶生物转化单宁的生化过程还不是很清楚,有待更深入的研究。
[1]Aguilar,C.and G.Gutirrez-Senchez.Review:sources,properties,applications and potential uses of tannin acyl hydrolase[J].Food Science and Technology International,2001,7(5):373-382.
[2]Aguilar,C.N.,et al.Production of tannase byAspergillus nigerAa-20 in submerged and solid-state fermentation:influence of glucose and tannic acid[J].Journal of industrial microbiology & biotechnology,2001,26(5):296-302.
[3]Aguilar,C.N.,et al.Microbial tannases:advances and perspectives[J].Appl Microbiol Biotechnol,2007,76(1):47-59.
[4]Bhat,T.K.,B.Singh,and O.P.Sharma.Microbial degradation of tanninsa current perspective[J].Biodegradation,1998,9(5):343-357.
[5]Chávez-González,M.,et al..Biotechnological Advances and Challenges of Tannase:An Overview[J].Food and Bioprocess Technology,2011,5(2):445-459.
[6]Barthomeuf,C.,F.Regerat,and H.Pourrat,Production,purification and characterization of a Tannase fromAspergillus nigerLCF 8[J].Journal of fermentation and bioengineering,1994,77(3):320-323.
[7]Lekha,P.K.and B.K.Lonsane.Comparative titres,location and properties of tannin acyl hydrolase produced byAspergillus nigerPKL 104 in solid-state,liquid surface an submerged fermentations[J].Process Biochemistry,1994,29(6):497-503.
[8]Cruz-Hernendez,M.,et al.Evaluation of culture conditions for tannase production byAspergillus nigerGH1[J].Food Technology and Biotechnology,2006,44(4):541-544.
[9]Renovato,J.,et al.Differential properties ofAspergillus nigertannase produced under solid-state and submerged fermentations[J].Appl Biochem Biotechnol,2011,165(1):382-395.
[10]Bradoo,S.,R.Gupta,and R.K.Saxena.Parametric optimization and biochemical regulation of extracellular tannase fromAspergillus japonicus[J].Process Biochemistry,1997,32(2):135-139.
[11]刘如石,谢达平,等.Asp.nigerNo.3液态发酵生产单宁酶条件的研究[J].食品科学,2001,22(2):28-32.
[12]Battestin,V.and G.A.Macedo.Tannase production byPaecilomyces variotii[J].Bioresour Technol,2007,98(9):1832-1837.
[13]保玉心,等.固态发酵黑曲霉产单宁酶发酵条件研究[J].食品与发酵工业,2008,34(6):54-56,61.
[14]Costa,A.M.,et al.Production of tannase byAspergillus tamariiin submerged cultures[J].Brazilian Archives of Biology and Technology,2008,51(2):399-404.
[15]Manjit,et al.Tannase production byAspergillus fumigatusMA under solid-state fermentation[J].World Journal of Microbiology and Biotechnology,2008,24(12):3023-3030.
[16]谷文,杨亦陈,王石发,等.产单宁酶真菌的筛选及产酶条件[J].南京林业大学学报(自然科学版),2010,34(03):6-10.
[17]王征,谢达平,谭周进,等.黑曲霉单宁酶提纯及其性质的研究[J].湖南农业大学学报:自然科学版,2001,27(1):60-62.
[18]刘如石,邱义兰,谢达平,等.Asp,NigerNo.3单宁酶纯化及性质的研究[J]食品与机械,2000,(4):15-16.
[19]Ramirez-Coronel,M.A.A novel tannase fromAspergillus nigerwith glucosidase activity[J].Microbiology,2003,149(10):2941-2946.
[20]Sharma,S.,L.Agarwal,and R.K.Saxena.Purification,immobilization and characterization of tannase fromPenicillium variable[J].Bioresour Technol,2008,99(7):2544-2551.
[21]Gaikaiwari,R.P.,S.A.Wagh,and B.D.Kulkarni.Extraction and purification of tannase by reverse micelle system[J].Separation and Purification Technology,2012,89:288-296.
[22]Mahendran,B.,N.Raman,and D.J.Kim.Purification and characterization of tannase fromPaecilomyces variotii:hydrolysis of tannic acid using immobilized tannase[J].Appl Microbiol Biotechnol,2006,70(4):444-450.
[23]Chhokar,V.,et al.Purification and characterization of extracellular tannin acyl hydrolase fromAspergillus heteromorphusMTCC 8818[J].Biotechnology and Bioprocess Engineering,2010,15(5):793-799.
[24]Sharma,K.P.and P.J.John.Purification and characterization of tannase and tannase gene fromEnterobactersp.[J].Process Biochemistry,2011,46(1):240-244.
[25]Sivashanmugam,K.and G.Jayaraman.Production and partial purification of extracellular tannase byKlebsiella pneumoniaeMTCC 7162 isolated from tannery effluent[J].African Journal of Biotechnology,2011,10(8):1364-1374.
[26]Kar,B.,R.Banerjee,and B.Bhattacharyya.Effect of additives on the behavioural properties of tannin acyl hydrolase[J].Process Biochemistry,2003,38(9):1285-1293.
[27]Qiu,Y.et al.Properties and secondary structure of tannase fromPenicillium herquei[J].Biotechnology and Bioprocess Engineering,2011,16(5):858-866.
[28]Abdolhassan,K.,A.Z.Mohammad,and J.Abbsali.Identification,isolation,cloning and sequencing of tannase gene fromAspergillus niger[J].Jundishapur Journal of Microbiology,2012,4(2(supl)):465-471.
[29]Da Costa,A.M.,et al.Production,purification and characterization of tannase fromAspergillus tamarii[J].African Journal of Biotechnology,2012,11(2):391-398.
[30]Marco,M.,et al.A novel tannase from the xerophilic fungusAspergillus nigerGH1[J].J Microbiol Biotechnol,2009,1:1-10.
[31]Hatamoto,O.,et al.Cloning and sequencing of the gene encoding tannase and a structural study of the tannase subunit fromAspergillus oryzae[J].Gene,1996,175(1):215-221.
[32]Albertse,E.H et al.Cloning,expression and characterization of tannase fromAspergillus species[D].South Africa:University of the Free State,2002.
[33]Iwamoto,K.,et al.Identification and cloning of a gene encoding tannase(tannin acylhydrolase)fromLactobacillus plantarumATCC 14917(T)[J].Syst Appl Microbiol,2008,31(4):269-277.
[34]Zhong,X.,et al.Secretion,purification,and characterization of a recombinantAspergillus oryzaetannase in Pichia pastoris[J].Protein expression and purification,2004,36(2):165-169.
[35]黄小凤,韦宇括,韦传东,等.单宁酶基因在黑曲霉st31中的克隆与表达[J].中国生物工程杂志,2005,25(9):74-77.
[36]Yu,X.W.and Y.Q.Li.Expression of Aspergillus oryzae tannase in Pichia pastoris and its application in the synthesis of propyl gallate in organic solvent[J].Food Technology and Biotechnology,2008,46(1):80-85.
[37]Boer,E.,et al.Atan1p-an extracellular tannase from the dimorphic yeast Arxula adeninivorans:molecular cloning of the ATAN1 gene and characterization of the recombinant enzyme[J].Yeast,2009,26(6):323-337.
[38]Antonio Curiel,J.,et al.Production and Physicochemical Properties of Recombinant Lactobacillus plantarum Tannase[J].Journal of Agricultural and Food Chemistry,2009,57(14):6224-6230.
[39]Boer,E.,et al.Large-scale production of tannase using the yeast Arxula adeninivorans[J].Appl Microbiol Biotechnol,2011,92(1):105-14.
[40]吴明波,彭小红,温华,等.乳酸杆菌中单宁酶基因的克隆、表达与纯化[J].四川大学学报(自然科学版),2012,49(2):479-483.
