基于OSVR的谷氨酸发酵过程建模
2013-07-07韩顺成李艳坡
韩顺成, 潘 丰, 李艳坡
(江南大学轻工过程先进控制教育部重点实验室,江苏无锡 214122)
谷氨酸[1]发酵过程具有较强的非线性和时变性,建立其机理模型较为困难,而复杂的机理模型也不易用于控制和优化。因此,建立一个精确有效的黑箱模型对谷氨酸发酵生产过程具有重要意义。
支持向量机(SVM)是基于结构风险最小原则和统计学理论的机器学习方法之一。SVM能较好地解决小样本、非线性、过学习、维数灾难和局部极小等问题,具有很强的泛化能力,目前已经广泛应用于模式识别、回归估计、概率密度估计等各个领域。M.Martin和J.S.Ma在Ger提出的支持向量机分类(Support Vector Machine Classification,SVC)在线算法[2]的基础上,提出了支持向量机回归(Support Vector Machine Regression,SVR)[3-4]在 线 算 法。OSVR(Online Support Vector Machine Regression)是一种先进的SVM训练方法,WANG等[5]运用标准的OSVR对谷氨酸发酵过程建模,实现了对于菌体质量浓度、基质质量浓度和产物质量浓度的在线建模。实验结果表现出良好的泛化能力并降低了模型的预测误差,优于基于神经网络(Neural Network)和SVM模型。但是研究发现在标准的OSVR算法中,由于核函数是非线性的,计算核函数需要大量的训练时间,增加了算法的计算量;并且核函数的数据并非在每一步中都被更新,且被更新的数据仅仅占据了一小部分。
因此,为了提高算法运算速度和预测精度,文中利用缓存保存核函数,将其核函数矩阵进行相应调整,对OSVR进行改进。文中应用改进的OSVR对谷氨酸进行模型建立,并在仿真软件Matlab上,与标准的OSVR进行比较。结果表明,基于改进的OSVR算法建立的谷氨酸动力学模型表现出良好的精确性,并提高了算法的在线学习速度。
1 改进的在线支持向量机回归
给出一训练集:T={(xi,yi),i=1,2,…,l},yi∈R和x∈RN,可以构造线性回归函数[6]:
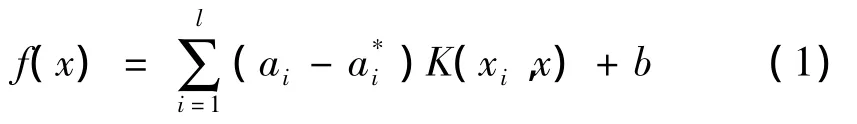
式中:l为训练集T中样本个数;ai,a为拉格朗日因子;K(xi,x)= φ(xi)·φ(x)为核函数;φ(xi)为非线性映射将数据xi映射到高维特征空间。
ai和b可以通过求解以下优化函数得到:
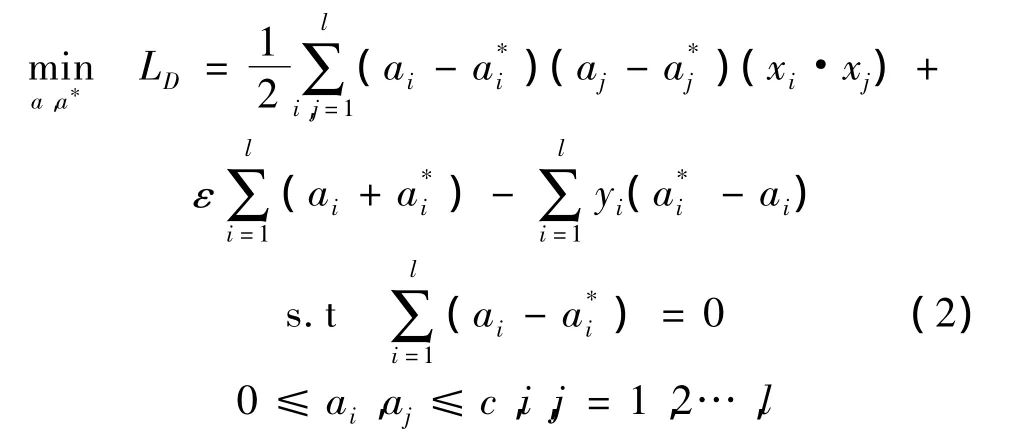
通过拉格朗日因子和 Karush-Kuhn-Tucke(KKT)条件[7](具体推导过程参见文献[4]),训练数据可以分为:
1)错误的支持向量集合E:
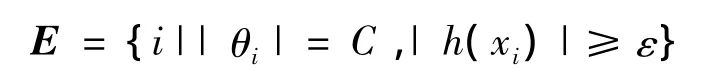
2)边缘支持向量集合S:
S={i|0 <|θi|< C,|h(xi)|= ε}
3)保留的支持向量集合R:
R={i||θi|=0,|h(xi)|≤ ε}
当增加一个新的样本xc至训练集T中,初始令θc=0,为使xc进入E,R,S其中一个集合中,并同时满足KKT条件,必须满足以下等式:
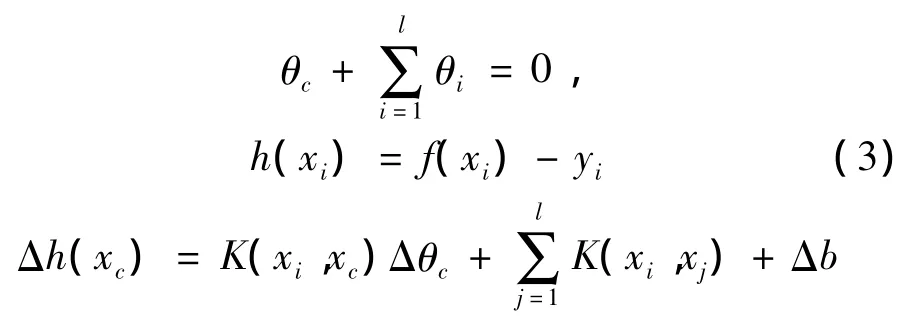
标准的OSVR算法[5]中,由于核函数是非线性的,计算核函数浪费了大量的训练时间,增加了算法的计算量;并且关于核函数的数据并非都被更新在每一步中,被更新的数据仅仅占据了一小部分。为了提高算法运算速度,用一个核矩阵K(xn,xG)来表示 K(xn,xE),K(xn,xR)和 K(xn,xS)。此时 K(xn,xG),K(xn,xc)可表示为

xGi∈ G,i=1,2,…,ln,ln是E ∪ R∪S∪c集合中样本的数目。G是E,S,R 3个集合中的其中一个。
当增加一个样本xc:

当移除一个样本xc:

在xc被增加或者被移除时,其余的某些样本可能被从原本的集合中更新至另外一个集合中,在每步更新过程中,一些样本会被从一个集合更新另一个集合中。当一样本xGli从集合G1更新至G2内时,有以下4种可能更新情况:
Case 1:G1=R,G2=S;Case 2:G1=S,G2=R;Case 3:G1=E,G2=S;Case 4:G1=S,G2=E。
在每一种情况下,矩阵 K(xn,xG1),K(xn,xG1i)和K(xn,xG2)的相应更新情况分别如下:
增量算法:当一个样品被增加至训练样本集,通过逐渐改变θc,直到xc进入R,S,E 3个子集的一个,这个过程中一些其他的样品也从G1更新至G2,通过计算和更新 K(xn,xG1),K(xn,xG1i),K(xn,xG2)来确保其他样本仍满足KKT条件。
减量算法:当一个样本的θc为零时,由于该样本不影响支持向量机模型;在式(4)的前提下,改变θc和h(xc),当xc样本被移除时,使得剩余其他样本仍继续满足KKT条件。
上述是通过核函数矩阵进行处理改进OSVR算法,其算法的有效性将在下面进行验证。
2 基于改进的OSVR谷氨酸模型
谷氨酸是一种重要的氨基酸物质,主要采用补料分批发酵方法生产。影响发酵过程的可在线测量变量主要有温度、通气流量、pH值、溶解氧质量浓度DO、补料量等,这些参数密切相关谷氨酸质量浓度。根据发酵机理的过程,文中选择温度xT(i)、pH值xpH(i)、溶氧质量浓度xDO(i)和尾气CO2排放量xCO2(i)作为输入变量的谷氨酸质量浓度软测量模型。温度、pH值、溶氧质量浓度和尾气CO2排放量在线数据通过相应传感器获取,谷氨酸的质量浓度由SBA-40B生物传感仪(山东省科学院生物研究所生产)测定。选取第i+1次谷氨酸质量浓度xG(i+1)作为输出预测量。
详细的动力学模型建立过程如下:
1)构建一个OSVR训练样本集X{Xi=(xTi,xpHi,xCO2i,xDOi,xGi)},总计有 l个样本。
2)当增加一个新样本xc,初始化θc=0,并计算 K(xn,xE),K(xn,xR),K(xn,xs),K(xn,xc);
3)利用增量算法对样本训练:(1)If|h(xc)|≤ε,xc→ R,并更新 K(xn,xR);(2)当一个样本被更新从G1→G2中时,通过式(8),(9),(10)计算出相应的K(xn,xG1),K(xn,xG1i),K(xn,xG2);
4)当训练样本数目溢出值max_sp=l,利用减量算法移除多余样本;
5)计算训练误差e=xG(i+1)-xG(i)(xG(i+1)表达式,参考公式(11),当e≤min_error,训练停止,否则继续3);
6)用该模型预测xG(i+1)。
OSVR算法一旦确定,就可以对变量进行预测,由式(1)可得基于支持向量回归机软测量的谷氨酸模型如下:
这里选用 RBF 核函数[8],即
支持向量机中σ,C,ε等参数的选取直接影响到训练结果。因此文中通过交叉算法经多次实验选取:σ =0.6,C=100,ε =0.1,此时训练效果为最佳,可确保建模具有优良的预测性能。均方误差
文中的数据来自某实验室提供由5 L发酵罐生产谷氨酸所获得的10批次正常发酵数据,每批数据表达一个完整的谷氨酸发酵过程。每批分为15个样品。7个批次数据作为训练数据,其余的作为测试数据。仿真计算机为Intel(R)Celeron(R)CPUE330,1 024 M内存,仿真软件为Malabr2010a。
可以得到谷氨酸质量浓度的预测结果,与实际测量值的比较如图1所示。
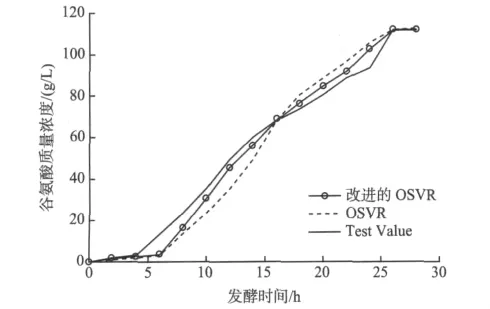
图1 基于改进的OSVR的谷氨酸质量浓度预测曲线Fig.1 Prediction curve based on the improved OSVR glutamate concentration
图1显示基于改进的OSVR的谷氨酸动力学模型与测试值比较表现出较好的拟合效果,尤其在发酵过程10 h后,优于OSVR。为了验证该模型具有较强的泛化能力,采用预测误差值来比较模型的精确度,预测误差值可以用相对误差的形式表示:

基于改进的OSVR相对误差如图2所示。
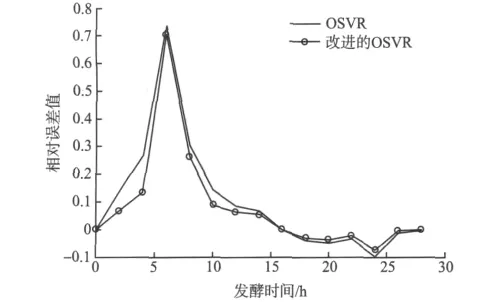
图2 基于改进的OSVR的相对误差Fig.2 Relative error based on of the improved OSVR
表1中的训练时间,改进的OSVR是3个方法之中速度最快的,且均方差MES最小。图1,2和表1,显示基于改进的OSVR谷氨酸动力学模型获得了良好的泛化能力,表现出最优的训练精度,其谷氨酸预测模型表现出良好的精确性,与此同时提高了算法的训练速度,取得了改进算法的预期效果。以上显示谷氨酸产物质量浓度获得了良好的预测精度,在保证良好精度的前提下,并提高了建模过程的运行速度,对谷氨酸实际生产具有一定参考价值。
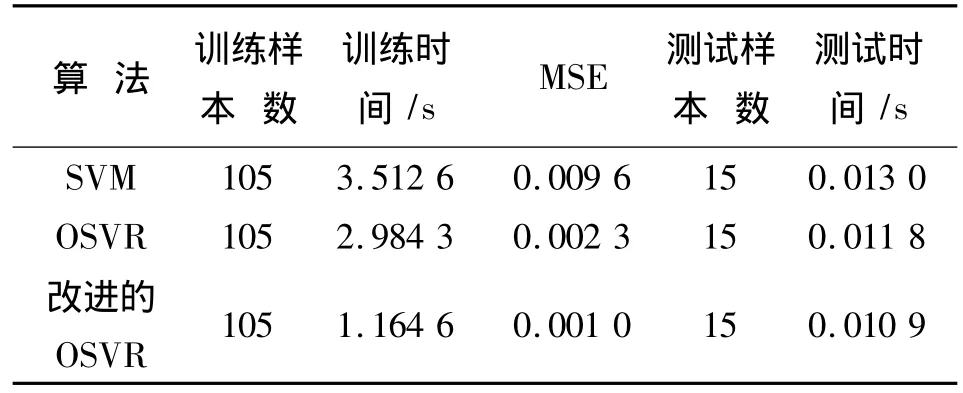
表1 训练时间、测试时间和MSE比较Tab.1 Comparison of training time, testing time and MSE
3 结语
近年来随着生物技术工业迅速发展,生物发酵过程建模已获得了高度的关注,由于缺乏可靠的传感器用于过程变量的在线检测等原因,其自动化水平与其他工业生产过程相比还较低。因此,一个精确的在线模型对减少发酵原材料和动力消耗、提高产物生产量具有重要意义。
微生物发酵具有高度的非线性和时变性,其内在机理复杂。在线支持向量机是一种较好的估计预测手段。基于在线支持向量机的模型具有较高的精度,该模型泛化能力强,动态性能好,能较好地描述实际对象的特性,该模型可用于发酵过程中状态变量的估算与预测。
[1]Kinoshita S,Udaka S.Shimono studies on the amino acid fermentation-Part I.Production of L-glutamic acid by various microorganisms[J].Journal of General and Applied Microbiology,1957,3:193-205.
[2] Gert Cauwenberghs,Tomaso Poggio.Incremental and decremental support vector machine learning[J].In Advances Neural Information Processing Systems(NIPS’2000)[J/OL].http://books.nips.cc/papers/files/nips13/CauwenberghsPoggio.pdf
[3]Martin M.Online support vector machines for function approximation,Technical Report LSI-02-11-R[R].Software Department,Universitat Politecnica de Catalunya,Spain,2002.
[4]MA J S,James T,SimonP.Accurate on-line support vector regression[J].Neural Compuing,2003,15(11):2683-2704.
[5]WANG X,DU Z,PAN F.Dynamic modeling of biotechnical process based on online support vector machine[J].Journal of Computers,2009,4(3):251-258.
[6]邓乃扬,田英杰.数据挖掘中的新方法-支持向量机[M].北京:科学出版社,2004.
[7]Takahashi N,Guo J,Nishi T.Global convergence of SMO algorithm for support vector rgression[J].IEEE Trans on Neural Networks,2008,19(6):971-982.
[8]Smola A J,Scholkopf B.Learning with Kernels[M].Cambridge,MA:MIT Press,2001.
