蛋白质-RNA相互作用界面预测与设计
2012-12-11黄阳玉阳秀凤李昊田纪晓峰程洪礼赵蕴杰郭大川刘士勇
黄阳玉 阳秀凤 李昊田 纪晓峰 程洪礼 赵蕴杰郭大川 李 林 刘士勇
(华中科技大学物理学院生物分子物理与模建小组,武汉430074)
蛋白质-RNA相互作用界面预测与设计
黄阳玉 阳秀凤 李昊田 纪晓峰 程洪礼 赵蕴杰郭大川 李 林 刘士勇*
(华中科技大学物理学院生物分子物理与模建小组,武汉430074)
蛋白质-RNA之间的相互作用是蛋白质在细胞里面行使功能的重要方式之一.结构生物学家利用实验手段可以得到蛋白质-RNA复合物的三维结构,通过原子水平的晶体结构来解释蛋白质与RNA的识别过程.但实验取得蛋白质-RNA的复合物结构非常困难,耗钱、耗时,同时受限于其相互作用强度.因而利用理论的方法对蛋白质-RNA相互作用界面进行预测与设计在生物医学研究中十分重要.本文主要综述了近期蛋白质-RNA相互作用界面预测与设计方面的进展,包括以下几个方面:(1)蛋白质-RNA分子对接算法以及对接前后存在的构象变化的处理;(2)蛋白质-RNA识别机制的研究;(3)基于蛋白质-RNA相互作用界面的分子设计.蛋白质-RNA分子对接算法逐步完善将有助于我们对大量未知功能的蛋白质与RNA进行功能注释,而基于生物大分子相互作用界面的分子设计将在药物设计领域中有广阔的应用前景.
蛋白质-RNA相互作用;分子对接;界面设计;复合物结构预测
1 引言
蛋白质是细胞中的主要功能分子之一.蛋白质功能主要是通过特异性的以不同的亲和力与其他各类分子(比如蛋白质分子、RNA、DNA等)结合形成复合物来实现.随着RNA的催化功能被发现,诺贝尔化学奖得主沃特·吉尔伯特1提出地球上早期的生命系统中可能存在“RNA世界”,之后才出现DNA.RNA分子同时拥有存储遗传信息以及催化功能.与蛋白质类似,RNA在溶液中可以折叠成特定的空间结构.RNA分子可以结合蛋白质形成比较稳定的复合物(比如核糖体)或者瞬时的复合物,这些复合物在基因表达、调控等过程发挥着重要的作用.高分辨率的复合物晶体结构是在原子水平深入理解这些重要生物学过程的基础.美国科学家Venkatraman Ramakrishnan、Thomas A.Steitz和以色列女科学家Ada E.Yonath因对核糖体的结构和功能的研究分享了2009年诺贝尔化学奖.尽管取得了相当大的进展,但是目前可以得到的RNA-蛋白质复合物结构数目还是相当有限的.因此,从理论方面进行蛋白质-RNA复合物结构预测与界面分子设计是非常重要的基本科学问题.本文将简明扼要介绍蛋白质-RNA复合物结构预测、基于蛋白质-RNA相互作用界面的分子设计两个方面的内容.
2 蛋白质-RNA相互作用
人类基因组测序完成后人们发现,人类基因组里编码蛋白质的基因大约不超过30000个,比某些植物还少.最近大量实验结果表明基因组中的非编码序列绝大部分可以表达成非编码RNA.人们发现这些非编码RNA(比如microRNA,piRNA,siRNA, g-RNA等)参与了许多重要的生物学过程.2-5除了通过RNA-RNA相互作用6来实现非编码RNA的功能之外,RNA与蛋白质特异性识别形成RNA-蛋白质复合物在基因调控、mRNA降解和翻译、RNA剪切、RNA代谢、RNA成熟与定位、RNA降解调控等生命过程中也起了关键性作用7,8(见表1).现在已经发现了大量的非编码RNA与蛋白质的相互作用,并收集整理成数据库.9,10很多结合RNA的蛋白质(RNA-binding proteins,简称RBP)由少量的结合RNA的结构域(RNA-binding domain)构成.11常见的结合RNA的结构域12-14有RRM(RNA recognition motif)、KH(K-homology)、dsRBM(double-stranded RNA-binding motif)、PAZ(取名于三个主要的Ago蛋白质:Piwi, Ago和Zwille)、PIWI、Pumilio、锌指等.这些结构域通过氢键、静电、堆积相互作用等方式结合各种各样的RNA.与这些蛋白质结合的RNA有单链、双链(详细情况见表115-28).结合双链的dsRBM蛋白质一般有60-70个氨基酸长度,采用αβββα折叠模式,而双链RNA采用A-form的构象,比较刚性,在与蛋白质结合前后构象变化不大.29蛋白质结合RNA有时主要通过静电相互作用进行识别,这样的结合没有序列特异性(比如MRP1/MRP2与g-RNA形成复合物30).
然而要深入理解和进一步认识RNA-蛋白质相互作用所发挥的生物学功能,需要知道它们的单体结构和复合物的结构.对于RNA单体结构预测,目前主流的FARFAR,31iFoldRNA,32RNA2D3D33和MC-Fold/MC-Sym34等方法对于较小或者拓扑结构较为简单的RNA小分子(<50 nt(nucleotide,核苷酸)可以给出精度较高的结构(均方根偏差(RMSD)为0.4 nm左右),然而对于分子较大或者拓扑结构较为复杂的RNA分子则很难给出较为精确的结果.2011年,Zhao等35开发的方法对于小RNA双螺旋和发卡精度可以达到0.28 nm,对于较为复杂的RNA分子精度为0.58 nm.这些RNA结构预测工具为基于序列的RNA功能研究与分子设计打下良好的基础.由于RNA结构复杂性,目前实验得到的RNA-蛋白复合物结构的数目远远少于蛋白质-蛋白质复合物.因此,从理论上预测RNA与蛋白质复合物结构非常必要.

表1 RNA-蛋白质相互作用结构类型与功能Table 1 Structural type and function of RNA-protein interaction
2.1 蛋白质-RNA相互作用的理论与实验研究
最近,文献中涌现出大量使用计算方法研究RNA与蛋白质相互作用的理论工作,包括构建RNA-蛋白质相互作用数据库,10,36,37模拟RNA与蛋白质相互结合的机制,38分析RNA-蛋白质相互作用界面性质,39-43从序列或者结构来预测RNA结合蛋白质(RNA-binding protein)、44RNA与蛋白质共同进化45以及结合位点,46-52模建RNA与蛋白质复合物结构.53-55然而,相对比较成熟的蛋白质-蛋白质复合物结构预测方法,56-61目前还没有一个完整的RNA-蛋白质复合物结构预测方法.文献中报道的工作主要是利用蛋白质-蛋白质复合物结构预测程序产生对接构象,并用RNA-蛋白质打分函数进行挑选.62-64蛋白-蛋白复合物结构预测分为两个主要步骤:第一,产生候选构象;第二,用打分函数对候选构象进行打分排序,挑选近天然的解.产生候选构象的算法主要有基于刚体的对接算法(比如基于表面立方格子和表面法向量匹配算法,65基于快速傅里叶变换(FFT)刚体对接算法56,57,60,61,66,67和基于蒙特卡罗的刚体对接算法58,68)和考虑柔性的对接算法.59,69-73相对蛋白质-蛋白质分子对接而言,RNA-蛋白质分子对接的研究还很少,目前仍然处于初始阶段.RNA-蛋白质复合物结构预测研究的这种状况,一方面是因为近年来人们才重视非编码RNA的作用,另一方面是RNA本身的复杂性,主要表现在如下几个方面.
首先,对RNA-蛋白质识别机制的认识是建立RNA-蛋白质分子对接算法的基础上,但是这方面目前理论和实验上的研究都还非常少.相对而言,对蛋白质-蛋白质以及蛋白质-DNA相互作用的机制理论74-76和实验77-81上都有比较多的研究,可以在RNA-蛋白质识别机制研究中借鉴.例如Sanchez等79利用突变实验研究了E2C-DNA相互识别机制,他们给出了蛋白质与DNA结合途径:初始扩散,然后形成一些有非天然接触的过渡态系综,接着形成了一个动力学陷阱,最后非天然接触缓慢重排并转变成近天然的相互作用.Tang等77用顺磁弛豫增强实验技术证实蛋白质-蛋白质在相互识别过程中达到平衡的条件下存在瞬时的、非特异性的构象系综,一旦由微弱的非特异性静电相互作用形成非特异性的偶遇复合物(encounter complex),一个蛋白质就可以在另一个蛋白质的表面进行二维搜索,最后掉入一个由互补的范德华相互作用和静电相互作用决定的狭窄的能量漏斗中.Tang等77以及后来的Kim等76的研究都表明蛋白质与蛋白质在形成特异性与非特异性的相互作用中长程静电相互作用起到非常重要的作用.另一方面,Fawzi等81利用顺磁弛豫增强实验技术与同位素标记手段研究了酶I的N端结构域(EIN)与含组氨酸的磷酸化载体蛋白质(HPr)的结合机制,结果发现两个蛋白质在结合位点附近形成偶遇复合物,而这类复合物只需要少许转动与平动就能变成天然复合物,此外,还观察到HPr在结合位点背面与天然复合物形成三体偶遇复合物,这类复合物在EIN蛋白质的活性位点被占据的情况下,提供了较高的HPr浓度.这些实验证据表明,蛋白质的结合过程是先远距离静电识别调整方向靠近,之后再由其它相互作用完成对接过程.我们最近提出了一套理论方法研究这个问题.对benchmark 4.082中的170个蛋白质-蛋白质复合物进行基于长程静电相互作用的结构采样,发现这种方向预调整过程发生的概率(14.1%)要高于随机概率(2.8%),而且这种基于长程静电相互作用的采样算法同样可以预测蛋白质-蛋白质相互作用的位点,准确性与目前最好的界面预测算法之一PINUP83相当.
其次,对于RNA-蛋白质相互作用体系,RNA分子主链上的每个磷酸基团都带有一个电子单位的负电荷,所以RNA分子主链带有很强的负电荷.已经有一些研究分析了RNA-蛋白质相互作用界面的残基出现的偏好性.39,41,43,46,47,49,51Pérez-Cano和Fernandez-Recio51提到统计分析的结果严重依赖数据集的大小,因此,我们仅对数据集中复合物数目大于100的研究46,51进行了综合分析.结果表明最偏好出现在界面的氨基酸是精氨酸(R),赖氨酸(K),组氨酸(H),其次是酪氨酸(Y),色氨酸(W),最不喜欢出现的是天冬氨酸(D),谷氨酸(E),半胱氨酸(C),缬氨酸(V),亮氨酸(L),异亮氨酸(I).从氨基酸的性质来看,R、K是带正电的,而H在pH值小于6时带正电,D、E带负电,C、V、L、I都是疏水氨基酸,H、Y、W具有芳香环.这些结果表明静电相互作用在RNA和蛋白质识别中应该发挥了重要作用,另外,H、Y、W的芳香环可能与碱基形成π-π堆积作用,40在近距离调整时发挥作用.因此,RNA和蛋白质可能通过长程静电吸引相互作用预先调整好相互作用方向,进而互相靠近,通过构象调整表面扩散完成结合过程.我们的初步研究表明,RNA和蛋白质确实可以通过静电相互作用在远距离识别.84但我们计算长程静电相互作用能量的方法还需要更加精确,需要考虑到溶液效应等.
由上面分析看见,蛋白质与RNA之间的相互作用类型主要有四种:静电相互作用(包含盐桥)、堆积相互作用(stacking interaction)、范德华相互作用(包括立体组装(steric packing)、氢键).这些相互作用除了可以用经典的分子力场项来描述之外,还可以用统计热力学(statistical thermodynamics)方法来进行描述.统计热力学是从物质的原子结构出发,统计出在晶体结构数据库中残基-残基(或者原子-原子)相互作用对出现的概率,通过Boltzmann定律导出残基-残基(或者原子-原子)之间的相互作用能,其概率π(x)与能量E(x)存在如下关系:85

这里的k和T分别为波尔兹曼常数和绝对温度,x为某一微观状态,依据选取的统计对象不一样,代表的对象不一样,可以为某残基-残基对,某原子-原子对等.配分函数Z(a)定义如下:

一般而言,Z(a)不太容易计算,可以通过选取某一参考态(E*(x))来计算有效的能量函数:
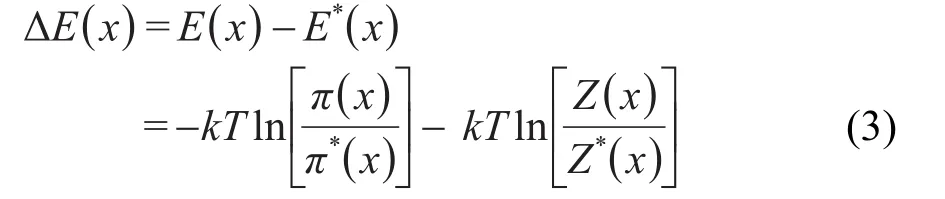
这里π(x)是微观状态x在参考态出现的概率,E*(x)是微观状态x对应的能量.对于给定的蛋白质或者蛋白质-RNA复合物体系而言,Z(a)和Z*(a)是一个常数,与x无关.如果我们假定就得到比较常用的形式:
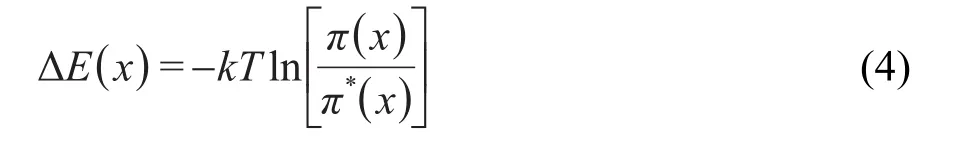
如果考虑微观状态x与残基类型(i,j),以及残基类型i和残基类型j之间的距离d之间的关系,上述方程可以写为:
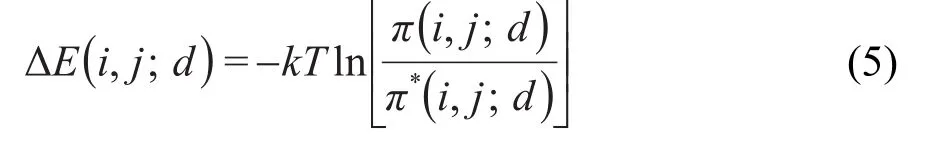
可见,相互作用能ΔE(i,j;d)主要由参考态的选取来决定,不同的参考态导致不同的相互作用能量函数.纯粹的基于距离的统计势还没有考虑到两对相同类型的残基对在距离相同时的取向问题,在基于距离的统计势中考虑残基相互作用对的方向性86,87显得比较重要.这实际上是微观态(统计对象)选取的方法问题,有时选取残基尺度的微观态,有时选取原子尺度的,有时是粗粒化模型,88有时选取氢键给体受体,53,89-92这里不再赘述.从热力学观点来看,蛋白质与RNA分子之间的识别和相互作用是一个热力学平衡过程,其形成的稳定蛋白质-RNA复合物构象是结合自由能最低的构象.在溶液环境下面,蛋白质与RNA的结合自由能ΔG可以估计为:

严格按照物理化学基本原理,计算蛋白质与RNA结合的ΔG由于计算量太大在目前的计算条件下不太可行,因此常常用简化的方法来计算结合自由能,这些简化的自由能就称之为打分函数.93传统的打分函数包括几何互补项,界面大小,范德华与静电相互作用,堆积密度,94统计势等等.本文讨论的打分函数主要是以统计势为主.
2.2 蛋白质-RNA相互作用打分函数构建
在蛋白质-RNA复合物结构预测中,打分函数构建是关键.2011年,基于ATTRACT程序,95Setny等63开发了一个粗粒化的蛋白质-RNA打分函数,对于7个自由态对接(unbound docking)体系,仅有1个例子的近天然解通过打分排名后在前100之内.同年,Tuszynska等64发表了两个基于知识的打分函数,在用GRAMM程序生成的RNA-protein对接候选构象中进行挑选,结果发现8个体系中4个可以找到近天然的复合物结构.同年,Li等62开发了一个基于残基的丰度势,他们发现RNA的二级结构状态对打分函数性能影响较大.总的来说,目前的蛋白质-RNA打分函数的性能还比较差,有较大的提升空间.为了构建有效的蛋白质-RNA相互作用打分函数,可以借鉴在蛋白质-蛋白质分子对接中的构建方法.在蛋白质-蛋白质分子对接中,打分函数主要有基于知识的打分函数,96-98基于经典分子力场优化的打分函数,58或者二者的组合.99单纯基于物理的力场(physical-based forcefield)在现阶段比基于知识(knowledge-based potential)的打分函数的性能要差,以后可能会更精确.86
对于基于知识的打分函数而言,如何定义参考态是关键,目前主要有三类方法定义参考态.第一类就是基于较大距离进行截断(比如DFIRE100)和体积校正,第二类就是随机混合残基或者原子类型(比如KBP101),第三类就是基于错误的构象或者decoys (decoys就是用计算机生成的蛋白质构象,少部分构象离天然构象比较接近,称为近天然构象(nearnative decoys),大部分都不是近天然构象)来定义(比如RAPDF,102PIPER67和DARS103).在基于知识的打分函数DECK104中,我们选择了蛋白质-蛋白质对接的decoy来定义参考态.考虑长程的残基-残基相互作用在蛋白质-蛋白质识别中是相当重要,我们通过考虑残基类型所处的二级结构状态来定义残基类型,从而间接考虑了残基-残基的长程相互作用.为了测试这点是否有效,我们把DECK与当时文献中最好的打分函数DCOMPLEX,105RosettaDock58和 ZRANK99进行了详细测试比较.在RosettaDock的蛋白质-蛋白质对接decoys中测试结果表明DECK比其它几个要好.104我们把原子溶剂化参数模型与FFT算法相结合,开发了全新的蛋白质-蛋白质分子对接程序ASPDock,60获得了比几何打分更好的效果.ASPDock与打分函数DECK连用,参加CAPRI (Critical Assessment of Prediction of Interactions,蛋白相互作用预测技术评估大赛,每次给定两个相互作用蛋白质在结合前的结构,预测其复合物结构)的对接比赛,准确预测了T40和T41(见图1106,107).在CAPRI打分预测比赛中,DECK准确地预测了T32, T40,T41,T50,T53.而王存新小组开发的HPNC-score93,108,109也准确地预测了T35,T37,T40,T41.93
比较好的打分函数主要由基于知识的势和基于经典分子力场项(范德华相互作用,静电相互作用等)进行线性拟合得到.58,110,111缺点是基于知识的势与经典分子力场项的混杂组合没有明确的物理意义,很难进一步改进.为了弥补这一缺陷,可以利用基于统计势的方法对基于分子立场项的参数进行确定(fluctuation matching),这样得到的打分函数的每项具有物理意义,而参数具有统计意义,目前在蛋白质结合前后构象变化研究112,113与蛋白质-RNA相互作用力场63中取得重要进展.该方法可能对蛋白质-蛋白质、蛋白质-RNA相互作用研究有帮助.另一个可能提高打分函数精度的方面是在传统的基于距离的统计势中考虑残基相互作用对的方向性.86,87
2.3 蛋白质-RNA分子对接中的构象变化
RNA-蛋白质分子对接构象采样遇到了很大的困难,主要原因是RNA分子存在很大的柔性.RNA与蛋白质对接前后,蛋白质构象变化不大,114RNA在结合前后构象变化较大(例如转录因子NF-κB二聚体体系.RNA在结合前后的RMSD为0.54 nm51). RNA-蛋白质在识别过程中存在的构象变化,并由此造成对接或者打分函数挑选近天然构象失败.蛋白质在结合配体过程中的构象变化可以分解为一些正则运动模式.70,112,115-119利用这些相关的正则运动模式的线性组合来生成新的受体构象,也许可以实现蛋白质-RNA的柔性分子对接.
2.4 蛋白质-RNA分子对接与蛋白质-蛋白质分子对接的区别与联系
RNA与蛋白质相互作用界面特征和蛋白质与蛋白质相互作用界面有很大的不同,不能把蛋白质-蛋白质对接方法直接用于RNA-蛋白质复合物结构预测.Chen等53用Rosetta程序包从5个RNA-蛋白质天然复合物出发进行微扰对接产生候选构象.基于距离的氢键相互作用势53和基于原子的统计势55都可以很好区分天然复合物与候选构象,但不能有效地从候选构象中挑选近天然的对接构象,而这一点在实际预测问题中更重要.此外,基于天然复合物的微扰对接与具有实际意义的分子对接还有很大距离.2010年,Perez-Cano和Fernandez-Recio51利用蛋白质-蛋白质对接FTDOCK程序66对RNA-蛋白质进行了分子对接的研究,利用基于统计的残基-核苷酸丰度势作为打分函数对分子对接结果进行挑选,并用于蛋白质相互作用预测技术评估大赛CAPRI的RNA-蛋白质体系.51,120这个结合丰度势与FTDOCK对接算法的方法在12个RNA-蛋白质的体系上进行了测试,结果表明FTDOCK能够在7个系统中产生近天然的复合物结构(RMSD<1 nm),而用基于丰度的势进行打分排序以后,仅有2个系统的近天然的解排名在10以内.这个结果说明了目前RNA-蛋白质对接方法还有很大局限性,直接把蛋白质-蛋白质对接方法FTDOCK应用到RNA-蛋白质体系存在一些问题,另外,基于统计的残基-核苷酸丰度势具有一定的挑选能力,但其有效性还不令人满意,需要进一步提高.这些结果表明几何互补性在蛋白质与RNA或蛋白质与蛋白质相互识别中都起到很重要的作用,但是其具体参数具有显著性差异.
基于以上分析,我们对蛋白质-蛋白质界面与RNA-蛋白质界面进行了比较,试图找寻二者之间的异同点以及原因.这样,我们可以对蛋白质-蛋白质对接方法进行改进,使之适应RNA-蛋白质相互作用系统.相比蛋白质-蛋白质复合物界面,RNA-蛋白质复合物界面有着明显的不同,即便在界面原子堆积上,也有着不同之处.我们选取我们自己挑选的80个复合物的数据集作为蛋白-RNA复合物的代表,而蛋白质分子对接数据集82作为蛋白质-蛋白质复合物的代表,来分析这两者之间的界面原子堆积的不同.分析结果表明RNA-蛋白质界面的几何互补特征与蛋白质-蛋白质界面的几何互补特征有显著不同.根据界面特性,我们初步建立了一个新的RNA-蛋白质对接算法RPDock,包括了基于FFT的对接和基于残基距离的粗粒化打分.在对接中,一方面,针对RNA-蛋白质界面和蛋白质-蛋白质界面的不同,我们优化了几何参数,使之更适于RNA-蛋白质对接;另一方面,RNA-蛋白质复合物形成过程中静电的作用非常重要,我们在对接中考虑了静电项.在粗粒化打分中,根据氨基酸残基的大小,侧链偶极矩和不同的二级结构类型,我们将氨基酸残基分为21类;根据核苷酸的类型和二级结构,我们将核苷酸分为8类.我们在Perez-Cano等121提出的包含有81个RNA-蛋白质复合物体系的测试集中进行了测试.结果表明,RPDock预测1000个构象的对接成功率是66%,而FTDock和GRAMM的成功率分别是56%和54%.通过基于残基距离的粗粒化打分后,RPDock预测1个构象的成功率达到16%,预测10个构象的成功率达到32%.可见,虽然对接的成功率比较高,但是打分函数的性能还不够好.
为了构建有效的打分函数,近期我们建立了一个蛋白质与RNA反应的结合常数的数据集(http:// biophy.hust.edu.cn/PRD/protein-RNA.html),收集了46个非冗余的蛋白-RNA复合物的平衡解离常数.在这个数据集中,除了给出了结合常数外,我们还给出了各个结合常数的测定方法与条件,并且由结合常数推出了在反应过程中的吉布斯自由能的变化值.这些数据可以用于蛋白质-RNA对接打分函数的构建.在我们的结合常数数据集文章投稿过程中,两个小组发表了2个非冗余的蛋白质-RNA分子对接测试数据集,121,122这两个数据集为大家评估蛋白质-RNA分子对接方法提供了方便.
2.5 蛋白质-RNA分子对接的解决方案
综上所述,RNA-蛋白分子结合机制与复合物结构预测研究中存在的主要问题以及解决方案有三点:其一,RNA与蛋白质在长程静电相互作用的约束下,RNA-蛋白质对接方向和对接界面不清楚.而我们在蛋白质-蛋白质相互作用体系里面开发了快速有效的研究长程静电相互作用的方法,可以用在这里给出RNA-蛋白质对接方向和对接界面;
其二,如何考虑RNA-蛋白质相互作用存在的构象变化?在分子对接形成初步的复合物构象时,我们可以对其进行分子动力学模拟,观察其构象变化.
其三,如何利用机制研究的结果改进RNA-蛋白质分子对接算法?机制研究可以得到大致的对接方向和位点信息,可以把这些信息整合到我们已有的对接算法RPDock中.以上三点的具体细节我们将在以后的研究中逐步展开.
3 基于蛋白质-RNA相互作用界面的分子设计
10多年前,Hermann123总结了一些基于RNA-蛋白质复合物的药物设计策略.受限于对RNA-蛋白质相互作用的理解,这个领域发展缓慢.2011年, Mackay等124提出既然识别双螺旋DNA的蛋白质已经商业化,那么显然就可以考虑一个类似的问题:能否设计一个蛋白质可以特异性识别RNA尾端序列?最近,两篇对蛋白质-RNA相互作用进行设计的文章125,126回答了这一重要问题并引起广泛的注意,Chen和Varani127专门为此写了评论.两个小组使用类似的酵母三杂交系统的实验方案找到了PUF蛋白质(Pumilio and FBF[fem-3 binding factor])的突变体(见图2),该突变体可以特异性地识别胞嘧啶,而在此之前发现的PUF蛋白质以及突变体都不能识别胞嘧啶.128这一发现使得研究人员可以人工设计出能够结合任意给定的RNA序列的PUF突变体.该新技术为人们研究生物学问题提供了一个很好的机会,比如Cooke等129马上利用该技术应用于FBF-2/GLD-2融合蛋白的翻译调控.PUF突变体与RNA之间的详细研究表明,氢键相互作用、范德华相互作用与堆积相互作用128,130决定了二者之间特异性识别,而大量的相关实验数据(比如蛋白质-RNA复合物晶体结构与结合常数)将快速推动蛋白质-RNA相互作用理论方法的发展.目前实现的例子是蛋白质与单链短片段RNA(线性片段)之间的识别,而且是纯粹的基于实验的方法.如果要实现蛋白质与双链RNA或者折叠后的RNA之间的特异性识别与设计,纯粹的基于实验的方法将面临组合爆炸困难,必然要借助于蛋白质-RNA分子对接工具或者蛋白质-蛋白质界面设计的策略来进行.下面简单介绍一种蛋白质-蛋白质界面设计的策略.

图2 设计出能够结合任意给定的RNA序列的PUF突变体Fig.2 Designed PUF mutant which can bind any given RNAsequence(a)interaction of wild-type PUF(NYxxQ)with U3 RNA(PDB 3BSB);(b)interaction of PUF-R6(SYxxR)with C3 RNA(PDB 2YJY).RNAbase and side chain of the key residues are shown as stick models.Hydrogen bonds are shown as dashed lines.After double mutation,the author126got a mutant PUF-R6,which can bind cytosine,however,the native PUF can not bind cytosine.The picture was plotted by pymol(http://www.pymol.org/).
蛋白质-蛋白质相互作用是蛋白质在细胞内发挥生物学功能的重要形式,因而是一个重要的药物设计全新靶标,131-135并受到越来越多的关注.促红细胞生成素(EPO)通过和它的受体(EPOR)相互作用,促进红细胞的分化和成熟.EPO已广泛应用于临床上各种贫血的治疗.但是用于生产重组EPO的哺乳动物细胞的培养成本很高,同时表达量不高,因而使用EPO治疗是相当昂贵的.利用已建立的一套“蛋白质关键残基嫁接”算法,Lai课题组136设计了具有EPO活性的功能蛋白质.生物测活实验表明设计的功能蛋白质PLC-δ1 PH结构域突变体具有拮抗EPO生物活性的功能,体外的平衡解离常数(Kd)为20 nmol·L-1,细胞实验得到的IC50为5.7 μmol·L-1.该PH蛋白质进行突变后与EPO只有几个功能残基一样,却具有了和EPOR结合的功能,而这种功能在自然界中是不存在的.这个例子验证了蛋白的疏水核心只起骨架的作用,功能由其局部表面决定的观点.这是首个将序列上非连续的配体-受体相互作用区嫁接到非同源的骨架蛋白质上的成功的例子.在2011年,华盛顿大学的计算生物学者Baker和他的同事在Science上发表文章,报道了他们通过蛋白质-蛋白质分子对接方法与热点残基库(hotspot library)相结合设计出来的蛋白HB36和HB80,能够结合到广泛流行性病毒1918 H1N1蛋白血凝素(HA).137随同发表在Science上的评论性的文章里, Der和Kuhlman138写道:“This strategy is reminiscent of a previous approach that involved grafting key residues from a known interaction onto a new protein scaffold to generate a new binding pair.”这说明Baker小组采用了和来鲁华课题组已发表的工作(嫁接关键残基)136比较类似的策略.最近,Zhang和Lai139将这一嫁接策略用于酶的活性位点设计.在不久的将来,类似的蛋白质嫁接策略可能会在其它生物功能体系中被成功实现.更复杂精细的策略可能需要在设计中考虑对蛋白质结合与解离的动力学过程有影响的因素.140显然,这一策略还可以用于蛋白质-RNA相互作用界面的分子设计.
4 小结
本文总结了蛋白质-RNA分子对接与设计.利用基于知识的统计势(打分函数)与多尺度的粗粒化模型(构象变化)对蛋白质与RNA的相互作用机制进行分析,有助于我们构建比较好的蛋白质-RNA相互作用计算方法.其次,把“蛋白质关键残基嫁接”算法应用到蛋白质-RNA相互作用体系,可能设计出特定功能的蛋白质或者具有特定功能的RNA分子.这类策略将在以蛋白质-RNA相互作用界面作为“药物靶标”的研究中具有应用前景.
(1) Gilbert,W.Nature 1986,319,618.
(2) Zhang,W.;Ye,K.Q.Chinese Bulletin of Life Sciences 2010,22, 608. [张 炜,叶克穷.生命科学,2010,22,608.]
(3) Zhao,S.;Liu,M.F.Chinese Bulletin of Life Sciences 2010,22, 623. [赵 爽,刘默芳.生命科学,2010,22,623.]
(4) Shao,W.;Fan,Y.J.;Xu,Y.Z.Chinese Bulletin of Life Sciences 2010,22,711.[邵 伟,樊玉杰,徐永镇.生命科学,2010,22, 711.]
(5) Chen,R.S.Sci.China Life Sci.2010,22,594. [陈润生.生命科学,2010,22,594.]
(6) Zheng,L.L.;Qu,L.H.Chinese Bulletin of Life Sciences 2010, 40,294. [郑凌伶,屈良鹄.中国科学生命科学,2010,40, 294.]
(7) Liu,M.F.;Wang,E.D.Chinese Bulletin of Life Sciences 2008, 20,178. [刘默芳,王恩多.生命科学,2008,20,178.]
(8) Licatalosi,D.D.;Mele,A.;Fak,J.J.;Ule,J.;Kayikci,M.;Chi, S.W.;Clark,T.A.;Schweitzer,A.C.;Blume,J.E.;Wang,X.; Darnell,J.C.;Darnell,R.B.Nature 2008,456,464.doi: 10.1038/nature07488
(9)Yang,J.H.;Li,J.H.;Shao,P.;Zhou,H.;Chen,Y.Q.;Qu,L.H. Nucleic Acids Research 2011,39,D202.
(10) Wu,T.;Wang,J.;Liu,C.;Zhang,Y.;Shi,B.;Zhu,X.;Zhang, Z.;Skogerbo,G.;Chen,L.;Lu,H.;Zhao,Y.;Chen,R.Nucleic Acids Research 2006,34,D150.
(11) Lunde,B.M.;Moore,C.;Varani,G.Nature Reviews.Molecular Cell Biology 2007,8,479.doi:10.1038/nrm2178
(12) Clery,A.;Blatter,M.;Allain,F.H.Current Opinion in Structural Biology 2008,18,290.doi:10.1016/j.sbi.2008.04.002
(13) Maris,C.;Dominguez,C.;Allain,F.H.The FEBS Journal 2005,272,2118.doi:10.1111/j.1742-4658.2005.04653.x
(14) Chen,Y.;Varani,G.The FEBS Journal 2005,272,2088.doi: 10.1111/ejb.2005.272.issue-9
(15)Antson,A.A.;Dodson,E.J.;Dodson,G.;Greaves,R.B.;Chen, X.;Gollnick,P.Nature 1999,401,235.doi:10.1038/45730
(16) Frazao,C.;McVey,C.E.;Amblar,M.;Barbas,A.;Vonrhein,C.; Arraiano,C.M.;Carrondo,M.A.Nature 2006,443,110.doi: 10.1038/nature05080
(17)Hudson,B.P.;Martinez-Yamout,M.A.;Dyson,H.J.;Wright,P. E.Nature Structural&Molecular Biology 2004,11,257.doi: 10.1038/nsmb738
(18) Lewis,H.A.;Musunuru,K.;Jensen,K.B.;Edo,C.;Chen,H.; Darnell,R.B.;Burley,S.K.Cell 2000,100,323.doi:10.1016/ S0092-8674(00)80668-6
(19) Lu,D.;Searles,M.A.;Klug,A.Nature 2003,426,96.doi: 10.1038/nature02088
(20) Ma,J.B.;Ye,K.;Patel,D.J.Nature 2004,429,318.doi: 10.1038/nature02519
(21) Ma,J.B.;Yuan,Y.R.;Meister,G.;Pei,Y.;Tuschl,T.;Patel,D. J.Nature 2005,434,666.doi:10.1038/nature03514
(22) Oberstrass,F.C.;Lee,A.;Stefl,R.;Janis,M.;Chanfreau,G.; Allain,F.H.Nature Structural&Molecular Biology 2006,13, 160.doi:10.1038/nsmb1038
(23) Oubridge,C.;Ito,N.;Evans,P.R.;Teo,C.H.;Nagai,K.Nature 1994,372,432.doi:10.1038/372432a0
(24)Ramos,A.;Grunert,S.;Adams,J.;Micklem,D.R.;Proctor,M. R.;Freund,S.;Bycroft,M.;St Johnston,D.;Varani,G.The EMBO Journal 2000,19,997.doi:10.1093/emboj/19.5.997
(25) Sachs,R.;Max,K.E.;Heinemann,U.;Balbach,J.RNA 2012, 18,65.doi:10.1261/rna.02809212
(26) Schumacher,M.A.;Pearson,R.F.;Moller,T.;Valentin-Hansen, P.;Brennan,R.G.The EMBO Journal 2002,21,3546.doi: 10.1093/emboj/cdf322
(27) Wang,X.;McLachlan,J.;Zamore,P.D.;Hall,T.M.Cell 2002, 110,501.doi:10.1016/S0092-8674(02)00873-5
(28) Cook,K.B.;Kazan,H.;Zuberi,K.;Morris,Q.;Hughes,T.R. Nucleic Acids Research 2011,39,D301.
(29) Tian,B.;Bevilacqua,P.C.;Diegelman-Parente,A.;Mathews, M.B.Nature Reviews.Molecular Cell Biology 2004,5,1013. doi:10.1038/nrm1528
(30) Schumacher,M.A.;Karamooz,E.;Zikova,A.;Trantirek,L.; Lukes,J.Cell 2006,126,701.doi:10.1016/j.cell.2006.06.047
(31) Das,R.;Karanicolas,J.;Baker,D.Nature Methods 2010,7, 291.doi:10.1038/nmeth.1433
(32) Sharma,S.;Ding,F.;Dokholyan,N.V.Bioinformatics 2008,24, 1951.doi:10.1093/bioinformatics/btn328
(33) Martinez,H.M.;Maizel,J.V.,Jr.;Shapiro,B.A.Journal of Biomolecular Structure&Dynamics 2008,25,669.doi: 10.1080/07391102.2008.10531240
(34) Parisien,M.;Major,F.Nature 2008,452,51.doi:10.1038/ nature06684
(35) Zhao,Y.;Gong,Z.;Xiao,Y.Journal of Biomolecular Structure &Dynamics 2011,28,815.doi:10.1080/07391102.2011. 10508609
(36) Shulman-Peleg,A.;Nussinov,R.;Wolfson,H.J.Nucleic Acids Research 2009,37,D369.
(37) Lewis,B.A.;Walia,R.R.;Terribilini,M.;Ferguson,J.;Zheng, C.;Honavar,V.;Dobbs,D.Nucleic Acids Research 2011,39, D277.
(38)Wang,Y.;Li,Y.;Ma,Z.;Yang,W.;Ai,C.PLoS Comput.Biol. 2010,6,e1000866.
(39)Jones,S.;Daley,D.T.;Luscombe,N.M.;Berman,H.M.; Thornton,J.M.Nucleic Acids Research 2001,29,943.doi: 10.1093/nar/29.4.943
(40)Allers,J.;Shamoo,Y.J.Mol.Biol.2001,311,75.doi:10.1006/ jmbi.2001.4857
(41) Ellis,J.J.;Broom,M.;Jones,S.Proteins 2007,66,903.
(42) Chen,Y.C.;Lim,C.Nucleic Acids Research 2008,36,7078. doi:10.1093/nar/gkn868
(43) Bahadur,R.P.;Zacharias,M.;Janin,J.Nucleic Acids Research 2008,36,2705.doi:10.1093/nar/gkn102
(44) Shazman,S.;Mandel-Gutfreund,Y.PLoS Comput.Biol.2008, 4,e1000146.
(45) Brandman,R.;Brandman,Y.;Pande,V.S.PLoS One 2012,7, e30022.
(46) Terribilini,M.;Lee,J.H.;Yan,C.;Jernigan,R.L.;Honavar,V.; Dobbs,D.RNA 2006,12,1450.doi:10.1261/rna.2197306
(47) Kim,O.T.;Yura,K.;Go,N.Nucleic Acids Research 2006,34, 6450.doi:10.1093/nar/gkl819
(48) Chen,Y.C.;Lim,C.Nucleic Acids Research 2008,36,e29.
(49)Kumar,M.;Gromiha,M.M.;Raghava,G.P.Proteins 2008,71, 189.doi:10.1002/prot.v71:1
(50) Zhao,H.;Yang,Y.;Zhou,Y.Nucleic Acids Research 2011,39, 3017.
(51) Perez-Cano,L.;Fernandez-Recio,J.Proteins 2010,78,25.doi: 10.1002/prot.22527
(52) Ma,X.;Guo,J.;Wu,J.;Liu,H.;Yu,J.;Xie,J.;Sun,X.Proteins 2011,79,1230.
(53)Chen,Y.;Kortemme,T.;Robertson,T.;Baker,D.;Varani,G. Nucleic Acids Research 2004,32,5147.doi:10.1093/nar/ gkh785
(54) Zheng,S.;Robertson,T.A.;Varani,G.The FEBS Journal 2007, 274,6378.doi:10.1111/j.1742-4658.2007.06155.x
(55) Perez-Cano,L.;Solernou,A.;Pons,C.;Fernandez-Recio,J. Pac.Symp.Biocomput.2010,293.
(56) Katchalski-Katzir,E.;Shariv,I.;Eisenstein,M.;Friesem,A.A.; Aflalo,C.;Vakser,I.A.Proceedings of the National Academy of Sciences of the United States of America 1992,89,2195.doi: 10.1073/pnas.89.6.2195
(57) Chen,R.;Weng,Z.Proteins 2002,47,281.doi:10.1002/(ISSN) 1097-0134
(58) Gray,J.J.;Moughon,S.;Wang,C.;Schueler-Furman,O.; Kuhlman,B.;Rohl,C.A.;Baker,D.J.Mol.Biol.2003,331, 281.doi:10.1016/S0022-2836(03)00670-3
(59)Dominguez,C.;Boelens,R.;Bonvin,A.M.J.Am.Chem.Soc. 2003,125,1731.doi:10.1021/ja026939x
(60)Li,L.;Guo,D.;Huang,Y.;Liu,S.;Xiao,Y.BMC Bioinformatics 2011,12,36.doi:10.1186/1471-2105-12-36
(61) Zhang,C.;Lai,L.Journal of Computational Chemistry 2011, 32,2598.doi:10.1002/jcc.v32.12
(62)Li,C.H.;Cao,L.B.;Su,J.G.;Yang,Y.X.;Wang,C.X. Proteins 2012,80,14.doi:10.1002/prot.v80.1
(63) Setny,P.;Zacharias,M.Nucleic Acids Research 2011,39,9118. doi:10.1093/nar/gkr636
(64) Tuszynska,I.;Bujnicki,J.M.BMC Bioinformatics 2011,12, 348.doi:10.1186/1471-2105-12-348
(65) Jiang,F.;Kim,S.H.J.Mol.Biol.1991,219,79.doi:10.1016/ 0022-2836(91)90859-5
(66) Gabb,H.A.;Jackson,R.M.;Sternberg,M.J.J.Mol.Biol. 1997,272,106.doi:10.1006/jmbi.1997.1203
(67)Kozakov,D.;Brenke,R.;Comeau,S.R.;Vajda,S.Proteins 2006,65,392.doi:10.1002/prot.21117
(68)Abagyan,R.;Totrov,M.J.Mol.Biol.1994,235,983.doi: 10.1006/jmbi.1994.1052
(69) Wang,C.;Bradley,P.;Baker,D.J.Mol.Biol.2007,373,503. doi:10.1016/j.jmb.2007.07.050
(70) May,A.;Zacharias,M.Proteins 2008,70,794.
(71) Chaudhury,S.;Gray,J.J.J.Mol.Biol.2008,381,1068.doi: 10.1016/j.jmb.2008.05.042
(72) Lesk,V.I.;Sternberg,M.J.Bioinformatics 2008,24,1137.doi: 10.1093/bioinformatics/btn093
(73) Smith,G.R.;Sternberg,M.J.;Bates,P.A.J.Mol.Biol.2005, 347,1077.doi:10.1016/j.jmb.2005.01.058
(74) Huang,Y.;Liu,Z.J.Mol.Biol.2009,393,1143.doi:10.1016/ j.jmb.2009.09.010
(75) Harel,M.;Spaar,A.;Schreiber,G.Biophys.J.2009,96,4237. doi:10.1016/j.bpj.2009.02.054
(76)Kim,Y.C.;Tang,C.;Clore,G.M.;Hummer,G.Proceedings of the National Academy of Sciences of the United States of America 2008,105,12855.doi:10.1073/pnas.0802460105
(77) Tang,C.;Iwahara,J.;Clore,G.M.Nature 2006,444,383.doi: 10.1038/nature05201
(78)Volkov,A.N.;Ubbink,M.;van Nuland,N.A.J.Biomol.NMR 2010,48,225.doi:10.1007/s10858-010-9452-6
(79) Sanchez,I.E.;Ferreiro,D.U.;Dellarole,M.;de Prat-Gay,G. Proceedings of the National Academy of Sciences of the United States of America 2010,107,7751.doi:10.1073/pnas. 0911734107
(80) Harel,M.;Cohen,M.;Schreiber,G.J.Mol.Biol.2007,371, 180.doi:10.1016/j.jmb.2007.05.032
(81) Fawzi,N.L.;Doucleff,M.;Suh,J.Y.;Clore,G.M. Proceedings of the National Academy of Sciences of the United States of America 2010,107,1379.doi:10.1073/ pnas.0909370107
(82) Hwang,H.;Vreven,T.;Janin,J.;Weng,Z.Proteins 2010,78, 3111.doi:10.1002/prot.v78:15
(83) Liang,S.;Zhang,C.;Liu,S.;Zhou,Y.Nucleic Acids Research 2006,34,3698.doi:10.1093/nar/gkl454
(84) Guo,D.;Liu,S.Y.;Huang,Y.Y.;Xiao,Y.Journal of Biomolecular Structure and Dynamics 2012,in press.
(85) Li,X.;Liang,J.Knowledge-Based Energy Functions for Computational Studies of Proteins.In Computational Methods for Protein Structure Prediction and Modeling;Xu,Y.,Xu,D., Liang,J.,Eds.;New York:Springer,2006;Vol.1;p 71.
(86) Zhou,Y.;Duan,Y.;Yang,Y.;Faraggi,E.;Lei,H.Theoretical Chemistry Accounts 2011,128,3.doi:10.1007/s00214-010-0799-2
(87) Zhou,H.;Skolnick,J.Biophys.J.2011,101,2043.doi: 10.1016/j.bpj.2011.09.012
(88) Czaplewski,C.;Liwo,A.;Makowski,M.;Oldziej,S.; Scheraga,H.A.Coarse-Grained Models of Proteins:Theory andApplications.In Multiscale Approaches to Protein Modeling;Kolinski,A.Ed.;Springer,2010;p 35.
(89) Kortemme,T.;Morozov,A.V.;Baker,D.J.Mol.Biol.2003, 326,1239.doi:10.1016/S0022-2836(03)00021-4
(90) Jiang,L.;Kuhlman,B.;Kortemme,T.;Baker,D.Proteins 2005,58,893.doi:10.1002/prot.20347
(91) Jiang,L.;Lai,L.The Journal of Biological Chemistry 2002, 277,37732.doi:10.1074/jbc.M204514200
(92) Jiang,L.;Gao,Y.;Mao,F.;Liu,Z.;Lai,L.Proteins 2002,46, 190.doi:10.1002/(ISSN)1097-0134
(93) Wang,C.X.;Chang,S.;Gong,X.Q.;Yang,F.;Li,C.H.; Chen,W.Z.Acta Phys.-Chim.Sin.2012,28,751. [王存新,常 珊,龚新奇,杨 峰,李春华,陈慰祖.物理化学学报, 2012,28,751.]doi:10.3866/PKU.WHXB201202022
(94) Liu,S.;Li,Q.;Lai,L.Proteins 2006,64,68.doi:10.1002/ prot.20954
(95) Zacharias,M.Protein Science:a Publication of the Protein Society 2003,12,1271.
(96) Liang,S.;Liu,S.;Zhang,C.;Zhou,Y.Proteins 2007,69,244. doi:10.1002/prot.v69:2
(97) Huang,S.Y.;Zou,X.Proteins 2008,72,557.doi:10.1002/ prot.21949
(98) Sun,W.T.Advances in Mechanics 2011,41,60.[孙卫涛.力学进展,2011,41,60.]
(99) Pierce,B.;Weng,Z.Proteins 2007,67,1078.doi:10.1002/ prot.21373
(100) Zhou,H.;Zhou,Y.Protein Science:a Publication of the Protein Society 2002,11,2714.
(101) Lu,H.;Skolnick,J.Proteins 2001,44,223.doi:10.1002/ (ISSN)1097-0134
(102) Samudrala,R.;Moult,J.J.Mol.Biol.1998,275,895.doi: 10.1006/jmbi.1997.1479
(103) Chuang,G.Y.;Kozakov,D.;Brenke,R.;Comeau,S.R.;Vajda, S.Biophys.J.2008,95,4217.doi:10.1529/biophysj. 108.135814
(104) Liu,S.;Vakser,I.A.BMC Bioinformatics 2011,12,280.doi: 10.1186/1471-2105-12-280
(105) Liu,S.;Zhang,C.;Zhou,H.;Zhou,Y.Proteins 2004,56,93. doi:10.1002/prot.20019
(106) Lensink,M.F.;Mendez,R.;Wodak,S.J.Proteins 2007,69, 704.doi:10.1002/prot.21804
(107) Mendez,R.;Leplae,R.;Lensink,M.F.;Wodak,S.J.Proteins 2005,60,150.doi:10.1002/prot.20551
(108) Chang,S.;Jiao,X.;Li,C.H.;Gong,X.Q.;Chen,W.Z.; Wang,C.X.Biophysical Chemistry 2008,134,111.doi: 10.1016/j.bpc.2007.12.005
(109) Li,C.H.;Ma,X.H.;Shen,L.Z.;Chang,S.;Chen,W.Z.; Wang,C.X.Biophysical Chemistry 2007,129,1.doi:10.1016/ j.bpc.2007.04.014
(110) Guerois,R.;Nielsen,J.E.;Serrano,L.J.Mol.Biol.2002,320, 369.doi:10.1016/S0022-2836(02)00442-4
(111)Chen,N.Y.;Su,Z.Y.;Mou,C.Y.Physical Review Letters 2006,96,078103.doi:10.1103/PhysRevLett.96.078103
(112) Korkut,A.;Hendrickson,W.A.Proceedings of the National Academy of Sciences of the United States of America 2009, 106,15673.doi:10.1073/pnas.0907684106
(113) Li,W.;Wolynes,P.G.;Takada,S.Proceedings of the National Academy of Sciences of the United States of America 2011, 108,3504.doi:10.1073/pnas.1018983108
(114) Ellis,J.J.;Jones,S.Proteins 2008,70,1518.
(115) Lindahl,E.;Delarue,M.Nucleic Acids Research 2005,33, 4496.doi:10.1093/nar/gki730
(116) Cavasotto,C.N.;Kovacs,J.A.;Abagyan,R.A.J.Am.Chem. Soc.2005,127,9632.doi:10.1021/ja042260c
(117) Petrone,P.;Pande,V.S.Biophys.J.2006,90,1583.doi: 10.1529/biophysj.105.070045
(118) Korkut,A.;Hendrickson,W.A.Proceedings of the National Academy of Sciences of the United States of America 2009, 106,15667.doi:10.1073/pnas.0907674106
(119) Dobbins,S.E.;Lesk,V.I.;Sternberg,M.J.Proceedings of the National Academy of Sciences of the United States of America 2008,105,10390.doi:10.1073/pnas.0802496105
(120) Pons,C.;Solernou,A.;Perez-Cano,L.;Grosdidier,S.; Fernandez-Recio,J.Proteins 2010,78,3182.doi:10.1002/prot. v78:15
(121) Perez-Cano,L.;Jimenez-Garcia,B.;Fernandez-Recio,J. Proteins 2012,80,1872.
(122) Barik,A.;Nithin,C.;Manasa,P.;Bahadur,R.P.Proteins 2012, 80,1866.
(123) Hermann,T.Angew Chem.Int.Edit.2000,39,1890.doi: 10.1002/(ISSN)1521-3773
(124) Mackay,J.P.;Font,J.;Segal,D.J.Nature Structural& Molecular Biology 2011,18,256.doi:10.1038/nsmb.2005
(125) Filipovska,A.;Razif,M.F.;Nygard,K.K.;Rackham,O. Nature Chemical Biology 2011,7,425.doi:10.1038/ nchembio.577
(126) Dong,S.;Wang,Y.;Cassidy-Amstutz,C.;Lu,G.;Bigler,R.; Jezyk,M.R.;Li,C.;Hall,T.M.;Wang,Z.The Journal of Biological Chemistry 2011,286,26732.doi:10.1074/jbc. M111.244889
(127) Chen,Y.;Varani,G.Chemistry&Biology 2011,18,821.
(128) Lu,G.;Dolgner,S.J.;Hall,T.M.Current Opinion in Structural Biology 2009,19,110.doi:10.1016/ j.sbi.2008.12.009
(129) Cooke,A.;Prigge,A.;Opperman,L.;Wickens,M. Proceedings of the National Academy of Sciences of the United States of America 2011,108,15870.doi:10.1073/ pnas.1105151108
(130) Koh,Y.Y.;Wang,Y.;Qiu,C.;Opperman,L.;Gross,L.;Tanaka Hall,T.M.;Wickens,M.RNA 2011,17,718.doi:10.1261/ rna.2540311
(131) Wells,J.A.;McClendon,C.L.Nature 2007,450,1001.doi: 10.1038/nature06526
(132) Arkin,M.R.;Wells,J.A.Nat.Rev.Drug.Discov.2004,3,301. doi:10.1038/nrd1343
(133)Arkin,M.Curr.Opin.Chem.Biol.2005,9,317.doi:10.1016/ j.cbpa.2005.03.001
(134) Bourgeas,R.;Basse,M.J.;Morelli,X.;Roche,P.PloS One 2010,5,e9598.
(135) Zhang,C.;Lai,L.Biochemical Society Transactions 2011,39, 1382.doi:10.1042/BST0391382
(136) Liu,S.;Zhu,X.;Liang,H.;Cao,A.;Chang,Z.;Lai,L. Proceedings of the National Academy of Sciences of the United States of America 2007,104,5330.doi:10.1073/ pnas.0606198104
(137) Fleishman,S.J.;Whitehead,T.A.;Ekiert,D.C.;Dreyfus,C.; Corn,J.E.;Strauch,E.M.;Wilson,I.A.;Baker,D.Science 2011,332,816.doi:10.1126/science.1202617
(138) Der,B.S.;Kuhlman,B.Science 2011,332,801.doi:10.1126/ science.1207082
(139) Zhang,C.;Lai,L.Proteins 2012,80,1078.doi:10.1002/prot. v80.4
(140) Bai,H.J.;Lai,L.H.Acta Phys.-Chim.Sin.2010,26,1988. [白红军,来鲁华.物理化学学报,2010,26,1988.]doi: 10.3866/PKU.WHXB20100725
August 23,2012;Revised:September 11,2012;Published on Web:September 11,2012.
Protein-RNA Interaction Interface Prediction and Design
HUANG Yang-Yu YANG Xiu-Feng LI Hao-Tian JI Xiao-Feng CHENG Hong-Li ZHAO Yun-Jie GUO Da-Chuan LI Lin LIU Shi-Yong*
(Biomolecular Physics and Modeling Group,School of Physics,Huazhong University of Science and Technology, Wuhan 430074,P.R.China)
RNA-protein interactions play key roles in many biological processes.The three dimensional (3D)structure of protein-RNA complexes can be determined experimentally by structural biologists.The recognition between protein and RNA can be understood from the 3D atomic structure.However,the structure determination of protein-RNA complexes by experimental methods is often difficult and costly, and limited to the binding strength.Thus,the prediction and design of protein-RNA complex structures is important in biological medical research.In this review,we will discuss the recent progress in protein-RNA interface prediction and design,which includes the following aspects:(1)protein-RNA docking and the conformational change on binding;(2)the recognition mechanism of protein-RNA binding;(3)the molecular design based on the protein-RNA interface.Improvement of the protein-RNA docking algorithm will help us annotate a large number of proteins and RNA with unknown function,and molecular design based on macromolecular interactions will be useful in drug design.
Protein-RNA interaction;Molecular docking;Interface design;Complex structure prediction
10.3866/PKU.WHXB201209111
∗Corresponding author.Email:liushiyong@gmail.com;Tel:+86-27-87558335-805.
The project was supported by the National Natural Science Foundation of China(31100522),National High Technology Research and Development Program of China(2012AA020402),Specialized Research Fund for the Doctoral Program of Higher Education,China(20110142120038).
国家自然科学基金(31100522),国家高技术研究发展计划(2012AA020402),高等学校博士学科点专项科研基金(20110142120038)资助
O641
