Shaker通道电压感受器S3-S4片段末端氨基酸分析
2012-12-03尚德志张丽艳汲坤沈薇牟均
尚德志,张丽艳,汲坤,沈薇,牟均
(沈阳医学院1.口腔医学院颌面外科;2.基础医学院病理生理教研室;3.国资处,沈阳11003 4)
电压门控钾通道(Kv)由电压感受器(S1-S4)和孔道区域(S5-P-S6)组成。传统的电压门控机制Shaker模型建立在大量电生理数据基础之上,认为S3-S4是跨膜片段,S4因带有大量电荷,位于由S1-S3片段及相邻亚单位的S5组成的水性环境中间[1]。随着古细菌电压门控KvAP蛋白X-ray结晶的成功,学者们又提出了1个新的电压门控机制模型,即KvAP模型。该模型认为S3-S4形成1个桨状物,游离于孔道区域之外,静息状态时,平行于膜的内表面,除极化时,通过杠杆式外移转为跨膜片段[2]。两大机制模型分歧的焦点在于电压感受器S4的结构特点,如S4在膜中所处的位置,S4与其他跨膜片段的空间位置关系等。膜拓扑结构分析是研究膜蛋白的各个跨膜片段在蛋白合成的同时如何整合到内质网膜上,并最终形成什么样的跨膜结构。糖基化反应常被用来分析膜蛋白的拓扑结构[3],而糖基化反应是否有效进行取决于其糖基化受体位点是否符合12-14规则[4],此规则已被用来分析膜蛋白跨膜区的边界[5]。本研究采用膜拓扑结构分析和糖基化扫描方法分析了Kv家族中具有代表性的Shaker通道的电压感受器S3-S4的膜拓扑结构,旨在推断其在膜中所处的位置,为分析电压门控机制提供了结构上的信息。
1 材料与方法
1.1 质粒构建
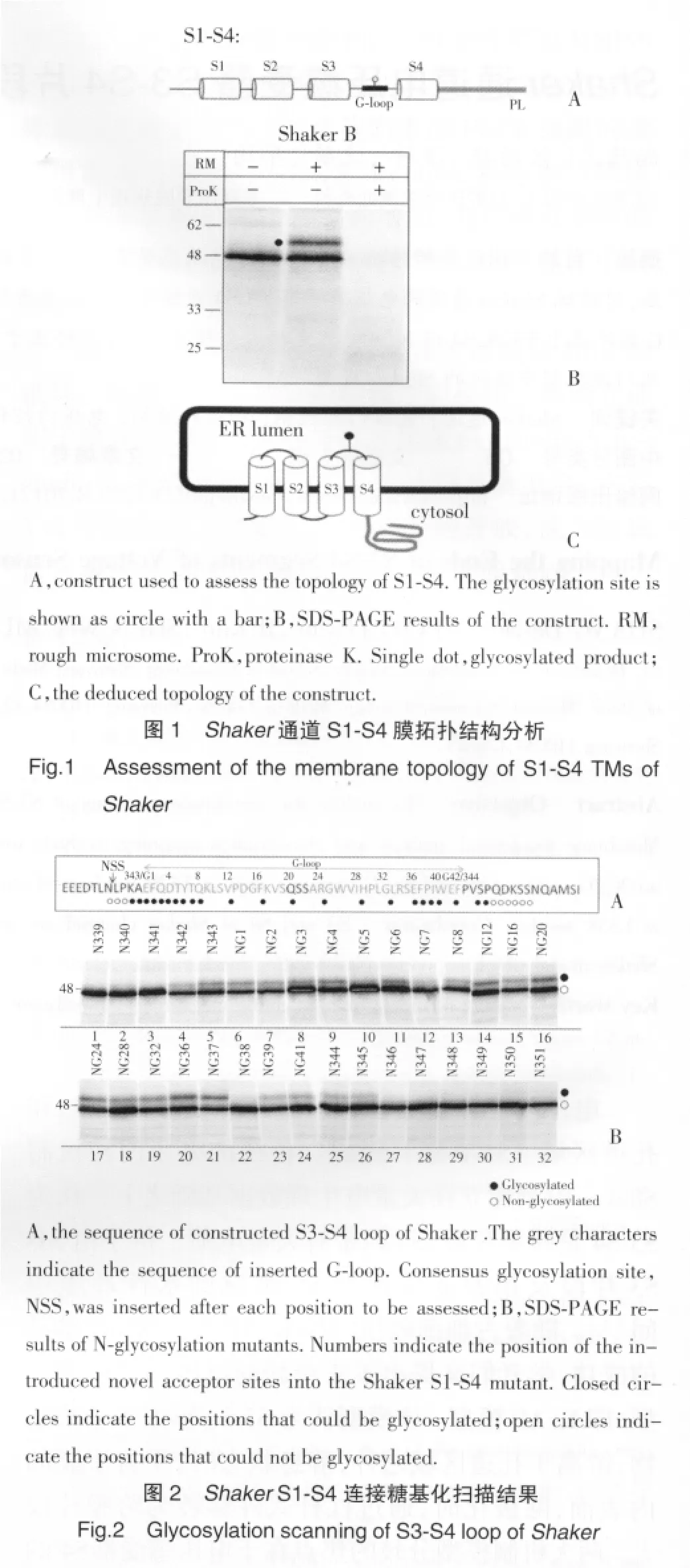
采用2步PCR方法,去除Shaker通道的N端,以增加该蛋白的体外合成效率,同时通过点变异的方式去除Shaker通道位于S1-S2连接中的2个内源性糖基化受体位点N259和N263,然后把来自于人带3蛋白中含有1个糖基化受体位点的G-loop插入到S3-S4连接中,以分析S3-S4的膜拓扑结构,最后把编码ShakerS1-S4(K198-F401)区域的DNA融合到携带有PL报告基因的pCITE-2a载体(No-vagen,Madison,WI)中(图1A)。
1.2 无细胞系统蛋白转录、翻译及转运实验
将所有质粒通过ScaI酶切线性化,在标准条件下利用T7 RNA多聚酶进行体外转录,37℃,1 h。把获得的mRNA在含有或不含有狗胰腺粗面内质网小泡(rough microsome,RM)的兔网织红细胞抽出液系统中进行体外蛋白合成和转运,30℃,1 h。采用蛋白激酶K对在含有RM条件下合成的蛋白产物进一步进行蛋白水解实验,用[35S]蛋氨酸标记蛋白,SDS-PAGE电泳分离蛋白产物,最后进行同位素自显影。
1.3 糖基化扫描方法
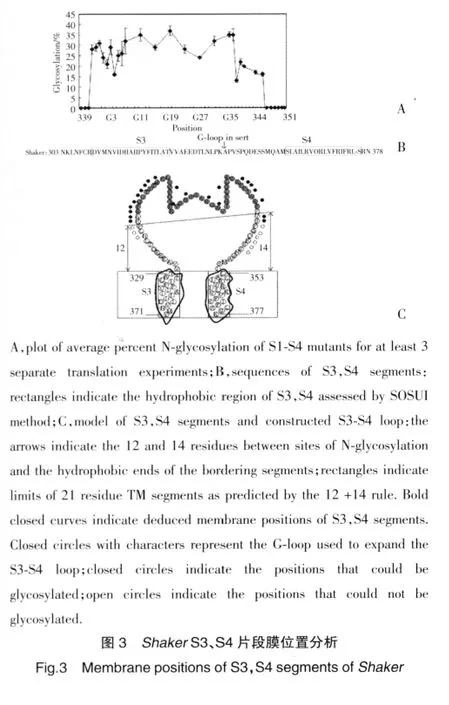
在已获得有效糖基化的ShakerS1-S4构建体基础上,把NSS受体位点分别引进到S3-S4连接的不同位置(图2A),然后根据糖基化的有无及其程度和12-14规则来推测可能的S3、S4跨膜末端。糖基化效率由糖基化的蛋白带的强度除以糖基化蛋白带与未被糖基化的蛋白带强度之和计算获得。有效的糖基化效率定义为至少应该达到最大糖基化效率的一半,突然的糖基化效率下降可视为该糖基化位点超出所谓的12-14距离。
2 结果
2.1 Shaker中S1-S4的膜拓扑结构分析
在未添加RM的条件下,ShakerS1-S4-PL经体外合成后,出现1条分子量约为50 kDa的蛋白带,这是未被糖基化的蛋白带(图1B)。在RM存在的条件下,出现1条明显的糖基化蛋白带,分子量比未被糖基化的蛋白带多出约3 kDa。在RM存在条件下获得的蛋白经过蛋白激酶K处理后,所有蛋白带消失。提示Shaker的S3-S4能被有效地转移整合到内质网膜上,S3是以Ncyt/Cexo的方向,S4是以Ncyt/Cexo的方向插入到膜上的(图1C)。
2.2 Shaker中S3、S4片断末端氨基酸残基分析
图2B为糖基化扫描的SDS-PAGE结果,其糖基化效率见图3A。NSS位点在G20的构建体是基础质粒(同图1),其糖基化效率(36.0%±1.8%)与图1相似,为有效糖基化。第1个被糖基化的受体位点位于残基342,其糖基化效率为28.0%±2%;最后1个被糖基化的受体位点位于残基345,其糖基化效率为18.0%±0.8%,说明这2个受体位点恰好位于能与寡糖基转移酶的活性位点结合的最短距离。虽然最后几个糖基化位点的效率偏低,但均能达到最大效率的50%,G38位点除外。根据12-14规则,可推测出Shaker中S3-S4跨膜片段的末端氨基酸位置(图3C)。Shaker通道中,S3的C端位于T329,S4的N端位于L358。
2.3 Shaker中S3、S4片段膜中位置分析
SOSUI疏水性分析发现S3的N端起始于V311,C 端终止于 T329;S4的 N 端起始于 L358,C端终止于R377,见图3B。其S3的C端残基和S4的N端残基与图3C推测出的数据一致,由此推论Shaker的S3与S4均为跨膜片段。
3 讨论
目前,常利用无细胞系统进行膜蛋白的体外转录、翻译和转运实验,然后根据合成的蛋白肽链是否发生糖基化反应,以及是否被蛋白水解酶降解等特点,分析膜蛋白的拓扑结构[3]。蛋白肽链的糖基化反应通常在内质网膜腔内进行,在寡糖基转移酶的作用下,1个糖基化受体位点(Nsn-X-Ser/Thr,其中X为任何一种氨基酸)接受1个高甘露糖基寡聚糖,其分子量会增加2.5 kDa。图1B中,在RM存在的条件下,糖基化蛋白带的出现说明S3-S4连接位于内质网腔内(细胞外),所以其人工导入的G-loop上的糖基化受体位点才能被糖基化。糖基化效率为(37.0±2.0)%,说明该位点被有效地糖基化。位于内质网腔内的蛋白肽链除了可发生糖基化反应外,因受到内质网膜的保护,不会被蛋白水解酶降解。图1B中,在RM存在下获得的蛋白肽链经过蛋白激酶K处理后,所有蛋白带消失,说明S1-S4蛋白肽链C端(含报告蛋白PL)位于内质网腔外(细胞内),因而被蛋白激酶K消化降解。由这些结果推断出S3是以Ncyt/Cexo的方向,S4是以Ncyt/Cexo的方向整合到膜上的。
膜蛋白的糖基化反应是否有效进行取决于膜蛋白的糖基化受体位点能否与膜上寡糖基转移酶的活性位点结合,而该活性位点距内质网膜有一定的距离。1993年Nilsson等[4]利用大肠杆菌前导肽酶提出了12-14规则,认为糖基化受体位点的N端距内质网膜表面至少有12个残基的距离,C端至少有14个残基的距离,才能被有效糖基化[4]。
本研究通过糖基化扫描的方法,根据12-14规则,推测出ShakerS3-S4片段的细胞外末端氨基酸残基,并由此推测出其在膜中可能的位置。因为膜蛋白在内质网翻译转运的同时进行折叠,形成蛋白的3级结构,因而其膜整合的一些结构信息可能代表着膜蛋白的最终结构信息。目前,对于Kv通道电压门控机制的研究主要集中在电生理和X-ray结晶方面[6,7],本研究试图从膜拓扑结构分析的角度对其进行评价。我们的糖基化扫描结果认为Shaker的S3、S4为跨膜片断,与传统的Shaker电压门控机制模型一致,为电压门控机制的研究提供了线索。
[1]Heginbotham L,Lu Z,Abramson T,et al.Mutations in the K+channel signature sequence[J].Biophys J,1994,66(4):1061-1067.
[2]Ahern CA,Horn R.Stirring up controversy with a voltage sensor paddle[J].Trends Neurosci,2004,27(6):303-307.
[3]Zhang L,Sato Y,Hessa T,et al.Contribution of hydrophobic and electrostatic interactions to the membrane integration of the Shaker K+channel voltage sensor domain [J].Proc Natl Acad Sci USA,2007,104(20):8263-8268.
[4]Nilsson IM,von Heijne G.Determination of the distance between the oligosaccharyltransferase active site and the endoplasmic reticulum membrane[J].J Biol Chem,1993,268(8):5798-5801.
[5]Popov M,Tam LY,Li J,et al.Mapping the ends of transmembrane segments in a polytopic membrane protein.Scanning N-glycosylation mutagenesis of extracytosolic loops in the anion exchanger,band3[J].J Biol Chem,1997,272(29):18325-18332.
[6]Koag MC,Papazian DM.Voltage-dependent conformational changes of KVAP S4 segment in bacterial membrane environment[J].Channels(Austin),20093(5):356-365.
[7]Schow EV,Freites JA,Gogna K,et al.Down-state model of the voltage-sensingdomainofapotassiumchannel[J].BiophysJ,2010,98(12):2857-2866.
