辽宁地区猪瘟病毒流行株E2基因序列分析
2012-11-23葛宝伟李晓成于学武段亚良
葛宝伟,张 志,李晓成,于学武,段亚良,高 丰
(1.吉林大学畜牧兽医学院,吉林 长春130062;2.辽宁省动物疫病预防控制中心,辽宁 沈阳110164;3.中国动物卫生与流行病学中心,山东 青岛266032)
猪瘟也称为古典型猪瘟(classical swine fever,CSF),是由猪瘟病毒(CSFV)引起的猪的高度接触性传染病。猪瘟病毒属于黄病毒科(Flaviviridae)、瘟病毒属(Pestivirus),基因组为单股正链RNA,大小约为12.3kb,包括5′-端非翻译区(5′UTR)、一个开放阅读框(ORF)和3′-端非翻译区(3′UTR)[1]。CSFV惟一的开放阅读框可翻译成分子量约438kDa的多聚蛋白,并进一步在宿主细胞和病毒蛋白酶作用下裂解为4种结构蛋白(C,E0,E1,E2)及8种非结构蛋白(Npro,p7,NS2,NS3,NS4A,NS4B,NS5A 和NS5B)[2]。目前认为猪瘟病毒只有一个血清型,但猪瘟病毒不同毒株毒力差异很大,可引起急性猪瘟、亚急性、慢性猪瘟和妊娠母猪繁殖障碍[3]。因此,在核酸序列比较基础上的猪瘟病毒基因分型对于追踪流行毒株来源和传播、阐述病毒遗传变异特征和流行规律具有重要意义。此外,CSFV的E2糖蛋白存在于感染细胞或病毒粒子表面,相对于其他结构蛋白保守性最低,最易发生变异。E2糖蛋白参与了CSFV对细胞的感染过程并与CSFV的毒力有关,携带有能刺激机体产生保护性免疫的抗原决定簇,是CSFV最主要的保护性抗原蛋白。国内外学者认为E2基因变异有可能是导致该病毒逃脱或抵御免疫保护的主要原因[4-6]。鉴于此,本试验利用 RT-PCR的方法对2006年~2011年辽宁省不同地区发病猪群中CSFV的感染情况进行了检测,并对所测定的20株CSFV E2基因与国内外参考毒株的相应序列进行同源性分析及氨基酸序列比对。研究结果,为了解辽宁省不同地区CSFV流行毒株的遗传变异规律以及制定更为科学合理的防控措施提供理论依据。
1 材料与方法
1.1 样品来源及处理 2006年~2011年间,从辽宁省不同地区采集发病猪群的病死及扑杀或流产胎儿组织脏器样品,主要为扁桃体、淋巴结、肾脏、肺脏;待检活猪,用注射器抽取颈静脉血3~5mL。
取待检组织与生理盐水按1∶5(质量浓度)比例,于研钵中充分研磨制成组织匀浆液,8 000r/min(4℃)离心2min,取上清液置于1.5mL灭菌离心管中备用;全血样品待血凝后取血清,置1.5mL灭菌离心管中备用。制备的样品在2℃~8℃保存不应超过24h,长期保存应分装后置-70℃以下,避免反复冻融。
1.2 主要试剂 禽源反转录酶(AMV)、dNTP Mix-ture、Ribonuclease Inhibitor、rTaqDNA聚合酶、DNA Marker DL-2 000、Agarose Gel DNA Purification kit、pMD18-T载体,均为大连TaKaRa公司产品;Trizol为Invitrogen公司产品;TIAN prep Mini Plasmid Kit,均购自北京天根生物有限公司产品。
1.3 引物设计与合成 对GenBank中公布猪瘟病毒基因组序列进行比较分析后,利用软件Primer 5.0针对猪瘟兔化弱毒株(AY805221)E2基因A区保守段设计一对特异性引物,序列如下:
Forward Primer:5′-TTG AGC TCC TGT TCG ACG-3′(18bp)
Reverse Primer:5′-CTG CGG TGG TCA CAC AAT C-3′(19bp)
预期扩增DNA片段长度为276bp,并含有1个型特有的HinfⅠ酶切位点。上述引物由北京博尚生物工程公司合成,上游用灭菌双蒸水稀释,下游用RNAfree水稀释,引物浓度为20pmol/μL,分装保存于-20℃冰箱备用。
1.4 总RNA的提取及cDNA的合成 采用Invitrogen公司Trizol试剂一步法提取总RNA,取已处理备用的样品200μL于1.5mL灭菌EP管中,加入600 μL Trizol LS Reagent试剂,上下颠倒混匀,4℃静置5 min;加入200μL氯仿,充分震荡至乳化均匀,4℃静置15min后,12 000r/min(4℃)离心15min;轻轻吸取上清约450μL转移入另一1.5mL灭菌EP管,加入500μL预冷的异丙醇,轻轻颠倒混匀,置-20℃过夜;12 000r/min(4℃)离心10min;弃去上清液,加入1mL预冷的75%乙醇,12 000r/min(4℃)离心10 min;弃去上清液,室温干燥后,溶于25μL DEPC水中;同时设阴性对照和阳性对照。
反转录反应体系及条件按反转录酶说明书进行,20μL反转录体系中含RNA模板1μg,5×AMV Buffer 4μL,dNTP Mixture(各10mmol/L)2μL,Ribonuclease Inhibitor(40U/μL)0.5μL,Reverse Primer 2μL,AMV(5U/μL)2μL,用 DEPC水补至20μL。轻轻混匀,室温放置10min后移入42℃恒温槽中水浴1h,然后冰浴2min,所得反应液用于PCR,或-20℃保存备用。
1.5 E2基因PCR扩增 以cDNA为模板,建立25μL的PCR反应体系,反应体系为:10×PCR Buffer 5μL,dNTP Mixture(2.5mmol/L)4μL,rTaq(5U/μL)0.125μL,上下游引物(20pmol/μL)各0.125μL,cDNA 10μL,用灭菌ddH2O补至25μL。反应条件为:95℃预变性5min,94℃30s,58℃30s,72℃45s共进行35个循环,最后72℃充分延伸10min。
1.6 PCR扩增片段的回收与克隆 利用凝胶回收试剂盒回收PCR扩增阳性产物,经纯化后连接入pMD-18T载体,转化至JM109感受态菌,挑选阳性克隆菌落,接种2mL LB培养基,37℃摇震培养过夜,然后提取经PCR鉴定为阳性克隆菌株的质粒。1.7 序列测定与分析 将阳性克隆质粒送交宝生物工程(大连)有限公司进行测序,对测定的序列首先应用DNAStar软件中Edit进行了编辑,然后用采MegAlign软件中Clustal W算法进行比较和分析,同时结合MEGA3.1软件进行NJ进化分析。
2 结果与分析
2.1 PCR扩增结果 利用RT-PCR方法检测在2006年~2011年辽宁省发病猪群中猪瘟病毒感染情况,共鉴定出20株猪瘟病毒分别命名为LN169-09、LN221-10、LN236-10、LN261-10、LN274-10、LN284-11、LN288-11、LN291-11、LN299-11、LN28-07、LN100-08、LN129-08、LN133-09、LN155-09、LN164-09、LN6-06、LN15-06、LN239-10、LN209-10、LN312-11(均为血毒),扩增获得大小为276bp猪瘟病毒的E2基因片段(见图1)。
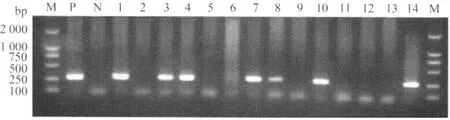
图1 病料中CSFV的检测结果
2.2 遗传演化分析 在序列测定的20株猪瘟病毒中,基因1型的毒株有2株分别为LN239-10和LN209-10,其余毒株均为基因2型。一方面,属于基因1型的毒株(LN239-10、LN209-10)全部为基因1.1亚型。另外,通过进化树看出,Shimen株和猪瘟兔化疫苗株HCLV都属于基因1.1亚型,而被进一步分型为疫苗株的1.1a亚型和野毒Shimen株的1.1b亚型。通过进化树可以看出,LN239-10和LN209-10分离毒株均属于1.1a亚型,有可能为疫苗株。另一方面,属于基因2型18株野毒株又分别属于基因2.1亚型(17株)和2.2亚型(1株为LN169-09)并且属于基因2.1亚型毒株的比例高达94.4%,这说明基因2.1亚型的猪瘟野毒株已经成为辽宁省猪瘟野毒的主要流行毒株(见图2)。
2.3 辽宁省猪瘟流行病毒变异分析 从上述分析可以看出,辽宁省20份野毒株分别属于基因2.1、2.2亚型和1.1亚型,通过软件分析发现,在这20株野毒株中,属于基因2型的18株野毒株与我国疫苗株HCLV的同源性在76.8%~80.5%之间,与我国经典强毒Shimen株的同源性在77.4%~81.1%之间,而属于基因1型的2株猪瘟毒株与疫苗株HCLV和Shimen株的同源性分别为97.4%和93.2%(见表1)。
此外,不同的基因亚群在E2基因高变区的氨基酸组成上有各自的特点,其中某些位置的氨基酸对毒株分类起着重要作用[6-7],如第720(K)、725(D)、729(N)、738(V)位的氨基酸是基因1.1亚型的特异性氨基酸,而,第720(R)、725(G)、729(D)、738(I)的氨基酸对于基因2.1亚型的流行毒株也起到重要作用。而利用软件分析表明,辽宁猪瘟流行毒株的变异位点主要分布在所测序列的前30个氨基酸。在流行毒株与弱毒疫苗之间有15个氨基酸的变异(15/63),与经典强毒石门株有14个氨基酸的变异(14/63),与强毒Shimen株、疫苗株相比其变异率高达20%以上(见表2和图3)。
3 讨论
猪瘟病毒参考序列来自NCBI网站,选择的参考序列包括国际猪瘟参考实验室提供的猪瘟基因1.1、1.2、1.3、3.2、3.3、3.4、2.1、2.2、2.3 型 等各型参考毒株。另外还选择了国内部分省市发表的猪瘟E2序列作为参考毒株进行比较。根据时间与地区分布,本试验选择性获得了20株猪瘟病毒大小为276bp的E2基因序列,在分析这些猪瘟病毒E2基因序列的基础上构建了近6年辽宁地区猪瘟流行毒株的系统进化树,进一步分析了辽宁猪瘟病毒的分子流行现状和趋势,分析结果表明,基因2.1亚型的猪瘟野毒已经成为辽宁猪瘟病毒的优势流行毒株。


?
根据常规流行病学和血清学的监测,迄今为止尚未发现CSF有不同的血清型[3]。但事实上,CSFV有了显著的变异,用单克隆抗体及核苷酸序列分为不同的群或型[7-10]。而本试验通过以基因测序为基础的系统树分析试验,明确了辽宁猪瘟流行毒株与国内外毒株的遗传相关性,2006年~2011年辽宁省流行毒株的优势基因型与传统的石门系强毒、兔化弱毒存在核苷酸序列上存在较大差异,已由基因1型转为基因2型,向远离疫苗毒的方向变化,猪瘟的流行毒株与疫苗株之间的同源性在76.8%~80.5%之间,与经典强毒Shimen株的同源性在77.4%~81.1%之间,这一变化趋势与国内其他地区一致[11-12]。本试验还发现,辽宁猪瘟流行毒株的变异主要分布在所测序列的前30个氨基酸,在流行毒株与弱毒疫苗毒株之间有15个氨基酸的变异(15/63),与经典强毒石门株有14个氨基酸的变异(14/63),变异率高达20%以上,表明近年来,辽宁流行毒株的优势基因型与传统的石门系强毒、兔化弱毒在抗原性上存在较大差异,从而使辽宁地区猪瘟流行毒株可能逃脱兔化弱毒株的免疫保护,导致不完全免疫。分析产生上述变化的可能原因是,猪瘟疫苗的长期、普遍使用、温和毒力毒株的出现以及持续性感染的存在等因素,使得猪瘟病毒在感染动物体内停留的时间更长,病毒面临的压力对病毒变异造成的影响作用更加明显。因此,长期开展猪瘟流行毒株分子流行病学的调查研究,密切关注流行毒株的分布,尤其应该对毒力发生改变或是对可能出现的疫苗逃逸株进行研究,特别是对类疫苗源毒株的来源有待于进一步研究是十分必要的。这不但对猪瘟病毒的病原生态学研究具有重要意义,对在世界范围内猪瘟的防控也具有重要影响。

表2 猪瘟病毒基因亚群毒株E2基因主要变异区氨基酸的变异情况

图3 辽宁省猪瘟流行毒株与疫苗株HCLV和Shimen株之间的氨基酸变异图
随着全球经济贸易一体化的不断提高,寻求动物饲养利益最大化的利益驱使,以及鉴于目前猪瘟的流行形式,全球养猪业均面临着有效控制猪瘟的严峻形势,开展猪瘟病毒的分子流行病学研究,建立病毒不同分离株之间的联系,追踪毒株来源,对流行毒株毒力、流行毒株抗原性与疫苗株抗原性差异等不可控制因素的研究,对猪瘟进行全球监控,发现防制策略中的薄弱环节,实现猪瘟病毒遗传信息的全球共享,建立一体化的猪瘟防控体系将具有极其重要的意义。
[1]Stark R,Rumenapf T,Meyers G,etal.Genomic localization of hog cholera virus glycoproteins[J].Virol,1990,174(1):286-289.
[2]Rumenapf T,Unger G,Strauss J H,etal.Processing of the envelope glycoprotein of pestivirus[J].Virol,1993,67(6):3288-3294.
[3]Moennig V.Introduction to classical swine fever virus,disease and control policy[J].Vet Microbio1,2000,73(2-3):93-102.
[4]Lowings P,Ibata G,Needham J,etal.Classical swine fever virus diversity and evolution[J].Gen Virol,1996,77(6):1311-1321.
[5]Stadejek T,Wang J,Ridpath J F,etal.Comparative sequence analysis of the 5′noncoding region of classical swine fever virus strains from Europe,Asia and American[J].Arch Virol,1996,141(3-4):771-777.
[6]Ning Chen,Hongxia Hu,Zhanfeng Zhang,etal.Genetic diversity of the envelope glycoprotein E2of classical swine fever virus:Recent isolates branched away from historical and vaccine strains[J].Vet Microbiol,2008,127(3-4):286-299.
[7]Kosmidou A,AhR,Thiel H J,etal.Differentiation of classical swine fevervirus(CSFV)strains using monoclonal antibodies against structural glycoproteins[J].Vet Microbiol,1995,47(12):111-118.
[8]Welland E,Stark R,Haas B,etal.Pestivirus glycoprotein which induces neutralizing antibodies forms part of a disulfidelinked heterodimer[J].Virol,1990,64(8):3563-3569.
[9]Edwars S,Moening V,Wensvoort G.The development of an international reference panel of monoclonal antibodies for the differentiation of hog cholera virus from other pestivirns[J].Vet Microbiol,1991,29(2):101-108.
[10]Susana Mendoza,Pablo Correa-Giron,Edgar Aguilera,etal.Antigenic differentiation of classical swine fever vaccinal strain PAV-250from other strains,including field strains from Mexico[J].Vaccine,2007,25(41):120-124.
[11]Tu C C,Lu Z J,Li H W,etal.Phylogenetics comparison of classical swine fever virus in China[J].Virus Res,2001,81(1-2):29-37.
[12]朱妍.中国猪瘟病毒流行毒株遗传多样性及抗原性研究[D].长春:吉林大学,2008.
