荣成湾孔鳐群体线粒体DNA cytb部分序列的遗传多样性分析
2012-10-13刘梦侠王丽娟孔令怡刘洪军吴志昊辛梦娇
刘梦侠, 王丽娟, 孔令怡, 刘洪军, 吴志昊, 辛梦娇, 张 伟, 尤 锋
(1. 山东海水养殖研究所, 山东 青岛 266002; 2. 中国科学院 海洋研究所, 山东 青岛 266071; 3. 中国海洋大学 海洋生命学院, 山东 青岛 266003)
孔鳐(Raja porosa), 俗称劳板鱼、劳子, 隶属于软骨鱼纲(Chondrichthyes)、鳐形目(Rajiformes)、鳐科(Rajidae)、鳐属(Raja), 主要分布于我国的黄海和东海, 为冷温性近海底栖鱼类[1-2]。该鱼无硬骨, 肉质细嫩鲜美, 深受消费者青睐, 是一种重要的地方性经济鱼类。近年来, 由于硬骨鱼资源的衰减和对软骨鱼肉、软骨、鱼鳍需求量的增加, 全球对软骨鱼类的捕捞量急增[3], 我国的孔鳐资源也因受过度捕捞、海洋环境污染等因素影响, 导致其资源衰退[4]。遗传多样性是生物进化和环境适应的物质基础, 是种质资源有效保护的前提。海洋生物的遗传结构受到很多海洋因素和生物因素的影响[5], 了解遗传结构可为物种的种质资源检测和恢复提供基本信息,对于鱼类的可持续管理是十分重要的[6]。目前国内外有关孔鳐的研究相对较少, 主要集中在摄食[7]、养殖与捕捞[4,8]、物种鉴定[9-10]、繁殖[11]及硫酸软骨素提取[12]等。
线粒体 DNA(mitochondrial DNA, mtDNA)具有母系遗传、结构简单、进化速度快、几乎不发生重组等特点[13], 其中细胞色素b基因(cytochromeb,cytb)由于研究背景清晰, 进化速度适中, 适合群体水平差异的检验, 经常被用于遗传多样性研究, 在鱼类种群结构的研究中发挥了积极作用。目前, 已经利用cytb部分序列对多种软骨鱼类包括多种鲨鱼和鳐科鱼类进行了群体遗传多样性研究[14-19], 但有关孔鳐群体的研究报道尚未见到。
本文对 2009年和 2011年采自荣成湾的孔鳐群体进行cytb部分序列的测定, 分析了其群体遗传多样性水平, 并比较了两个年度群体遗传差异, 以期了解荣成湾孔鳐的群体遗传结构、不同年度间的遗传分化程度, 为孔鳐资源的开发利用和管理保护提供有效依据。
1 材料与方法
1.1 样品采集
实验用孔鳐为2009年2~11月、2011年2~8月间分别以底拖网方式采自荣成湾海域(122°37′29″~122°46′16″E, 37°20′38″~37°13′26″N), 各为 10尾(编号为 RCRP0901-10)和 44尾(编号为RCRP1101-44)。经过形态鉴定以后, 取样品背部肌肉置于–20℃冻存备用。
1.2 基因组DNA的提取与PCR扩增
取孔鳐肌肉组织约 30 mg, 使用海洋动物组织基因组提取试剂盒(康为世纪生物科技有限公司)提取基因组 DNA, 操作步骤按照使用说明进行。参考GenBank上孔鳐线粒体全基因组序列(AY525783.1),设计一对引物cytbF: 5'-TCATCCGCAACATTCACGCCAAT-3',cytbR: 5'-GGCAAGTGGGAGAAGGGTGAGA-3', 扩增孔鳐cytb部分序列。PCR反应总体积 50 μL, 包括: 2× ES PCR master mix 25 μL(康为世纪生物科技有限公司), 引物(10 μmol/L)各 2 μL, 基因组DNA 50 ng, 补足灭菌双蒸水至终体积50 μL。PCR扩增循环参数为: 94℃预变性5 min; 94℃变性40 s, 58℃退火40 s, 72℃延伸1 min, 共35个循环;72℃再延伸10 min。
PCR扩增产物经1%琼脂糖凝胶电泳检测后, 用胶回收试剂盒(Omega Bio-Tek)对目的片段回收纯化,然后送至上海桑尼生物科技有限公司利用 ABI3730 DNA自动测序仪进行双向测序。
1.3 DNA序列的数据处理
54个样品的测序结果用BioEdit软件进行剪切、拼接和序列核对, 并辅以人工校对。采用MEGA5.05软件包计算群体相应序列的碱基组成及2009年, 2011年度样品间的遗传距离, 构建基于 Kimura-2参数模型的单倍型邻接(Neighbor-Joining, NJ)树, 以Bootstrap 1 000次重复抽样检验分支置信度。用DnaSP5.10软件计算群体单倍型多样性指数(H)、核苷酸多态性指数(π)、平均核苷酸差异数(K)以及群体间遗传分化系数(Fst), 绘制单倍型的歧点分布。用Arlequin3.5软件包计算Tajima’s D和Fu's Fs值, 进行中性检验, 以检验荣成湾孔鳐cytb这段序列是否符合中性变异, 并根据 pairwise difference模型, 进行分子方差分析(AMOVA)。用Network4.0构建单倍型网络结构图。
2 结果与分析
2.1 孔鳐的cytb部分序列的特征
经比对校正后获得了54尾孔鳐的长度为782bp的cytb基因序列, 未发现碱基的插入与缺失, 碱基T, C, A, G平均含量分别为0.275±0.105, 0.325±0.098,0.265±0.061, 0.135±0.054, 其中 A+T(54%)平均含量略大于G+C(46%), 与其他硬骨鱼类相似[20]。序列中共出现11个单倍型, 检测到多态位点15个, 占核苷酸总数的1.92%, 简约信息位点6个, 单一变异位点9个。序列中发生转换14次, 颠换1次, 转换明显高于颠换, 转换与颠换的比值远高于 Li[21]所提出的期望值 0.5, 表明此片段没有饱和, 适合进行遗传变异分析。其中, 2009年群体序列中共出现3个单倍型,检测到多态位点4个, 全部为单一变异位点; 2011年群体序列中出现9个单倍型, 检测到多态位点12个,包括简约信息位点6个, 单一变异位点6个。
在检测到的11个单倍型中, Hap1在2个年度中均有出现, 为共享单倍型, 其频率也最高, 为70.4%, 其他单倍型则仅在1个年度出现, 在独享单倍型中, 除Hap5、Hap7外, 其余单倍型均只检出一次(表 1)。

表1 荣成湾2009年和2011年孔鳐群体线粒体cytb基因单倍型分布Tab. 1 The cytb haplotype distribution in 2009 and 2011 Raja porosa groups in the Rongcheng Bay
2.2 荣成湾孔鳐群体的系统发育与遗传分化
荣成湾孔鳐群体的单倍型数、单倍型多样性和核苷酸多样性指数见表 2。其单倍型多样性指数为0.498, 核苷酸多样性指数为0.0018。2009年孔鳐群体单倍型多样性指数为0.378, 2011年群体较2009年略高, 为0.532。2个年度核苷酸多样性指数分别为0.0010和0.0020, 也是2011年的略高于2009年的。

表2 荣成湾2009, 2011年孔鳐群体cytb基因部分序列的遗传多样性指数Tab. 2 Genetic diversity parameters of partial cytb gene sequences of R. porosa groups in the Rongcheng Bay in 2009 and 2011
2009年和 2011年孔鳐群体内的 K2p (Kimura 2-parameter)遗传距离分别为 0.0015和 0.0013, 而 2个年度群体间的遗传距离则为0.0013,Fst为0.01778,表明 2个年度群体间无显著遗传分化。荣成湾孔鳐群体年度间和年度内的 AMOVA结果显示, 遗传变异主要集中在相同年度内的个体间, 占 98.22%, 年度间变异仅占1.78%, 年度内变异大于年度间变异。2009年和 2011年群体并不存在显著的遗传结构差异。
使用K2p模型构建的11种单倍型的邻接系统树如图 1所示, 11种单倍型明显形成了两支, 其中Hap4, Hap7, Hap9聚成一支, 其余单倍型聚成一支。2009年和2011年样品来源的单倍型分布于各支, 没有表现出明显的聚群。利用个体序列构建的邻接系统树与单倍型所建进化树结果一致(图2)。
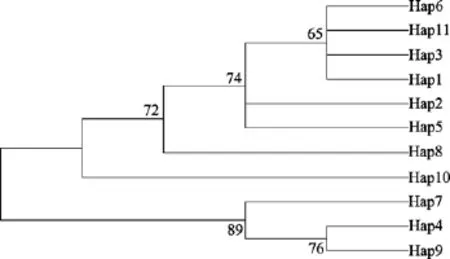
图1 荣成湾孔鳐群体线粒体cytb部分序列单倍型邻接树Fig. 1 Haplotype neighbor-joining tree constructed from partial sequences of cytb in Raja porosa stock of the Rongcheng Bay
用Network的Median-joining方法构建的单倍型网络结构如图3, 所有单倍型通过单一突变和多步突变相连接, 单倍型 Hap1是共有的单倍型, 在 2009,2011年群体中所占比例均最高, 据此推测Hap1是原始单倍型, 其他单倍型是由其演化而来[22]。单倍型网络图的结果也支持不存在与年度相对应的单倍型分支。
2.3 种群历史动态
荣成湾孔鳐群体的Tajima’s D和Fu’s Fs中性检测都呈显著性负值(表 2), 此外, 荣成湾群体的歧点分布也呈单峰(图 4), 表明荣成湾孔鳐群体在过去可能发生了种群的快速扩张[23-24]。
3 讨论
线粒体DNAcytb部分序列的单倍型多样性和核苷酸多样性指数是衡量群体遗传多样性水平的重要指标。通过与目前已报道的其他 8种软骨鱼类的相关数据比较可知(表 3), 荣成湾孔鳐的H与同属的Raja clavata相近, 略高于西大西洋护士鲨(Ginglymostoma cirratum)和西南大西洋舒氏星鲨(Mustelus schmitti), 而低于其他5种软骨鱼类。但其π值却明显低, 除了与西南大西洋舒氏星鲨接近和略低于三种睡鲨Somniosus pacificus,S. antarcticus和S. microcephalus外, 远远低于同科的鳐类(Amblyraja radiata)以及鹞鲼科的Aetobatus narinari、甚至同属的Raja clavata欧洲群体。荣成湾孔鳐具有如此低的H和π值(H<0.5,P<0.005), 不仅显示该群体的遗传多样性水平较低, 根据Grant等[25]的理论推测, 荣成湾孔鳐群体近期也可能发生了遗传瓶颈效应或奠基者效应, 其原因是由于孔鳐的迁徙性不强, 少与其他群体发生基因交流所致, 还是其他原因引起的,尚需进一步予以考证。

图2 荣成湾孔鳐群体线粒体cytb部分序列个体邻接树Fig. 2 Neighbor-joining tree constructed from partial sequences of cytb in R. porosa stock of the Rongcheng Bay
群体遗传多样性是生物适应环境与进化的基础,物种遗传多样性低, 则更容易受到环境变化和过度捕捞的影响[26]。从上述分析的多个群体遗传多样性参数来看, 荣成湾孔鳐的遗传多样性水平相对较低,仅从遗传学角度而言, 该物种的种质资源现状不容乐观。作为一种需求量较大的传统渔业资源, 其种质资源遗传多样性的保护应该得到足够的重视, 并进一步采用多种分子标记分析对孔鳐种质资源进行中长期评估, 从而为其种质资源保护政策制定及实施提供基础, 避免群体遗传多样性的急剧下降。
目前关于水生生物群体遗传多样性在时间动态上的研究相对空间水平研究较少, 而且主要集中在分析亲代和子代之间, 以及不同历史时期之间样本的遗传差异[27]。本文对荣成湾2009年和2011年的孔鳐群体的cytb部分序列进行分析, 通过对反映群体间遗传分化程度的重要指标—遗传分化系数计算发现, 孔鳐在2个年度群体间的Fst为 0.01778, 表明2个年度群体间没有明显的分化[28]。AMOVA结果也显示, 全部的变异几乎都来源于相同年度内, 年度间变异仅占 2%。采用 Kimura双参数模型计算孔鳐群体不同年度间的遗传距离发现, 其值也很小, 仅为0.0015。综合分析, 说明该海域孔鳐群体在时间上不存在明显的群体遗传结构差异, 认为这两个年度的孔鳐属同一个群体。但是, 由于2009年所采集到的样品数较少, 今后还需逐年采取足够的样品数进行研究, 以更准确全面地了解荣成湾孔鳐的群体遗传结构及其年度变化。

图3 荣成湾孔鳐群体线粒体cytb部分序列单倍型的中接网络图Fig. 3 Median Joining Haplotype Network Based on partial sequences of cytb in R. porosa stock of the Rongcheng Bay

图4 荣成湾孔鳐群体核苷酸不配对分析图Fig. 4 Mismatch distribution analysis of cytb partial sequences in R. porosa stock of the Rongcheng Bay

表3 软骨鱼类群体cytb部分序列的H和πTab. 3 The haplotype diverisity (H)and nucleotide diversity (π)parameters of cytb partial sequences in elasmobranches
致谢: 本文所分析的孔鳐底拖网样品的采集得到中国科学院海洋研究所线薇微老师、李文龙老师的帮助, 谨此一并致谢。
[1]成庆泰, 郑葆珊. 中国鱼类系统检索[M]. 北京: 科学出版社, 1987: 35- 36.
[2]朱元鼎, 孟庆闻. 中国动物志(圆口纲 软骨鱼纲)[M].北京: 科学出版社, 2001: 385-388.
[3]Stehmann M F W. Proposal of a maturity stages scale for oviparous and viviparous cartilaginous fishes (Pisces, Chondrichthyes)[J]. Arch Fish Mar Res, 2002,50:23-48.
[4]于诗群, 王世党, 郑春波. 孔鳐的生物学特性与养殖技术[J]. 齐鲁渔业, 2005, 22(8): 24-25.
[5]Palumbi S R. Genetic divergence, reproductive isolation, and marine speciation [J]. Annu Rev Ecol Syst,1994, 25: 547-572.
[6]Dudgeon C L, Blower D C, Broderick D, et al. A review of the application of molecular genetics for fisheries management and conservation of sharks and rays[J]. Journal of Fish Biology, 2012, 80: 1789-1843.
[7]Baeck G W, Park C I, Choi H C, et al. Feeding habits of ocellate spot skate,Okamejei kenojei(Müller & Henle,1841), in coastal waters of Taean, Korea [J]. Journal of Applied Ichthyology, 2011, 27: 1079-1085.
[8]赵伟东, 宋丽莉, 黄永松. 孔鳐刺网的设计与装配[J].捕捞技术, 2004, 5: 76.
[9]Boeseman M. Some remarks on the identity of the Japanese raysRaja kenojeiMüller & Henle, 1841, andRaja meerdervoortiiBleeker, 1860 [J]. Zoologische Mededelingen, 1979, 53(25):273-281.
[10]Yoon H K, Jeong D, Chung I H, et al. Rapid species identification of elasmobranch fish (skates and rays)using oligonucleotide microarray [J]. Biochip Journal,2009, 3: 87-96.
[11]Ishiharal H, Mochizuki T, Homma K, et al. Reproductive strategy of the Japanese common skate (Spiny rasp skate)Okameji kenojei[C]. Fowler S L, Reed T M,Dipper F A. Elasmobranch Biodiversity, Conservation and Management: Proceedings of the International Seminar and Workshop, July 1997. Sabah, Malaysia,IUCN, Gland, Switzerland and Cambridge: IUCN SSC Shark Specialist Group, 2002: 236-240.
[12]刘坤, 刘飒. 孔鳐硫酸软骨素的制备[J]. 中国海洋药物, 2004, 9:19-22.
[13]Gray M W. Origin and evolution of mitochondrial DNA[J]. Annual Review of Cell Biology, 1989 (5): 25-50.
[14]Castro A L F. Use of molecular tools on surveys of genetic variation and population structure in three species of sharks [D]. Tampa, FL, USA: University of South Florida, 2009.
[15]Pereyra S, Garcia G, Miller P, et al. Low genetic diversity and population structure of the narrownose shark(Mustelus schmitti)[J]. Fisheries Research, 2009, 106:468-473.
[16]Murray B W, Wang J Y, Yang S C, et al. Mitochondrial cytochrome b variation in sleeper sharks (Squaliformes:Somniosidae)[J]. Marine Biology, 2008, 153:1015-1022.
[17]Schluessel V, Broderick D, Collin SP, et al. Evidence for extensive population structure in the white spotted eagle ray within the Indo-Paci fi c inferred from mitochondrial gene sequences [J]. Journal of Zoology(London), 2010, 281: 46-55.
[18]Chevolot M, Hoarau G, Rijnsdorp A D, et al. Phylogeography and population structure of thornback rays(Raja clavataL., Rajidae)[J]. Molecular Ecology, 2006,15: 3693-3705.
[19]Chevolot M, Wolfs P, P’alsson J, et al. Population structure and historical demography of the thorny skate(Amblyraja radiata, Rajidae)in the North Atlantic [J].Marine Biology, 2007, 151: 1275-1286.
[20]Hochachka P W, Mommsen T. Biochemistry and Molecular Biolodgy of Fishes: Environmental and Ecological Biochemistry [J]. Elsevier Science, 1993, 2:1-38.
[21]Li Wen Hsiung. Molecular Evolution [M]. Sunderland Massachusetts, USA: Sinauer Associates, 1997.
[22]Posada D, Crandall K A. Intraspeci fi c gene genealogies:trees grafting into networks[J]. Trends Ecol Evol, 2001,16: 37-45.
[23]Tajima F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism [J].Genetics,1989, 123: 585-595.
[24]Rogers A R, Harpending H C. Population growth makes waves in the distribution of pairwise genetic differences[J]. Molecular Biology and Evolution, 1992,9: 552-569.
[25]Grant W S, Bowen B W. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation [J]. The Journal of Heredity, 1998, 89: 415-426.
[26]Ward R D. The importance of identifying spatial popu-lation structure in restocking and stock enhancement programmes [J]. Fisheries Research, 2006, 80: 9-18.
[27]牛东红, 刘达博, 陈慧. 缢蛏种群遗传多样性的周年变异分析[J]. 生物技术通报, 2012, 1: 168-171.
[28]Wright S. Evolution and the genetics of population:variability within and among natural population[M]. Chicago: University of Chicago Press, 1978,79-103.
