Hsa-miR-1在人食管癌细胞株中的表达及生物信息学分析
2012-07-31付海龙徐广峰王秀芳赵亚萍杜云翔
蒋 森,付海龙,徐广峰,王秀芳,赵亚萍,杜云翔
(1蚌埠医学院,安徽蚌埠 233000;2中国人民解放军第82医院)
微小RNA(microRNA,miRNA)是一类具有调控功能的非编码小分子RNA[1],参与机体多种生理、病理机制。近年研究结果表明,人类Hsa-miR-1参与细胞生长、分化、凋亡及心脏、内耳发育等生理过程,并与鼻咽癌、肺癌、胃癌等肿瘤发生有关[2~5]。食管鳞状细胞癌(Esophageal squamous cell carcinoma,ESCC)是我国常见的消化道恶性肿瘤,HsamiR-1与ESCC发病关系的研究目前鲜见报道。2011年11月,我们观察了Hsa-miR-1在ESCC细胞株中的表达情况,并通过多个在线数据库、运用多个生物信息学软件对Hsa-miR-1的生物学特征以及功能进行预测分析,旨在为后续的Hsa-miR-1功能研究提供思路。
1 材料与方法
1.1 材料 人食管癌细胞系ECA109、TE-1、KYSE-150和正常食管上皮细胞株Het-1A(均购自中科院上海细胞库);胎牛血清(Gbico公司),1640培养基(Gbico公司),miRNeasy总RNA抽提试剂盒(德国QIAGEN公司),逆转录试剂盒(美国 ABI公司),TaqMan MicroRNA Assay(美国 ABI公司),ABI7500荧光定量PCR仪(美国ABI公司)。
1.2 细胞培养及Hsa-miR-1表达水平检测 取上述KYSE-150及Het-1A细胞分别加入含10%胎牛血清的1640培养基,置于37℃,5%CO2培养箱中培养。按照试剂盒说明书步骤提取细胞总RNA,紫外分光光度仪测定总RNA浓度与纯度,使A260/A280值在1.8~2.0。根据试剂盒说明书操作步骤进行逆转录,采用实时荧光定量-PCR(RT-qPCR)检测Hsa-miR-1水平,每组实验至少重复3次。选择miRNA-U6为内参照,应用2-△CT方法计算目的基因相对表达水平。
1.3 数据来源 利用 miRBase(http://www.mirbase.org/)数据库查找Hsa-miR-1序列,选择三种在线软 件 TargetScan6.1[6](http://www.targetscan.org/)、PicTar[7](http://pictar.mdc-berlin.de/) 和miRanda[8](http://www.microrna.org/) 预测 HsamiR-1的靶基因,取交集,结合 DIANA LAB-Tar-Base5.0(http://diana.cslab.ece.ntua.gr/tarbase/)预测的Hsa-miR-1靶基因,进行后续生物信息学分析。
1.4 数据分析 采用GeneOntology对预测发现的Hsa-miR-1靶基因进行功能富集分类,利用Cytoscape中的插件BINGO[9]实现基因的GO富集分析。采用 DAVID 数据库 (http://david.abcc.ncifcrf.gov/)对Hsa-miR-1预测靶基因进行生物通路富集分析。使用超几何分布计算P值,以P≤0.05为显著性阈值,分别得到相对于背景具有统计意义的高频率注释。
2 结果
2.1 人食管癌细胞中Hsa-miR-1的表达 食管癌细胞株KYSE-150中Hsa-miR-1表达水平显著低于正常食管上皮细胞株Het-1A细胞(P<0.05),见图1。

图1 KYSE-150及Het-1A中Hsa-miR-1表达
2.2 miR-1同源性 成熟体 miR-1的5'端种子序列(Seed sequence)在人(Hsa)、猕猴(mml)、家马(eca)、文昌鱼(bfl)、鸭嘴兽(oan)、安乐蜥(aca)、红鳍东方鲀(fru)、斑胸草雀(tgu)等近100个物种上均显示了很好的同源性(见表1)。
2.3 Hsa-miR-1所在基因组特征 Hsa-miR-1家族由2个miRNAs即Hsa-miR-1-1和Hsa-miR-1-2组成,有两个Hsa-miR-1位点分别编码Hsa-miR-1-1、Hsa-miR-1-2。Hsa-miR-1-1位于人的第20号染色体C20orf166基因初级转录本的第2个内含子区。C20orf166基因包括4个外显子、2个转录产物,长度分别为1 220 bp和426 bp,C20orf166基因开放阅读框(Openreading frame,ORF)大小为 354 bp。MiR-1-2位于人18号染色体上蛋白质编码基因MIB 1的第12个内含子区,MIB1基因组全长为5 356 bp,属于锌指基因家族(Zinc fingers,ZZ-type[ZZZ]),有21个外显子,转录产物长度为9 576 bp,开放阅读框大小为3 021 bp。
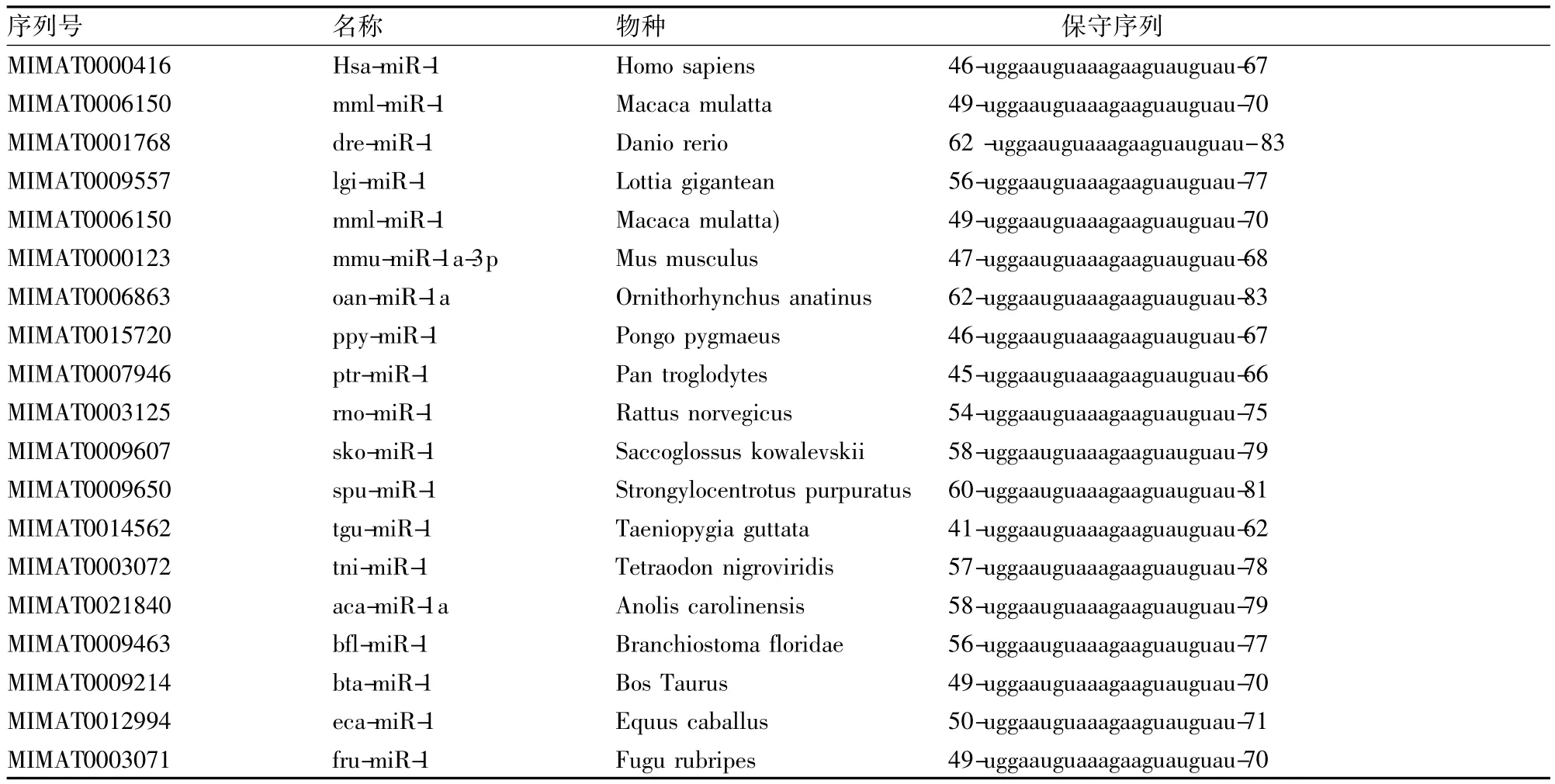
表1 部分物种miR-1成熟序列
2.4 Hsa-miR-1 靶基因预测 DIANA LAB-Tar-Base5.0数据库提供已经有实验数据支持microRNA的确切靶基因,通过检索DIANA LAB-TarBase 5.0数据库发现Hsa-miR-1确认靶标有196个(表略)。应用 TargetScan、PicTar及 miRanda预测 Hsa-miR-1的靶基因,取三者预测结果的交集有106个基因,结合已证实的靶标基因,共267个。
2.5 Hsa-miR-1预测靶基因的GO分析及通路分析对所发现的267个Hsa-miR-1靶基因进行的GeneOntology注释层次分类及富集分析结果发现,miR-1靶基因在GO生物学过程注释中,有5个基因个数超过预测靶基因数50%的富集群,这些富集群主要与生物代谢和调控等过程有关(表略);HsamiR-1靶基因在GO细胞组成分析注释中,共有6个富集群基因个数超过预测靶基因数50%,主要参与细胞和细胞组分的形成(表略)。利用DAVID数据库对267个靶基因进行的生物通路富集分析结果显示,在经典通路数据库KEGG中,miR-1预测的靶基因富集于30个通路中,其中显著的通路有癌症通路(Pathways in cancer)、幽门螺旋杆菌感染的上皮细胞信号(Epithelial cell signaling in Helicobacter pylori infection)、肥厚型心肌病(Hypertrophic cardiomyopathy)、扩张型心肌病(Dilated cardiomyopathy)、硫酸软骨素的合成(Chondroitin sulfate biosynthesis)等7个通路(表略)。
3 讨论
miRNA是近几年分子医学领域中最重要的发现之一,Calin等[10]研究发现,miRNA多定位于肿瘤相关的脆性位点,与肿瘤的发生、发展有密切联系,可能具有致癌基因或抑癌基因作用。最近一些研究证实,miR-1在多种肿瘤中表达异常,具有抑癌基因的作用,与肿瘤细胞增殖、侵袭、迁移和凋亡密切相关,对于肿瘤的诊断、治疗和预后具有重要意义[11~13]。本研究结果显示,与正常食管上皮细胞相比,ESCC细胞中Hsa-miR-1表达明显下调,提示Hsa-miR-1可能参与ESCC的发病机制,具有抑癌基因的作用。
miRBase上的数据显示,至2012年6月,在植物、病毒、动物和人体内发现的miRNA已有18 000多种,其中人类miRNA有2 000多种。已有研究表明,一条miRNA通常具有调控多个基因的功能,而同一基因也可能同时受多个miRNA时空特异的精密调控,此种调控方式导致miRNA的功能研究颇具复杂性,仅依靠实验手段研究miRNA已变得相当困难。生物信息学在miRNA研究中扮演着越来越重要的角色,其可通过对海量和复杂信息进行分析和处理而为下一步实验提供指导,其在miRNA研究中的作用主要包括识别miRNA、对miRNA基因注释、预测miRNA靶基因及分析miRNA芯片数据、miRNA功能、miRNA关联疾病等。为更进一步了解Hsa-miR-1的生物学特征和功能,本研究分析了各物种miR-1的序列,发现成熟体 miR-1的5'端种子序列具有高度保守性。miRNA主要在转录后水平调控基因的表达,其5'端2~8个核苷酸被称为种子序列,对于其功能的正常发挥至关重要,种子序列主要通过与靶mRNA 3'端区域互补配对识别靶基因,负调控靶基因的表达[14]。在同源性miRNA中,这段序列通常是保守的,本研究中的序列比对分析也证实了此点。Hsa-miR-1在物种进化过程中的高度保守性,提示其在生命过程中可能发挥了重要功能。
已有研究发现,miRNA多位于肿瘤相关的染色体区域[15],Hsa-miR-1 所处 染 色体也出 现 断点[16~18]。本研究中靶基因预测出现与肿瘤相关的基因富集群,提示其可能参与了人类肿瘤的发生;ESCC细胞株Hsa-miR-1表达水平显著下调的实验结果亦进一步证实了生物信息学的推测。由于单个miRNA靶基因预测软件具有一定局限性,且每个预测软件的结果也不尽相同,本研究同时采用多个数据库预测Hsa-miR-1靶基因结果、取交集,并结合实验证实的靶基因进行分析,进一步提高了预测结果的准确性。本研究中GO分析显示,Hsa-miR-1的靶基因可能参与细胞形成过程的代谢和调控,通路分析表明其靶基因集合显著富集在癌症通路、与癌症发生相关的信号通路以及心肌通路中。提示HsamiR-1可能参与恶性肿瘤及心脏疾病的发生机制。
总之,Hsa-miR-1可能参与了食管癌的发病机制,其预测靶基因集合富集于多个生物学过程且显著富集于癌症通路;此为后续Hsa-miR-1生物学功能的研究奠定了基础。
[1]Ambros V.The functions of animal microRNAs[J].Nature,2004,431(7006):350-355.
[2]Wu CD,Kuo YS,Wu HC,et al.MicroRNA-1 induces apoptosis by targeting prothymosin alpha in nasopharyngeal carcinoma cells[J].J Biomed Sci,2011,18(1):80.
[3]Nasser MW,Datta J,Nuovo G,et al.Down-regulation of micro-RNA-1(miR-1)in lung cancer.Suppression of tumorigenic property of lung cancer cells and their sensitization to doxorubicin induced apoptosis by miR-1[J].J Biol Chem,2008,283(48):33394-33405.
[4]Liu R,Zhang C,Hu Z,et al.A five-microRNA signature identified from genome-wide serum microRNA expression profiling serves as a fingerprint for gastric cancer diagnosis[J].Eur J Cancer,2011,47(5):784-791.
[5]Nohata N,Hanazawa T,Kikkawa N,et al.Identification of novel molecular targets regulated by tumor suppressive miR-1/miR-133a in maxillary sinus squamous cell carcinoma[J].Int J Oncol,2011,39(5):1099-1107.
[6]Lewis BP,Shih IH,Jones-Rhoades MW,et al.Prediction of mammalian microRNA targets[J].Cell,2003,115(7):787-798.
[7]Krek A,Grün D,Poy MN,et al.Combinatorial microRNA target predictions[J].Nat Genet,2005,37(5):495-500.
[8]John B,Enright AJ,Aravin A,et al.Human microRNA targets[J].PLoS Biol,2004,2(11):e363.
[9]Maere S,Heymans K,Kuiper M.BiNGO:a Cytoscape plugin to assess overrepresentation of gene ontology categories in biological networks[J].Bioinformatics,2005,21(16):3448-3449.
[10]Calin GA,Sevignani C,Dumitru CD,et al.Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers[J].Proc Natl Acad Sci U S A,2004,101(9):2999-3004.
[11]Leone V,D'Angelo D,Rubio I,et al.MiR-1 is a tumor suppressor in thyroid carcinogenesis targeting CCND2,CXCR4,and SDF-1alpha[J].J Clin Endocrinol Metab,2011,96(9):E1388-E1398.
[12]Wang F,Song G,Liu M,et al.miRNA-1 targets fibronectin1 and suppresses the migration and invasion of the HEp2 laryngeal squamous carcinoma cell line[J].FEBS Lett,2011,585(20):3263-3269.
[13]Nohata N,Sone Y,Hanazawa T,et al.miR-1 as a tumor suppres-sive microRNA targeting TAGLN2 in head and neck squamous cell carcinoma[J].Oncotarget,2011,2(1-2):29-42.
[14]Bartel DP.MicroRNAs:genomics,biogenesis,mechanism,and function[J].Cell,2004,116:281-297.
[15]Chen C,Ridzon DA,Broomer AJ,et al.Real-time quantification of microRNAs by stem-loop RT-PCR[J].Nucleic Acids Res,2005,33(20):e179.
[16]Hu Y,Lam KY,Wan TS,et al.Establishment and characterization of HKESC-1,a new cancer cell line from human esophageal squamous cell carcinoma[J].Cancer Genet Cytogenet,2000,118(2):112-120.
[17]Hu YC,Lam KY,Law SY,et al.Establishment,characterization,karyotyping,and comparative genomic hybridization analysis of HKESC-2 and HKESC-3:two newly established human esophageal squamous cell carcinoma cell lines[J].Cancer Genet Cytogenet,2002,135(2):120-127.
[18]Thye T,Vannberg FO,Wong SH,et al.Genome-wide association analyses identifies a susceptibility locus for tuberculosis on chromosome 18q11.2[J].Nat Genet,2010,42(9):739-741.
