无启动子转化策略用于分离组织特异性启动子的研究
2012-07-27吴旭乾何光源
吴旭乾,何光源
(1.武汉软件工程职业学院环境与生化工程系,湖北 武汉 430205;2.华中科技大学生命学院,湖北 武汉 430074)
启动子按作用方式可分为广谱启动子和组织特异性启动子。近年来,人们相继分离、鉴定了许多特异表达基因的启动子,如玉米醇溶蛋白基因B4 启动子、水稻胚乳特异性谷蛋白基因启动子、大豆7S 种子特异性启动子[1]、水稻花药特异表达基因启动子[2]、烟草花药特异表达基因启动子[3]等,但麦类作物的此类启动子报道甚少。
tritordeum[4]为硬粒小麦(Triticumdurum)和大麦(Hordeumchilense)的杂交种,其基因组含有小麦的AA、BB和大麦的HchHch。供试材料转基因tritordeum中使用的转化质粒载体的构建如下:首先从pAct1-DGUS中用双酶切BamHⅠ/XbaⅠ分离出细菌uidA基因和nos终止子片段;随后引入到pBluescriptⅡ骨架形成质粒pPLGUS;再从pAHCI7中以酶切Bg/Ⅱ/BamHⅠ获得含有来源于玉米的40 bp的外显子序列和ubiquitin-1内含子序列,已知该序列的插入会增强启动子的驱动,有利于检测出所标定的弱启动子序列,并将其克隆到pPLGUS的BamHⅠ位点,最后形成转化载体——pPLubiGUS,如图1所示[5]。
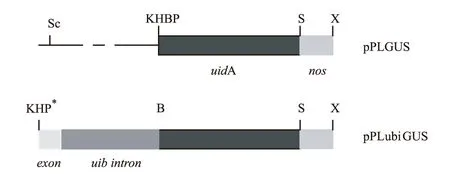
图1 含无启动子标记载体的部分结构
基因枪法是小麦遗传转化中应用最为广泛的一种方法,以植物组织、愈伤组织、悬浮细胞系、甚至直接以胚作为转受体,具有无宿主限制、靶受体类型广泛、可控度高、操作简便快速等优点。应用基因枪转化技术将pPLubiGUS载体转化到受体材料tritordeum中,得到了一批转基因材料,为无启动子转化策略用于分离组织特异性启动子的研究提供了可能性。
1 实验
1.1 材料、试剂与仪器
生长期的转基因植株:以tritordeum为受体的转基因材料的后代经过种子的发芽处理、播种、日常看护等过程得到。
NLS(Nuclei lysis solution),PPS(Protein precipitate solution),10×RNase缓冲溶液(10 mmol·L-1Tris,15 mmol·L-1NaCl,pH值7.5),RNase溶液(10 mg·mL-1),异丙醇,100×TE缓冲溶液(1 mol·L-1Tris-HCl,0.1 mol·L-1EDTA),液氮,10×TBE缓冲溶液,EB溶液,琼脂糖凝胶,6×载样缓冲溶液,λDNA Marker,X-Gluc缓冲溶液,Triton X-100,70%~100%梯度乙醇等。
T1 Thermocycler;稳压稳流电泳仪;精密pH计;HH-W21-420型多用恒温水箱;Gel-doc型凝胶成像仪,BIO-RAD公司;红外线加热微波炉;303AS-2型电热培养箱;96孔微滴定板;XSJ-2型光学显微镜;数码摄影机。
1.2 方法
1.2.1 叶片总DNA的提取
叶片总DNA的提取参照文献[4]。
1.2.2 PCR扩增uidA基因
(1)引物设计
基于uidA序列设计引物:
上游P1:5′-AGTGTACGTATCACCGTTTGTGTGAAC-3′
下游P2:5′-ATCGCCGCTTTGGACATACCAT-CCGTA-3′
分别起始于uidA序列的376、1431位点。
(2)PCR反应体系
DNA模板1.0 μL、引物P1和P2各0.2 μL、AB1.1 Master mixture 9.7 μL,混匀。
(3)PCR运行程序
94 ℃变性4 min;60 ℃退火30 s,72 ℃延伸2 min,94 ℃变性30 s,30个循环;60 ℃退火30 s;72 ℃延伸8 min;4 ℃ 保温。
1.2.3 琼脂糖凝胶电泳
根据欲分离DNA片断大小配制适宜浓度的琼脂糖溶液,检测总DNA时用0.6%凝胶,检测PCR产物时用1.0%凝胶;待熔化的凝胶稍冷却后加入EB溶液,使终浓度达到0.5 μg·mL-1,轻轻旋转以混合均匀;电泳结束,用凝胶成像系统检测EB染色的凝胶。
1.2.4 GUS组织化学检测
由于uidA基因的表达产物是β-葡糖醛酸酶,若以5-溴-4-氯-3-吲哚-β-D-葡萄糖苷酸(X-Gluc)为反应底物,在酶的活性部位会产生蓝色沉淀,显微观察即可分析uidA表达的组织特异性。
取材部位:三叶期(叶尖、叶中、叶基、叶鞘及根尖)、3~4个分蘖期(叶尖、叶中、叶基、叶鞘及根尖)、刚抽穗之前两节点及中间部分、幼穗、发育早期的花药及花粉粒、成熟期花药及花粉粒。
检测步骤:将72 μL X-Gluc缓冲溶液加入到微孔板中,再加入0.1 BV的1% Triton X-100;将植物材料切成小片段,用70%乙醇漂洗后再用无菌双蒸水清洗,置于含X-Gluc的微孔中;用粘性盖或Nescofilm密封微孔板,置于潮湿间内,防止小体积溶液蒸发干燥;37 ℃过夜,然后于室温放置2~3 d(37 ℃是酶反应的最适温度,产生蓝色沉淀,而室温放置会使颜色加深);若材料呈深绿色,蓝色显色反应将难以检测。可用70%~100%梯度乙醇代替X-Gluc缓冲溶液去除叶绿素(根据颜色深浅可多次重复),用1 mol·L-1Tris-HCl(pH值9.5)代替乙醇置4 ℃长时间保存;记录蓝色显色反应情况。
2 结果与讨论
2.1 琼脂糖凝胶电泳结果
提取出的叶片总DNA用0.6%的琼脂糖凝胶电泳检测提取效果,结果见图2。
2.2 PCR扩增结果(图3)

1.DNA Marker Ⅱ 2.阳性对照B73-6-1 3~11.供试样品
两引物间的物理距离为1055 bp,uidA基因的PCR结果显阳性的单株,其特异性的扩增条带应与此相当。由图3可看出,4#、7#供试样品含有uidA基因。
2.3 GUS组织化学检测结果
2.3.1 阳性对照材料中Gus的表达情况
阳性对照材料B73-6-1(uidA由广谱型启动子CaMV35S驱动表达)中外源Gus的表达结果见图4。

a~f:根,叶,叶基,发育早期的花药,花药,花粉粒
由图4可看出,阳性对照材料的各组织部位均出现了uidA的强烈表达。
2.3.2 供试材料中Gus的表达情况
经PCR所筛选出的转基因tritordeum中外源Gus的表达结果见图5。
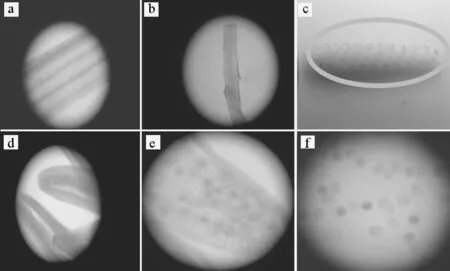
a~f:叶片,根,幼穗,发育早期的花药,花药,花粉粒
由图5可看出,除了幼穗中花药原基部位有β-葡糖醛酸酶活性外,其它组织或器官中均未发现有Gus表达的现象。
2.4 讨论
应用已知的基因序列和未知基因进行分离,是目前常用的启动子分离策略。第一种方法已经应用了很长时间,主要是利用已知基因序列制得的探针来搜寻基因文库,或者是从已知基因序列的5′端进行“步移技术”(Walking)。第二种是基因标记(Tagging)探索技术,通过将无启动子的标记基因(如Gus)导入受体细胞来实现的,标记调节序列的效果通过检测转基因植物中标记基因的表达情况来确定[6],这种方法已被成功地应用于拟南芥、土豆和烟草等植物的调节序列的标记[7]。有时,在构建含无启动子uidA基因转化载体时,有意识地在紧靠uidA位点上游插入内含子序列,已知该序列有增强基因表达的效应,此内含子的插入,可增加那些被标定的弱的内源性启动子的检出机会[8]。
3 结论
通过研究外源uidA基因在体内表达的时空特异性,利用PCR、GUS组织化学检测等技术,从以tritordeum为受体、经无启动子uidA转化策略得到的转基因供试材料中成功筛选出了外源uidA基因在花药原基特异表达的单株。本研究结果表明,在进行遗传转化时,无启动子的uidA插入到tritordeum某一内源性调控序列的后面,能够引起uidA的表达,并且表达部位仅限于花药原基部位。可以确定,该序列是花药原基特异表达基因的启动子。
应用无启动子转化策略,为标定内源性启动子提供了一种有效的手段,有着广阔的应用前景。
[1] 朱亚兰,姚伟,窦建华,等.种子特异表达启动子的克隆分析及其植物表达载体的构建[J].华北农学报,2007,22(2):6-10.
[2] 张晓国,刘玉乐,康良仪,等.水稻花药特异表达基因启动子的扩增及克隆[J].武汉大学学报(自然科学版),1997,43(4):480-484.
[3] 彭仁旺,周雪荣,周奕华,等.烟草花药特异表达基因启动子的克隆及序列分析[J].生物工程学报,1996,12(3):247-250.
[4] 何光源,吴旭乾,涂知明,等.转基因tritordeum的遗传分析[J].华中科技大学学报(自然科学版),2005,33(8):94-96.
[5] Salgueiro S,Matthes M,Gil J,et al.Insertional tagging of regulatory sequences in tritordeum,a hexaploid cereal species[J].Theor Appl Genet,2002,104(6-7):916-925.
[6] Springer P S.Gene traps:Tools for plant development and genomics[J].The Plant Cell,2000,12(7):1007-1020.
[7] Kertbundit S,de Greve H,Deboeck F,et al.Invivorandomβ-glucuronidase gene fusions inArabidopsisthaliana[J].Proc Natl Acad Sci USA,1991,88(12):5212-5216.
[8] McFadden E S,Sears E R.The origin ofTriticumspeltaand its free-threshing hexaploid relatives[J].J Hered,1946,37(3):81-89.
