DiversiLab系统沙门氏菌分子分型评价
2012-06-01许龙岩袁慕云林文丽
许龙岩,袁慕云,林文丽,张 旺,焦 红
(广东出入境检验检疫局技术中心食品实验室, 广东 广州 510623)
DiversiLab系统沙门氏菌分子分型评价
许龙岩,袁慕云,林文丽,张 旺,焦 红
(广东出入境检验检疫局技术中心食品实验室, 广东 广州 510623)
目的:评价DiversiLab(DVL)系统对沙门氏菌(SA)的分型能力。方法:用rep-PCR技术为原理的DVL系统对进出口食品中分离的44株SA进行分子分型,并与脉冲场凝胶电泳(PFGE)结果比较,探讨DVL对SA的种群分类能力和分辨率。结果:44株SA经rep-PCR分型分成22个群,分离自不同国家、不同食品中的相同血清型SA分布在同一个群,而且菌株之间相似性非常高。在rep-PCR结果中分在同一个群中的SA,在PFGE结果中除SA2和SA15外,其他菌株之间相关性较低。结论:DVL在SA的种群的分类能力上优于PFGE,但分辨率低于PFGE。
沙门氏菌;DiversiLab系统;rep-PCR;PFGE;分子分型
基因外重复回文序列(repetitive ext ragenic palindrome,rep)在细菌基因组中存在着菌株、种、属水平上的分布和拷贝数量的差异,而序列本身在进化过程中又具有较强的保守性,非常适合作为PCR引物的扩增对象。在rep的中心有一段保守性很高的重复序列,根据这段重复序列设计引物,扩增两段rep之间的片段,发展出了rep-PCR技术[1]。DiversiLab(DVL)是以rep-PCR技术为原理的自动化的分子分型系统。本研究用DVL对食品中分离的沙门氏菌进行rep-PCR分型,并与脉冲场凝胶电泳(PFGE)结果相比较,探讨DVL对SA的分型能力,为DVL应用于SA的溯源工作提供参考数据。
1 材料与方法
1.1 菌株
进出口食品中分离的SA 44株,菌株信息见表1。上述菌株为广东出入境检验检疫局技术中心食品实验室和中国检验检疫科学研究院食品安全研究所保存菌株,所有菌株均经过API 20E生化鉴定确定为SA。PFGE相对分子质量标准用布伦登卢普(Braenderup)沙门氏菌H9812,由广东省疾病预防控制中心赠送。
1.2 试剂与仪器
API 20E、rep-PCR试剂盒、微流体芯片 法国梅里埃公司;限制性内切酶Xba I 美国Promega公司;蛋白酶K 德国Merck公司;SeaKem Gold琼脂糖、SLS(Sodium lauroyl Sarcosine)、SDS、EDTA 美国Sigma公司。
Agilent Bioanalyzer 2100型DVL分型系统 美国Agilent公司;PTC-200型PCR仪、CHEF MAPPER脉冲场电泳仪、Gel Doc XR凝胶成像系统 美国Bio-Rad公司。
1.3 方法

图1 44株沙门氏菌rep-PCR分子分型树状图和矩阵图Fig.1 Dendrogram and matrix diagram constructed by rep-PCR molecular typing

表1 44株沙门氏菌菌株信息Table 1 Details of 44 Salmonella isolates investigated in this study
1.3.1DNA提取
刮取血琼脂平板上分离纯化的SA菌落,使用DVL配套核酸提取试剂盒,按照说明书推荐步骤提取DNA,DNA质量浓度控制在25~50ng/μL。
1.3.2rep-PCR分型
以提取的SA DNA为模版,使用DVL 配套的rep-PCR试剂盒,按照说明书进行PCR扩增,PCR循环参数为:94℃、2min;94℃、30s,50℃、30s,70℃、90s,35个循环;72℃、3min,反应体积为25μL。
PCR扩增产物注入微流体芯片放入DVL系统进行电泳,DVL自动生成分析报告,包括聚类分析树状图、凝胶图像虚拟图、矩阵图、菌株之间相似度。DVL根据Pearson相关系数确定菌株间的距离矩阵,用非加权配对算术平均法(Upgma法)建立树状图。
1.3.3rep-PCR分型与PFGE分型比较
44株SA中选24株进行PFGE,PFGE方法按文献[2]进行。限制性内切酶用XbaⅠ。产生的电泳图用BioNumerics软件构建聚类分析树状图。
2 结果与分析
2.144 株SA rep-PCR分型结果
44株SA 经rep-PCR分型分成22个群,由图1可见,分离自不同国家、不同食品中的相同血清型的SA分布在同一个群,而且菌株之间相似性非常高。鼠伤寒沙门氏菌SA4、SA6、SA27、SA31、SA36分别从欧洲冻猪肚、广东冻鸡、欧洲猪舌、丹麦冻猪后脚、广东罗非鱼中分离,5株SA在聚类图上分布在第4群,而且菌株之间的相似度在97%~99%。不同时间从欧洲冻猪肚中分离的德尔卑沙门氏菌SA2、SA5、SA29 和广东冻猪肚中分离的SA15分布在第2群,菌株之间相似度在95.1%~97.9%。相同的现象在其他血清型SA中也发现,乌普沙拉沙门氏菌SA10、SA11、SA13分布在第6群;阿贡纳沙门氏菌SA1、SA16、 SA35分在第1群,菌株之间相似度97.8%~98.5%;姆班达卡沙门氏菌SA22和SA23分布在第5群,伊斯坦布尔沙门氏菌SA18、SA38分布在第20群。
2.2SA rep-PCR与PFGE分型结果比较
24株SA经PFGE,根据电泳产生的条带位置和数量的不同,共分为22个PFGE型,带型较分散。由图2可见,德尔卑沙门氏菌SA15、SA2和乌普萨拉沙门氏菌SA13之间的相似度达到100%,3株SA有相同的PFGE型,相似度达到100%。分析PFGE型相同而且同源性100% 的SA13、SA2、SA15菌株在rep-PCR图上的分布,发现3株菌株在rep-PCR树状图上分布在不同的群中。德尔卑沙门氏菌SA2、SA15在第2群,乌普萨拉沙门氏菌SA13在第6群,而且在树状图的空间距离上与前两株菌相隔较远,表现出不相关性。这可能是SA13、SA15、SA2对限制性内切酶XbaⅠ有相同的酶切位点,3株SA有相同的PFGE型但有不同的rep-PCR型。分析鼠伤寒沙门氏菌在PFGE和rep-PCR中的分布,SA4、SA6、SA27、SA36在rep-PCR图上均分布在第4群,SA4和SA6菌株之间同源性为97.8%,SA27和SA36同源性达到98.3%,按rep-PCR判断标准SA4和SA6、SA27和SA36菌株间是不可分辨的关系,但PFGE结果,SA4和SA6之间是紧密相关菌株,SA27和SA36之间是不相关菌株。相同的现象在阿贡纳沙门氏菌SA16和SA1之间也发现,两株菌株在rep-PCR图上分在第1群,菌株之间相似度98.5%,是不可分辨的菌株,但PFGE结果两株菌株的相似度83.3%,相关性较弱。
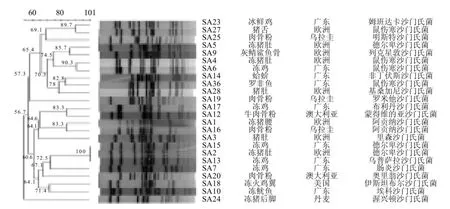
图2 XbaⅠ酶切24株沙门氏菌PFGE聚类分析树状图Fig.2 Dendrogram constructed by PFGE cluster analysis based on Xba I digestion
3 讨 论
rep-PCR已经被认为是一种细菌分型的有效方法,rep-PCR已经被商业化成为一个自动化模式,即DiversiLab系统[3]。Healy等[3]在一次对爆发和非爆发相关的不同血清型脑膜炎奈瑟菌进行的分析中发现,DiversiLab系统表现出了极其优秀的血清型和物种水平鉴别能力。Cangelosi 等[4]用DiversiLab系统对结核分枝杆菌和鸟型分枝杆菌进行分型时,产生其他方法不能分型的分枝杆菌的指纹,本研究用DVL系统对不同来源的SA进行rep-PCR分型,结果相同血清型的菌株分布在同一个群。5株鼠伤寒沙门氏菌SA4、SA6、SA27、SA31、SA36,4株德尔卑沙门氏菌SA2、SA5、SA29、SA15,乌普沙拉沙门氏菌SA10、SA11、SA13,阿贡纳沙门氏菌SA1、SA16、SA35,姆班达卡沙门氏菌SA22、SA23,均分别分布在同一个群中。值得一提的是,与德尔卑沙门氏菌SA2、SA15 PFGE型别完全相同的乌普萨拉沙门氏菌SA13,在rep-PCR树状图上并非与上述两株菌株分在一个群,而是与相同血清型的SA10、SA11分在同一个群。DVL对SA种群分析时,是基于分类群之间的亲缘关系进行分离,在SA种群的分类方面表现出较强的能力。
rep-PCR与PFGE分型结果比较发现,在rep-PCR树状图中分在同一个群中的相同血清型SA在PFGE图谱上并非分在同一个群,而且树状图中相隔较远、相似性较低。在rep-PCR图谱判断标准上,DVL的厂家建议菌株之间相似性大于97%、没有条带的差异,判断为不可辨别的菌株;相似性大于95%、有1~2条不同的条带,判断为相似菌株;相似性小于95%、有多条不同的条带,则判断为不同菌株(DVL的rep-PCR图谱不会给出100%的相似性)。曲燕燕等[5]按上述判断标准用DVL对鲍曼不动杆菌进行分子分型,Healy等[6]也用上述标准对阪崎肠杆菌进行分群。本研究rep-PCR图谱中分在同一群中的相同血清型SA之间相似性均在97%以上,按照DVL判断标准这些菌株之间是不可分辨的。但在PFGE结果中观察,只有德尔卑沙门氏菌SA2、SA15是完全相同的菌株外,阿贡纳沙门氏菌SA16和SA1、鼠伤寒沙门氏菌SA27和SA36、德尔卑沙门氏菌SA2和SA5,在树状图上相隔较远,彼此之间是不相关的菌株。这一观察结果表明,rep-PCR在进行SA种群分类时比PFGE更优胜,但对同种菌株的分型分辨率上PFGE更优于rep-PCR。在实验操作方面,DVL是自动化的rep-PCR分型系统,提供标准化的试剂和方法,对实验人员的操作技能要求没有PFGE高[7-8]。在分型时间上DVL系统可以在4h内自动化分析13个样品,包括获得聚类分析树状图、凝胶图像虚拟图、矩阵图等报告,而PFGE需要3~5d时间[9]。总之,在细菌的分型方法中,DVL系统是一种快速、简便的分型方法,对SA的种群分类能力和分辨率也比较高,作为SA快速分型和溯源工具具有潜在的实用性。
[1]VERSALOVIC J, KOEUTH T, LUPSKI J R. Distribution of repetitive DNA sequences in eubacteria and application to fingerprinting of bacterial genomes[J]. Nucleic Acids Res, 1991, 19(24): 6823-6831.
[2]CDC. Standardized laboratory protocol for molecular subtyping of Escherichia coli O157:H7, non-typhoidal Salmonella serotypes,and Shigelloses sonnei by Pulsed Field Gel Electrophoresis (PFGE)[EB/ OL]. [2011-03-01]. http://www. cdc.gov/pulsenet/protocols/ecoli-salmonella-shigella-protocols. pdf.
[3]HEALY M, HUONG J, BITTNER T, et al. Microbial DNA typing by automated repetitive-sequence-based PCR[J]. Journal of Clinical Microbiology, 2005, 43(1): 199-207.
[4]CANGELOSI G A, FREEMAN R J, LEWIS K N, et al. Evaluation of a high-throughput repetitive-sequence-based PCR system for DNA fingerprinting of Mycobacterium tuberculosis and Mycobacterium avium complex strains[J]. J Clin Microbiol, 2004, 42: 2685-2693.
[5]曲燕燕, 王文飞, 周华, 等. DiversiLab系统鲍曼不动杆菌基因分型可行性研究[J]. 中华检验医学杂志, 2010, 33(5): 425-429.
[6]HEALY B, MULLANE N, COLLIN V, et al. Evaluation of an automated repetitive sequence: based PCR system for subtyping Enterobacter sakazakii[J]. Journal of Food Protection, 2008, 71(7): 1372-1378.
[7]马俊英, 刘健华, 陈杖榴, 等. 细菌分型的分子生物学技术研究进展[J]. 中国兽医科学, 2007, 37(10): 914-918.
[8]王丽丽, 徐建国. 脉冲场凝胶电泳技术( PFGE)在分子分型中的应用现状[J]. 疾病监测, 2006, 21(5): 276-279.
[9]ROSS T L, MERZ W G, FARKOSH M. Comparison of an automated repetitive sequence: based PCR microbial typing system to pulsed-field gel electrophoresis for analysis of outbreaks of methicillin-resistant Staphylococcus aureus[J]. Journal of Clinical Microbiology, 2005, 43(11): 5642-5647.
Molecular Typing Evaluation of Salmonella in Diversilab System
XU Long-yan,YUAN Mu-yun,LIN Wen-li,ZHANG Wang,JIAO Hong
(Food Laboratory of Technology Center, Guangdong Entry-Exit Inspection and Quarantine Bureau, Guangzhou 510623, China)
Objective: To evaluate the typing performance of DiversiLab system (DVL) for Salmonella. Method: A DVL system, which is based on rep-PCR, was used to type 44 Salmonella isolates from exported and imported foods and compared with pulsed-field gel electrophoresis (PFGE). The Salmonella isolates were typed by rep-PCR into 22 groups. The same serotypes of Salmonella isolates from different countries and foods were distributed in the same group with a high similarity. The PFGE results obtained for Salmonella isolates assigned to the same group using rep-PCR indicated a low similarity among all other isolates except No. 2 and No. 15. Conclusion: DVL shows better typing ability but lower resolution for Salmonella than PFGE.
Salmonella;DiversiLab system;rep-PCR;PFGE;molecular typing
Q939
A
1002-6630(2012)03-0203-04
2011-03-27
广东省科技计划项目(2009B0600013);广东出入境检验检疫局科研项目(2009GDK35)
许龙岩(1970—),男,高级工程师,硕士,研究方向为食源性病原菌分子检测。E-mail:xlyooo@sohu.com
