甲苯生物降解的关键酶基因克隆及表达
2012-01-13杨凯缪晓燕陈琼斯章婷婷叶薇吕建新
杨凯,缪晓燕,陈琼斯,章婷婷,叶薇,吕建新
(温州医学院 检验医学院,浙江 温州 325035)
甲苯生物降解的关键酶基因克隆及表达
杨凯,缪晓燕,陈琼斯,章婷婷,叶薇,吕建新
(温州医学院 检验医学院,浙江 温州 325035)
目的:克隆、表达恶臭假单胞菌生物降解甲苯的关键酶基因。方法:恶臭假单胞菌WZ-1在含甲苯的MS培养基中培养以鉴定其降解甲苯的能力。设计引物扩增甲苯生物降解的关键酶——甲苯双加氧酶和邻苯二酚1,2双加氧酶基因,PCR扩增目的片段后进行T-A克隆,筛选阳性克隆并鉴定。构建甲苯双加氧酶-pET28a进行体外诱导表达。结果:恶臭假单胞菌WZ-1在含甲苯的MS培养基的生长曲线证明其具有降解甲苯的能力;通过重组技术获得阳性克隆,并以酶切、DNA测序进行鉴定;IPTG诱导后获得重组甲苯双加氧酶的高表达。结论:成功获得了用于降解甲苯的甲苯双加氧酶基因和邻苯二酚1,2双加氧酶基因,并在体外诱导表达了目的蛋白,为后续利用模式生物对苯系物的生物降解建立了实验基础。
恶臭假单胞菌;甲苯;克隆;原核表达
苯(benzene)、甲苯(toluene)、乙苯(ethylbenzene)和二甲苯(xylene)等单环芳烃化合物,缩写为BTEX,是一类重要的环境污染物,广泛来源于石油冶炼、农药生产、印染化工等工农业的生产、运输等各个过程。由于其化学性质稳定,且对人体的中枢神经具有急性危害作用,其一旦进入环境,不会自行消失,对环境和人类健康造成极大威胁。目前,国内外对BTEX的研究主要集中在两个方面:一是环境污染的降解研究,这部分多数以BTEX泄漏对地下水、土壤造成的污染为研究对象,进而研究生物净化修复技术;二是BTEX的高灵敏度分析方法技术研究[1-5]。在BTX的生物降解研究中,常利用质粒载体构建生物反应器,但由于质粒的遗传具有相对的独立性,经过几代的培养,很可能丢失,导致生物反应器失效;进入环境的质粒也可能污染其他的菌种,易造成潜在的环境风险性;而直接从环境中筛选的分解菌对生长环境要求高,适应性差,生长增殖速度慢,不利于工业上的大规模应用。因此将降解酶基因整合至无降解能力但易生长的细菌(如大肠杆菌)染色体,构建降解BTX的生物传感器,以此来实现对污染物的降解,无疑是一种安全、稳定、高性价比的解决方案。
本研究以具有降解甲苯的菌株——恶臭假单胞菌(Pseudomonas putida)为研究对象,对参与降解甲苯的生物酶基因——甲苯双加氧酶(toluene dioxygenase,TOL)基因和邻苯二酚1,2双加氧酶(catechol 1,2-dioxygenase,CAT)基因进行分子克隆、鉴定后进行体外诱导表达,为后续构建降解甲苯的生物反应器提供前期准备。
1 材料和方法
1.1 材料
1.1.1 菌株:恶臭假单胞菌(命名为恶臭假单胞菌WZ-1,由本实验室提供),分离于环境水源并已经初步生化鉴定;大肠杆菌DH5a,由本实验室提供。
1.1.2 试剂:Taq DNA聚合酶、T4 DNA连接酶、DNA Marker、PCR反应试剂盒、PCR产物纯化试剂盒、pMD 19-T Vector、胶回收试剂盒、质粒提取试剂盒、限制性内切酶等均购自宝生物工程(大连)有限公司。
1.1.3 培养基:LB固体培养基、LB液体培养基、无机盐培养基(MS培养基)。
1.1.4 引物:用于扩增TOL基因的上游引物T1:5’-ATGATTGATTCAGCCAACAGAGCCG-3’,下游引物T2:5’-CTAGAAGAAGAAGAAACTGAGGTTATTGGCCAG-3’,目的片段长度为564 bp。为构建TOL-pET28a重组质粒,分别在引物T2 5’端加Nde I酶切位点及保护性碱基,在引物T1 5’端加Xho I酶切位点(CTCGAG)及保护性碱基。扩增CAT基因的上游引物C1:5’-ATGACCGT GAAAATTTCCCACAC-3’,下游引物C2:5’-TCAGCCCCTCC TGCAACGC-3’,目的片段长度936 bp。
1.2 方法
1.2.1 恶臭假单胞菌WZ-1降解甲苯能力的初步鉴定:配置MS培养基:①配制缓冲体系和铁剂:KH2PO40.1809 g、K2HPO40.0339 g、(NH4)2SO40.1650 g,加双蒸水充分溶解并定容至100 mL;准确称取FeSO4(NH4)2SO4·6H2O 0.0784 g至50 mL离心管中,加水充分溶解后定容至20 mL;取1 ml配置的铁剂加到100 mL的缓冲体系中,摇匀后高压灭菌待用;②配置微量成分:准确取CaCl20.03 g、MgSO40.2 g、MnSO4·H2O 0.06 g分别置于三个50 mL离心管中,并分别加水充分溶解后定容至20 mL,上述三管溶液经高压灭菌后备用;③各取1 mL高压灭菌后的CaCl2、MgSO4、MnSO4·H2O在无菌条件下加到已经高压的缓冲体系中,配制成MS培养基。
恶臭假单胞菌30 ℃ 200 r/min的摇床过夜培养于LB液体培养基中,次日分别取20μL的恶臭假单胞菌菌液于含100、200、400、800μL甲苯的200 mL MS液体培养基中,30 ℃ 200 r/min的摇床上培养24 h,并记录24 h内MS培养基恶臭假单胞菌液OD600值。
1.2.2 恶臭假单胞菌WZ-1 TOL基因和CAT基因的克隆:菌株基因组DNA的制备:菌株于LB固体培养基上30 ℃培养过夜,挑取单个菌落重悬于100μL双蒸水中,加热煮沸后,降温,于4 ℃冰箱中保存,以备用。
PCR扩增(20μL):10×Ex Taq Buffer(含MgCl2)2μL、dNTPs Mixture(各2.5 mmol/L)2 μL、Ex Taq酶(5 U/μL)0.1μL、菌株基因组DNA 1μL、引物(10μmol/L)0.2μL,灭菌水补足20 μL。扩增条件为94 ℃ 5 min预变性后,94 ℃ 30 s、52 ℃ 45 s、72 ℃ 1 min共 35个循环,72 ℃延伸6 min。PCR产物进行1%琼脂糖凝胶电泳检测并切胶回收纯化。
PCR产物克隆:目的条带经电泳切胶回收后与pMD19-T质粒16 ℃连接过夜。连接产物转化至感受态大肠杆菌DH5α,用LB/Amp/X-Gal/IPTG培养基进行蓝白斑筛选获得阳性克隆。提取TOL-pMD19-T重组质粒和CAT-pMD19-T重组质粒用PCR进行验证,并经测序分析鉴定(由大连宝生物公司测序部完成)。
1.2.3 构建TOL-pET28a重组质粒及蛋白诱导表达:PCR扩增(20μL):10×Ex Taq Buffer 2μL、dNTPs Mixture(各2.5 mmol/L)1.5μL、Ex Taq酶(5 U/μL)0.1μL、上述重组质粒TOL-pMD19-T 1μL、引物(10μmol/L)各1μL、MgCl21.5μL,灭菌水补足20μL。扩增条件为94 ℃ 5 min预变性后,94 ℃ 30 s、52 ℃ 45 s、72 ℃ 1 min共 35个循环,72 ℃延伸6 min。PCR产物进行1%琼脂糖凝胶电泳检测并切胶回收纯化。
PCR产物克隆:扩增上述TOL-pMD19-T重组质粒上的TOL基因。分别用限制性内切酶XholI和NedI双酶切PCR产物和pET28a质粒,胶回收后16 ℃连接过夜,连接产物转化至大肠杆菌中,用LB/Kana培养基筛选阳性克隆。提取阳性克隆质粒双酶切进行验证,并测序鉴定。
上述阳性克隆菌培养于含Kana(1:1000)的LB液体培养基中,37 ℃ 250 r/min培养过夜,次日取10 mL菌液于1L LB培养基,37 ℃ 250 r/min培养至OD值为0.6~0.8,加IPTG 400μM进行蛋白诱导表达。取诱导后的菌液,经超声裂解后,做SDS-PAGE电泳鉴定。
2 结果
2.1 恶臭假单胞菌在含不同浓度甲苯的MS培养基中的生长情况 经初步培养鉴定,本实验室保存的该株恶臭假单胞菌(WZ-1),生长条件较为严苛,在LB培养基中培养,需30 ℃培养48 h。在无机盐培养基(MS培养基)中培养几乎不生长,而在含有不同浓度甲苯的MS培养基中培养24 h生长情况良好,但在培养24 h后,出现生长停滞(见图1)。

图1 恶臭假单胞菌在含不同浓度甲苯的MS培养基中的生长情况
2.2 TOL基因和CAT基因的克隆和鉴定 经PCR扩增及序列测定鉴定,从该恶臭假单胞菌基因组中获得TOL基因和CAT基因。经T-A克隆后筛选阳性克隆,将该克隆进行质粒测序以及经BLAST比对分析后证实获得TOL-pMD-19-T重组质粒和CAT-pMD-19-T重组质粒,结果见图2-3。
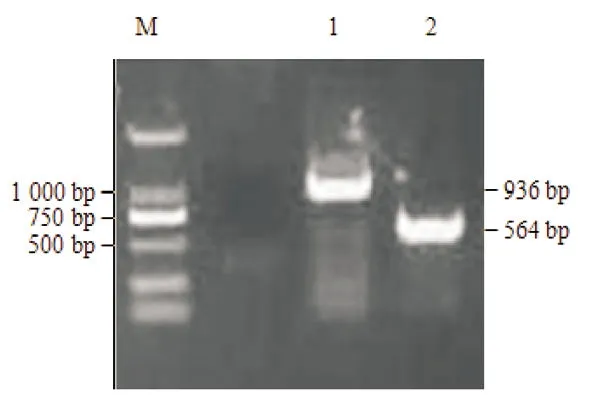
图2 TOL基因和CAT基因PCR扩增结果
2.3 重组质粒TOL-pET28a酶切鉴定 提取TOL-pET28a重组质粒,进行双酶切验证,结果见图4,并经测序分析鉴定。

图3 TOL-pMD-19-T重组质粒和CAT-pMD-19-T-重组质粒测序后序列比对结果

图4 重组质粒TOL-pET28a-TOL酶切鉴定
2.4 重组TOL在大肠杆菌中的表达 结果见图5。
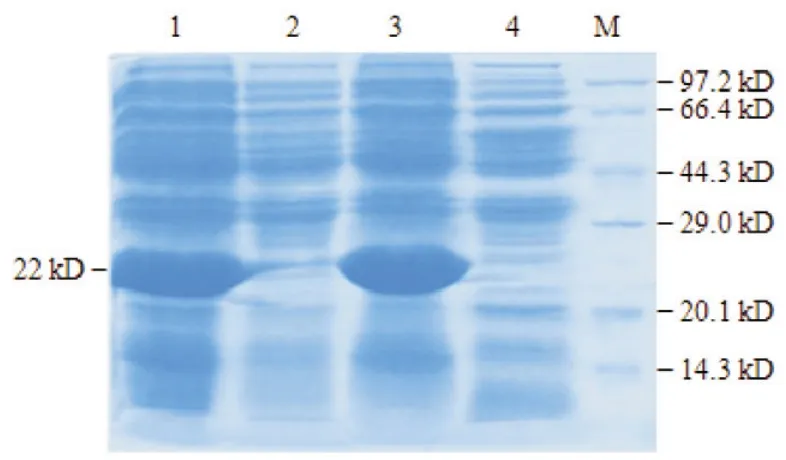
图5 重组菌诱导表达后SDS-PAGE电泳鉴定结果
3 讨论
在过去几年中,随着人们对苯系物污染的重视程度的提高以及科学研究的深入,有关苯系污染物的检测和治理已引起了极大关注[6-23],其中利用模式生物对特定苯系物反应的生物检测和治理更受研究人员的关注[15-23]。至今,已经有多种生物反应器用于芳香族污染物的检测[15-20]。
本实验室已从环境水源中分离到菌株,经初步生化鉴定为恶臭假单胞菌,命名为Pseudomonas sp.WZ-1(恶臭假单胞菌WZ-1),经过进一步的分子分型和进化树绘制,发现该亚型与恶臭假单胞菌NPO-JL-67(Pseudomonas sp. NPO-JL-67)具有相近的亲缘性,为一种新发现的恶臭假单胞菌亚型。为验证该亚型是否具备降解苯系物的能力,我们以甲苯为底物,利用恶臭假单胞菌在含甲苯的无机盐培养基(MS培养基)中是否利用甲苯的生长实验结果为依据,若菌株能有效生长,则证明其能够降解甲苯并利用其降解产物作为碳源以维持生长状态;若生长情况差或者不生长,则说明该菌株在无营养供给的无机盐培养基中无法利用甲苯,即无降解甲苯的能力。本菌株在不同含量甲苯的无机盐培养基中生长情况良好,但在培养24 h后,出现生长停滞或稍有下降趋势,可能由于在培养基中的甲苯已被完全消耗所致,该结果提示,恶臭假单胞菌WZ-1具备降解甲苯的能力,因此可以作为克隆降解甲苯生物酶基因的生物材料。
由于为新分离的菌株,本研究参考了NCBI中提供的恶臭假单胞菌F1全基因组的序列信息,设计引物,经过PCR扩增、产物T-A克隆并测序后证实从该恶臭假单胞菌WZ-1中顺利获得两种酶的基因,即TOL基因和CAT基因,该结果也证明该菌株可能与恶臭假单胞菌F1的序列存在高度同源性。
TOL是苯、甲苯及苯酚在降解途径中的第一步关键酶,本研究中将该酶基因克隆至表达载体pET28a中,获得阳性克隆并转化后,进行体外诱导表达。经电泳鉴定在体外获得大量的TOL,但由于其表达于包涵体,其活性有待进一步鉴定。
获取目的基因是开展基因工程的重要步骤。本实验成功获得了用于降解甲苯的TOL基因和CAT基因,并经测序证实,该成果为后续重组克隆的构建,以及利用模式生物对苯系物的生物降解建立了实验基础。
[1] Richardson SD, Ternes TA. Water analysis: emerging contaminants and current Issues [J]. Anal Chem,2005,77(12):3807-3838.
[2] Brcic I. Determination of benzene, toluene, ethylbenzene,and xylenes in urine by purge and trap gas chromatography[J]. Journal of Separation Science,2003,26(14):1225-1229.
[3] Rosell M, Lacorte S, Ginebreda A, et al. Simultaneous determination of methyl tert.-butyl ether and its degradation products, other gasoline oxygenates and benzene, toluene,ethylbenzene and xylenes in Catalonian groundwater by purge-and-trap-gas chromatography-mass spectrometry [J].J Chromatogr A,2003,995(1-2):171-184.
[4] 周留柱,方黎,孔祥和,等. 大气压力下激光电离技术探测环境痕量苯系物实验研究 [J]. 量子电子学报, 2003, 20(2):203-207.
[5] Kubinec R, Berezkin VG, GorovaR, et al. Needle concentrator for gas chromatographic determination of BTEX in aqueous samples [J]. J Chromatogr B Analyt Technol Biomed Life Sci,2004,800(1-2):295-301.
[6] Zhao X, Suo Y. Analysis of primary aromatic amines using precolumn derivatization by HPLC fluorescence detection and online MS identification[J]. J Sep Sci,2008,31(4):646-658.
[7] Zhu S, Li L, Thornton C, et al. Simultaneous determination of benzo[a]pyrene and eight of its metabolites in Fundulus heteroclitus bile using ultra-performance liquid chromatography with mass spectrometry [J]. J Chromatogr B Analyt Technol Biomed Life Sci,2008,863(1):141-149.
[8] Panariello M, Apicella B, Armenante M, et al. Analysis of polycyclic aromatic hydrocarbon sequences in a premixed laminar flame by on-line time-of-flight mass spectrometry[J]. Rapid Commun Mass Spectrom,2008,22(4):573-581.
[9] Xu Y, Wang H, Zhao J, et al. Analysis of alkylbenzene samples by comprehensive capillary liquid chromatography capillary gas chromatography[J]. J Chromatogr A,2008,1181(1-2):95-102.
[10]Luan T, Fang S, Zhong Y, et al. Determination of hydroxy metabolites of polycyclic aromatic hydrocarbons by fully automated solid-phase microextraction derivatization and gas chromatography-mass spectrometry[J]. J Chromatogr A,2007,1173(1-2):37-43.
[11]Barra R,Castillo C,Torres JP. Polycyclic aromatic hydrocarbons in the South American environment [J]. Rev Environ Contam Toxicol,2007,191:1-22.
[12]Murkovic M. Analysis of heterocyclic aromatic amines[J].Anal Bioanal Chem,2007,389(1):139-146.
[13]Pozzoli L, Gilardoni S, Perrone MG, et al. Polycyclic aromatic hydrocarbons in the atmosphere: monitoring, sources,sinks and fate. I:Monitoring and sources[J]. Ann Chim,2004,94(1-2):17-32.
[14]Sato H, Aoki Y. Mutagenesis by environmental pollutants and bio-monitoring of environmental mutagens[J]. Curr Drug Metab,2002,3(3):311-319.
[15]Applegate BM, Kehrmeyer SR, Sayler GS. A chromosomally based tod-luxCDABE whole-cell reporter for benzene,toluene, ethybenzene,and xylene(BTEX) sensing[J]. Appl Environ Microbiol,1998,64(7):2730-2735.
[16]Willardson BM, Wilkins JF, Rand TA,et al. Development and testing of a bacterial biosensor for toluene-based environmental contaminants [J]. Appl Environ Microbiol,1998,64(3):1006-1012.
[17]Stiner L, Halverson LJ. Development and characterization of a green fluorescent protein-based bacterial biosensor for bioavailable toluene and related compounds. Appl. Environ[J].Microbiol,2002,68(4):1962-1971.
[18]Casavant NC, Thompson D,et al. Use of a site-specific recombination based biosensor for detecting bioavailable toluene and related compounds on roots [J]. Environ Microbiol,2003,5(4):238-249.
[19]Paitan Y, Biran I, Shechter N, et al. Monitoring aromatic hydrocarbons by whole cell electrochemical biosensors[J].Anal Biochem,2004,335(2):175-183.
[20]Kim MN, Park HK, Lim WK, et al. Construction and comparison of Escherichia coli whole-cell biosensors capable of detecting aromatic compounds[J]. J Microbiol Methods,2005,60(2):235-245.
[21]Keane A, Lau PC, Ghoshal S. Use of a whole-cell biosensor to assess the bioavailability enhancement of aromatic hydrocarbon compounds by nonionic surfactants[J]. Biotechnol Bioeng,2008,99(1):86-98.
[22]Tizzard AC, Lloyd-Jones G. Bacterial oxygenases:in vivo enzyme biosensors for organic pollutants[J]. Biosens Bioelectron,2007,22(11):2400-2407.
[23]Paitan Y, Biran I, Shechter N, et al. Monitoring aromatic hydrocarbons by whole cell electrochemical biosensors[J].Anal Biochem,2004 335(2):175-183.
Cloning and expression of the genes of key enzymes in toluene degeneration
YANG Kai,MIAO Xiaoyan,CHEN Qiongsi,ZHANG Tingting,YE Wei,LU Jianxin.
School of Laboratory Medicine,Wenzhou Medical College,WenZhou,325035
Objective:To construct biosensor for the degeneration of toluene,the enzymes genes from Pseudomonas putida were cloned and expressed.Methods:A strain of Pseudomonas putida isolated from the water previously was cultured in MS medium for 24 h to identify the ability of degeneration of toluene firstly. Primers were designed according to toluene dioxygenase and catechol 1,2-dioxygenase gene sequence. PCR was performed as the template of gDNA from the Pseudomonas putida,T-A cloning as followed. Positive colonies were identified by restriction enzyme reactions and sequencing. Toluene dioxygenase gene was inserted to pET28a vector and the recombinant protein was expressed by IPTG induction.Results:This strain of Pseudomonas putida was proved high ability of degeneration of toluene. From its gDNA,acquirement of the two important genes in degeneration of toluene were demonstrated, and toluene dioxygenase gene was induced high level of expression in vivo.Conclusion:In this study,we complete in obtaining toluene dioxygenase and catechol 1,2-dioxygenase gene sequence from pseudomonas putida and inducing the expression of toluene dioxygenase,which is significant for further construction of biosensors.
Pseudomonas putida;toluene;clone;prokaryotic expression
Q781;Q786
A
1000-2138(2012)06-0556-05
2012-03-28
浙江省大学生科技创新活动计划(新苗人才计划)(2010R413013);浙江省教育厅科研基金资助项目(Y20080 4470)。
杨凯(1990-),男,云南曲靖人,本科生。
叶薇,讲师,Email:yewei80@gmail.com。
丁敏娇)
·论 著·
