花鲈微卫星标记分离及其多态性分析
2011-12-25季相山曾勇庆杨萍萍
赵 燕, 季相山, 曾勇庆, 丁 雷, 杨萍萍, 王 慧
(山东农业大学 动物科技学院, 山东 泰安 271018)
花鲈微卫星标记分离及其多态性分析
赵 燕, 季相山, 曾勇庆, 丁 雷, 杨萍萍, 王 慧*
(山东农业大学 动物科技学院, 山东 泰安 271018)
该文利用FIASCO法(fast isolation by AFLP of sequences containing repeats)和GenBank数据库搜索法开发花鲈微卫星标记, 并对筛选的标记进行多态性检测。两种方法共获得54条能够设计引物的序列, 扩增结果显示15对引物具有多态性, 多态性微卫星位点的等位基因数为 2~10个。15个多态性位点中, 4个位点偏离了Hardy-Weinberg平衡; 各位点间没有连锁不平衡现象; 仅位点SP52可能存在无效等位基因; 除SP17和SP468外,其余引物的PIC值均在0.5以上, 可用于花鲈群体遗传分析等研究。
FIASCO法; 数据库查询法; 花鲈; 微卫星
微卫星具有多态性丰富、易于快速检测等特点,在种质资源保存利用、遗传结构和遗传变异及遗传连锁图谱构建等方面的研究中发挥着非常重要的作用, 已经发展成为当前主流的分子标记之一(Wang et al, 2009)。FIASCO法(fast isolation by AFLP of sequences containing repeats)是基于AFLP技术的一种非常有效的分离微卫星位点的方法(Zane et al, 2002)。它将AFLP与杂交选择相结合, 能够简单快速提高阳性率, 发展前景较大, 已经在黄鳝(Monopterus albus) (Li et al, 2007)、鳜鱼(Siniperca chuatsi) (Kuang et al, 2009)等多种鱼类微卫星的开发上使用。数据库搜索法是从公用的DNA 序列数据库(GenBank、EMBL和DDBJ等)中查找所研究物种的序列信息, 并用于开发微卫星标记的一种简单经济有效的方法(Brown et al, 1996)。
当前已开发的花鲈微卫星标记较少, 仅 Jiang et al (2007, 2008)发表了22个微卫星标记, 其中有13个微卫星标记为混合微卫星, 不适于种群遗传学研究, 远远满足不了花鲈遗传育种等研究的需要。在微卫星开发的几种方法中, 从基因组文库中筛选,耗时、费力, 效率较低; 从近缘中扩增, 成功率较低。本研究选择目前应用较广泛的FIASCO法构建花鲈部分基因组文库及参照 GenBank上花鲈的相关序列设计微卫星引物两种方法, 对花鲈微卫星位点的多态性进行研究, 这可为花鲈群体遗传结构的分析、遗传资源的保护利用和分子标记辅助育种等打下基础。
1 材料与方法
1.1 实验材料
实验所用花鲈活体样本捕自山东烟台近海, 共30个个体, 取其尾鳍置于75%酒精中, 保存于-20 ℃冰箱中备用。
1.2 微卫星富集文库的构建与序列测定
用蛋白酶K消化和酚/氯仿抽提法从花鲈的鳍条中分离、纯化基因组DNA (Sambrook & Russell, 2002)。
基因组DNA用限制性内切酶MseⅠ酶切后回收250~1 000 bp的片段, 回收产物与接头oligo-MseⅠA (HO 5'-TACTCAGGACTCAT-3' OH)和oligo-MseⅠB (HO 3'-GAGTCCTGAGTAGCAG-5' OH) 连接。建立17.5 μL连接体系(接头20 μmol/L, 2.5 μL; T4 DNA ligase 6 U, 10×buffer 1.75 μL, 纯化的酶切产物 10 μL), 置16 ℃水浴中过夜连接。利用预扩引物MseⅠ- N(5'-ATGAGTCCTGAGTAAN-3')对连接了接头的DNA片段进行PCR扩增, 程序为94 ℃预变性5 min, 94 ℃变性30 s, 53 ℃退火30 s, 72 ℃延伸 1 min, 共进行 30个循环, 最后 72 ℃延伸10 min。回收250~1 000 bp的预扩增片段用于杂交分析。
杂交体系包含生物素标记的(GT)13的探针 6.0 μL(40 μmol/L), 杂交液(6×SSC + 0.1% SDS)70 μL,预扩增回收产物 20 μL。磁珠经 250 μL的TEN100(10 mmol/L Tris-HCl, 1 mmol/L EDTA, 100 mmol/L NaCl, pH 7.5)洗涤3次, 每次5 min后, 将杂交产物和300 μL TEN100加入到预处理过的磁珠中, 室温反应30 min。分别用400 μL TEN1000(0.2 ×SSC + 0.1% SDS)和洗脱液(0.2×SSC + 0.1% SDS)各洗涤磁珠3次, 每次5 min, 其间不时用移液器轻轻吹打, 重悬磁珠。最后用400 μL 1×SSC洗涤磁珠5 min。加50 μL TE, 95 ℃水浴中变性5 min, 洗脱含有微卫星序列的单链 DNA。以洗脱的含有微卫星的单链 DNA为模板再次用引物MseI-N 进行扩增, 扩增程序同上, 回收 250~1 000 bp的片段。
将回收的目的片段连接到pMD18-T载体中并转化至大肠杆菌 DH5α中, 涂平板后挑取单克隆,用引物MseI-N进行PCR检测, 挑选阳性克隆送上海生物工程技术服务有限责任公司测序。利用DNAMAN Version 4.0 和GenBank中的Blastn软件对测得序列进行比对, 去掉冗余序列。
1.3 GenBank数据库搜索
输入“Laterolabrax japonicus”作为搜索关键词,在 NCBI公共数据库(http: // www.ncbi.nlm.nih.gov)搜索核苷酸序列, 下载所有相关序列以 FSATA格式保存备用。
1.4 序列分析及微卫星引物设计
利用 SSRHUNTER 1.3(http://en.bio-soft.net/ dna/SSRHunter.html)软件, 对以上两种方法得到的序列进行分析, 含二碱基、三碱基、四碱基重复 5次以上的为含微卫星的序列。根据微卫星的侧翼序列, 利用软件Premier 5.0设计引物, 并交由上海生物工程技术服务有限责任公司合成。
1.5 微卫星标记的多态性检测
利用合成的引物分别在 30个花鲈个体中进行PCR扩增用于分析其多态性。PCR反应总体积为20μL , 内含20 ng的模板DNA, 0.2 μmol/L的引物, 200 μmol/L的dNTPs, 1.5 mmol/L 的Mg2+, 1 X PCR 反应缓冲液, 1 U 的Taq DNA 聚合酶。PCR反应为 30个循环, 每个循环包括: 94℃变性 30 s,退火30 s, 72 ℃延伸1 min; 首次循环前预变性5 min; 最后一次循环结束后 72 ℃再延伸 10 min。PCR 扩增产物经 6%变性聚丙烯酰胺凝胶电泳, 硝酸银染色后数码相机拍照保存。
1.6 数据分析
微卫星标记呈共显性, 可直接根据每个个体产生的条带的位置确定基因型, 利用Popgene32 处理数据, 计算多态性位点的等位基因数目、观测杂合度(HO)、期望杂合度(HE), 并按下列公式计算多态性信息含量(PIC):
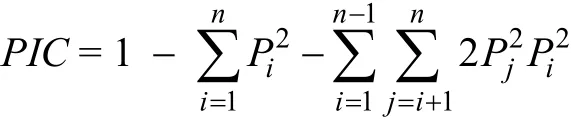
其中,Pi和Pj分别为第i和第j个等位基因的频率,n为等位基因个数。
利用 MICRO-CHECKER Version 2.2.3软件估计位点的无效等位基因频率。所有多重比较均经Bonferroni校正。
2 结 果
2.1 FIASCO法筛选的微卫星标记
从构建的文库中随机挑取150个克隆, 利用预扩引物进行PCR鉴定, 共得到阳性克隆94个, 阳性率为62.7%; 对阳性克隆测序得到66个含微卫星序列的克隆, 阳性序列比例为70.2%。66条微卫星序列中共含微卫星座位108个, 其中CA/GT重复序列97个, 占89.8%, GA/CT重复4个, 占3.7%, 其他重复序列7个, 占6.5%。在这些含微卫星重复的序列中, 微卫星重复数在10次以上的, 占70.3%。在这些位点的重复序列中, 共3个复合型(sp52、sp416、sp468), 其他为完美型。利用 DNAMAN 4.0 和GenBank中的Blastn软件对这66条微卫星序列进行比对, 去掉冗余序列后全部提交 GenBank, 并利用 Premier 5.0软件共设计引物42对, 部分微卫星序列及引物信息见表1。
2.2 GenBank 数据库搜索结果
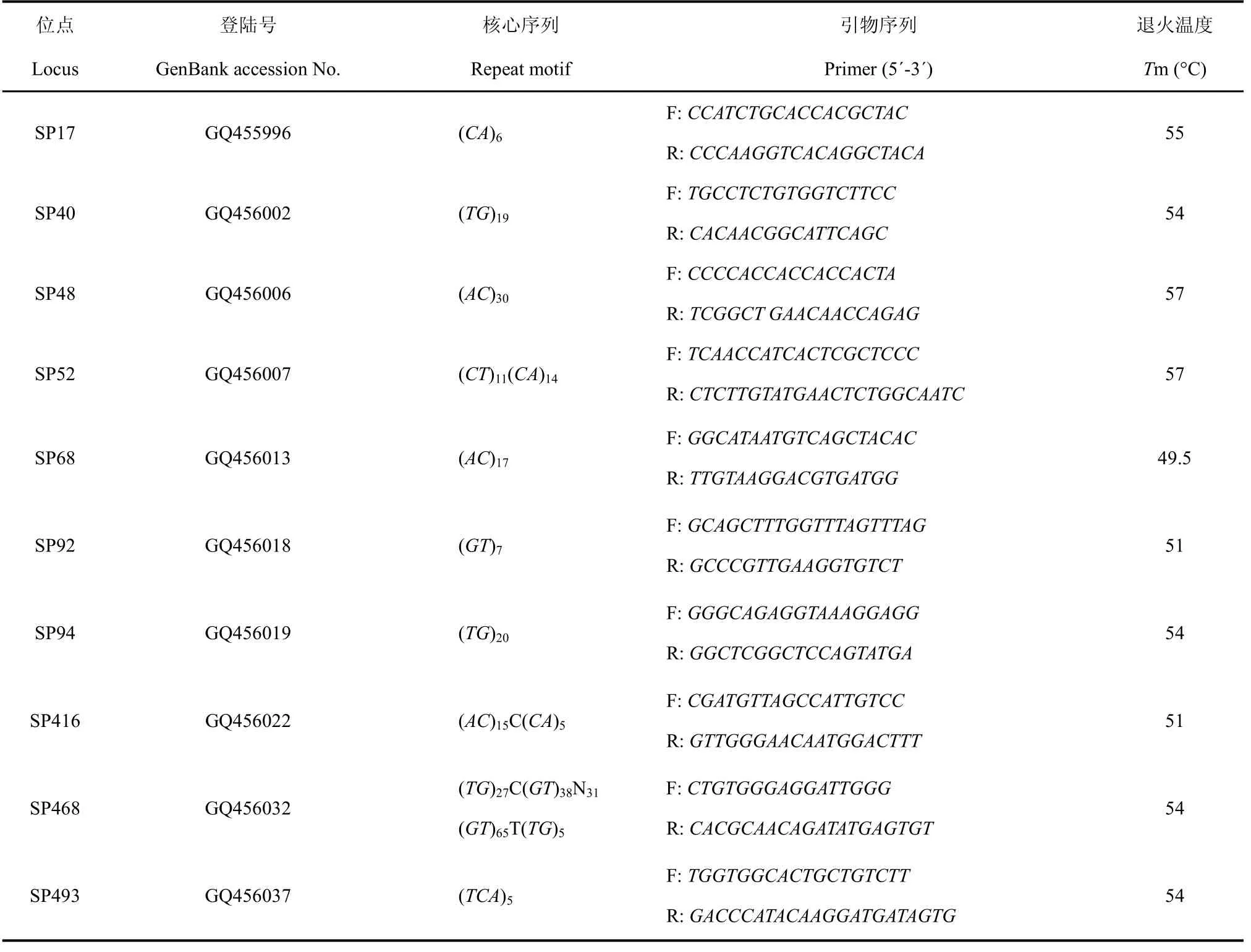
表1 FIASCO法获得的部分花鲈微卫星序列Tab. 1 Microsatellite sequences obtained by FIASCO
截至2010年2月, 在GenBank数据库中共搜索到540条花鲈核苷酸序列及132条EST序列, 经SSRHUNTER 1.3软件分析, 含微卫星的序列为22条, 其中8条为基因的全长cDNA序列; 1条为基因的部分cDNA序列; 13条为EST序列。在22条微卫星序列中含微卫星座位 29个, 其中CA/GT重复序列14个, 占48.3%,GA/CT重复10个, 占34.5%,三碱基重复序列5个, 占17.2%。在这些含微卫星重复的序列中, 微卫星重复数在10次以上的仅6个,重复数在5~7次间的为22个(75.8%)。在8条基因的全长 cDNA序列中, 微卫星座位多数位于内含子、3′-UTR、5′-UTR等区域, 仅有1个位于外显子区域(表2)。
2.3 微卫星标记多态性分析
两种方法筛选的微卫星序列共设计引物54对,利用8尾花鲈的DNA对各引物扩增情况作初步筛选, 发现32对能扩增出与期望PCR产物大小相等或相近的片段, 其中 15对引物的扩增片段有稳定的多态性(表3), 其余的都为单态。应用这15对引物对30个个体进行了PCR扩增, 扩增条带清晰、稳定, 其中微卫星引物SPd10扩增产物的聚丙烯酰胺凝胶电泳图谱见图1。
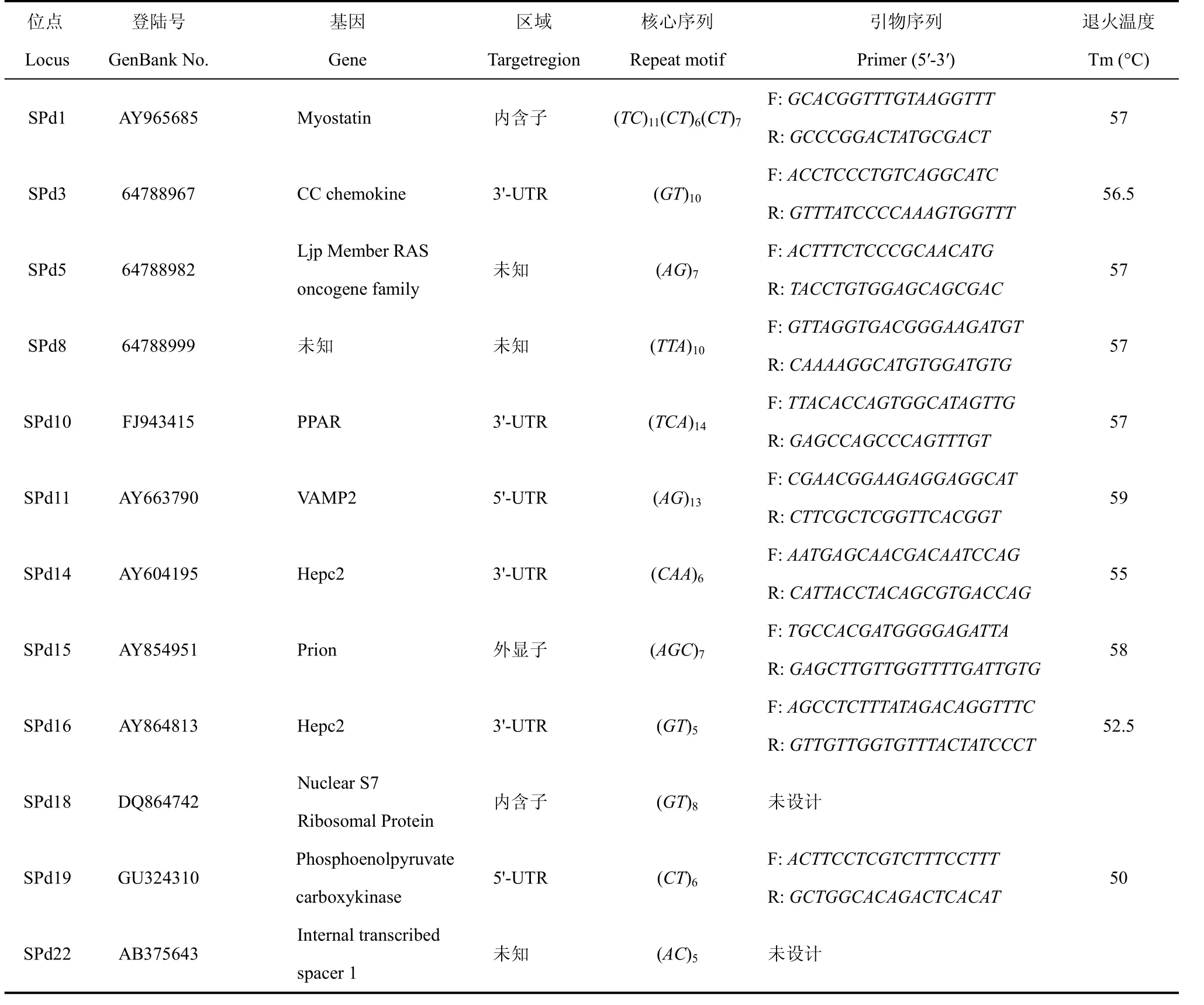
表2 GenBank数据库查询得到的部分微卫星序列Tab. 2 Microsatellite sequences obtained by bioinformatic screening in GenBank
在野生花鲈30个个体中, 15个花鲈多态性微卫星位点共检测到89个等位基因, 平均每个位点5.9个等位基因, 表现出高度多态性, 各等位基因大小在141~349之间(表3)。Popgene32 分析结果显示, 15个多态性微卫星位点的观测杂合度在0.6000~序的微卫星序列中, CA/GT重复序列97个, 占1.0000之间, 期望杂合度在 0.5079~0.8890之间。经Bonferroni 校正后(P< 0.003), 各位点间没有连锁不平衡现象, 4个微卫星位点(SP17、SP52、 SP94 和SP468)偏离了 Hardy-Weinberg平衡。MICROCHECKER 2.2.3软件分析表明仅位点SP52可能存在无效等位基因。同时, 对 15对微卫星引物的多态性信息含量(PIC)进行了评估, SP17位点提供的信息含量为 0.4189; SP468为 0.3747, 其余引物的PIC值均在0.5以上, 可用于花鲈群体遗传分析等研究。
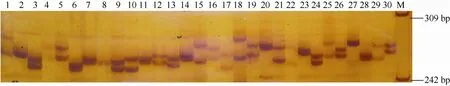
图1 微卫星标记SPd10在花鲈30个个体中的扩增图谱Fig. 1 Amplification results of microsatellite locus SPd10 in 30 Lateolabrax japonicus individuals
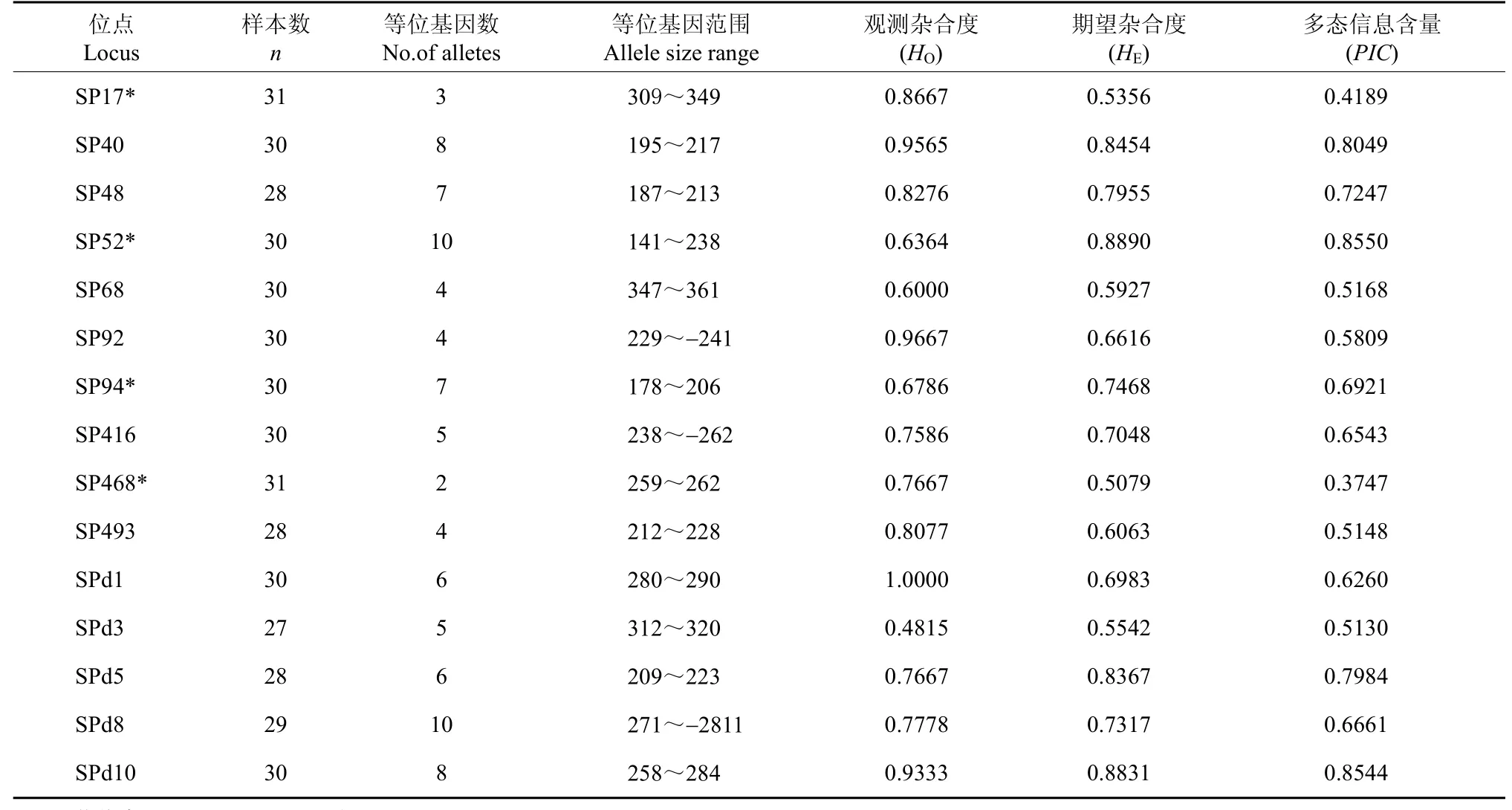
表3 15个多态性花鲈微卫星标记分析结果Tab. 3 Characteristics of 15 polymorphic microsatellite markers in Lateolabrax japonicus
3 讨 论
目前开发微卫星标记的方法很多, 概括起来有3种: (1)从基因组文库中筛选微卫星; (2)从数据库或有关文章中查询; (3)从近缘物种中筛选微卫星(Zhan et al, 2008)。本文研究发现, 数据库搜索法得到的花鲈微卫星位点的多态率为 41.7%, 多态性较高。其他的研究也得到类似的结果, 如Ji et al(2009)搜索 GenBank数据库中的牙鲆(Paralichthys olivaceus)全cDNA序列, 得到15个具有高多态性的微卫星位点, 多态率为 46.8%; Furukawa et al (2004)用同样的方法搜索到11个具有多态性的河豚微卫星位点, 多态率为73%。从数据库中查找微卫星位点, 虽然省时省力, 但受限于数据库中提交序列的数量, 多数物种因提交的序列较少只能开发出有限的多态性微卫星标记。FIASCO法是Zane et al (2002)年提出的一种高效的筛选微卫星标记的方法。此法一经提出, 便被迅速广泛地应用于许多物种的微卫星位点的筛选(Reed et al, 2001)。本文利用FIASCO法构建了花鲈(GT)13的微卫星富集文库,在所有测序的微卫星序列中,CA/GT重复序列 97个,占89.8%。而通过传统的筛选小插入片段基因文库的方法分离微卫星标记, 得到的阳性克隆比例极低(0.1%~6.4%, 平均1.96%) (Zane et al., 2002)。
虽然微卫星可分布于基因组的任何地方, 但是在真核生物中, 微卫星位于非编码区的频率远高于位于编码区的(Toth et al, 2000)。本文研究发现, 在花鲈8条基因的全长cDNA序列中, 微卫星座位多数位于内含子、3'-UTR、5'-UTR等区域, 仅有1个位于外显子区域, 占 12.5%, 这与其它脊椎动物中微卫星在外显子区的分布频率(9%~15%)(Moran et al, 1993; Jurka et al, 1995)基本一致。这种微卫星位点在编码区的低频现象可能是为了防止蛋白质翻译时发生移码突变的一种保护机制(Metzgar et al, 2000; Li et al, 2007)。
本研究得到的15个多态性微卫星位点中有4个(SP17、SP52、SP94和SP468)偏离了Hardy-Weinberg平衡。偏离HWE的原因有很多, 例如, 无效等位基因、检测个体不具有代表性、交配群体等。软件分析表明, 仅位点SP52可能存在无效等位基因, 造成其他3个位点偏离HWE的原因可能是检测个体数目过小, 检测的个体不具代表性等所致。
微卫星位点的多态性一般用3个指标评价: 等位基因的数目、杂合度和多态性信息含量值(PIC)。其中等位基因数目和杂合度能较好地反映群体中等位基因的丰富程度和均匀程度, 而多态性信息含量值反映了微卫星位点能够提供的遗传信息容量。本研究得到的花鲈 15个多态性微卫星位点共检测到 89个等位基因, 平均每个位点 5.9个等位基因,表现出高度多态性, 且 15个多态性微卫星位点的观测杂合度在 0.6000~1.0000之间, 期望杂合度在0.5079~0.8890之间; 而Jiang et al(2007)对22个微卫星位点的多态性分析显示, 其观测杂合度在0.114~0.914之间, 期望杂合度在0.111~0.938之间,这表明花鲈野生群体具有较高的遗传多样性。据Gu et al (2008), 当PIC>0.5 时, 表明该遗传标记可提供丰富的遗传信息。本文研究结果表明, 15个花鲈多态性微卫星位点中的13个位点PIC值均大于0.5, 说明本文新开发的微卫星位点提供的遗传信息容量大,能为群体遗传性分析提供有力的工具。
本文利用 FIASCO法和数据库查询法两种方法开发了花鲈微卫星位点 15个, 并对其多态性进行了研究, 结果表明数据库查询法简便快捷, 多态性好; FIASCO法富集效率高, 适合用于花鲈微卫星标记大规模的开发。开发的多态性微卫星标记可为花鲈群体遗传学研究、连锁图谱构建等提供有力的支持。
Brown SM, Hopkins MS, Mitchell SE. 1996. Multiple methods for the identification of polymorphic simple sequence repeats (SSRs) in sorghum [sorghum bicolor(L.) Moench] [J].Theol Appl Genet, 93: 190-198.
Furukawa S, Takeshima H, Otaka T, Misuboshi T, Shirasu K, Ikeda D, Kaneko G, Nishida M, Watabe S. 2004. Isolation of microsatellite markers by in silico screening implicated for genetic linkage mapping in Japanese pufferfishTakifugu rubripe s[J].Fish Sci, 70: 620-628.
Gu XY, Zeng QG, You ZJ, Lin ZH.2008. Isolation and characteristics of six microsatellite primers oftegillarca granosa[J]. Oceanol Limnol Sin, 39: 661-664. [顾晓英, 曾庆国, 尤仲杰. 2008. 泥蚶(Tegillarca granosa)6个微卫星引物的分离和鉴定. 海洋与湖沼. 39: 661-664.]
Ji XS, Chen SL, Ma HY, Xu TJ, Liao XL, Jiang YL. 2009. Isolation and characterization of 19 EST-linked polymorphic microsatellite loci for olive flounder (Paralichthys olivaceus) [J].Aquac Res, 40: 980-983.
Jiang X, Liao MJ, Liu YJ, Gao T, Yang GP. 2007. Isolation and characterization of 22 polymorphic microsatellite DNA markers of Japanese sea bass (Laterolabrax japonicus). [J].Mol Ecol Notes, 7: 492-494.
Jiang X, Yang G, Liao M, Liu Y, Gao T, Wang D, Wei Q, Zou G. 2008. Microsatellite DNA polymorphism of Japanese sea bass (Laterolabrax japonicus) inhabiting Chinese and Japanese coasts[J].J Appl Ichthy, 24: 180-186.
Jurka J, Pethiyagoda C. 1995. Simple repetitive DNA sequences from primates: compilation and analysis[J].J Mol Evol, 40: 120-126.
Kuang GQ, Lu SQ, Zheng SM, Wu Q. 2009. Isolation and evaluation of 18 microsatellite markers inSiniperca chuatsi(Basilewsky) [J].Mol Ecol Resour, 9: 1473-1475.
Li WT, Liao XL, Yu XM, Cheng L, Tong J. 2007. Isolation and characterization of polymorphic microsatellites in a sex-reversal fish, rice field eel (Monopterus albus) [J].Mol Ecol Notes, 7: 705-707.
Metzgar D, Wills C. 2000. Evolutionary changes in mutation rates and spectra and their influence on the adaptation of pathogens[J].Microbes Infect, 2: 1513-1522.
Moran C. 1993. Microsatellite repeats in pig (Sus domestica) and chicken (Gallus domesticus) genomes[J].J Hered, 84: 274-280.
Reed K M, Beanie C W. 2001. Isolation of 105 microsatellite loci from an ovine genomic library enriched for microsatellites[J].Anim Biotech, 12: 77- 86.
Toth G, Gaspari Z, Jurka J. 2000. Microsatellites in different eukaryotic genomes: survey and analysis[J].Genome Res, 10: 967-981.
Wang JP, Yu XW, Shen PY, 2009. Microsatellite loci of portunus trituberculatus: isolation and characterization[J].Oceanol Limnol Sin, 40: 83-87. [王建平, 余晓巍, 沈庞幼, 2009. 三疣梭子蟹(Portunus trituberculatus)微卫星位点的分离和序列分析。海洋与湖沼, 40: 83-87.]
Zane L, Bargelloni L, Patarnello T. 2002. Strategies for microsatellite isolation: a review[J].Mol Ecol, 11: 1-16.
Zhan AB, Bao ZM, Hu XL, Wang S, Peng W, Wang ML, Hu JJ, Liang CZ, Yue ZQ. 2008. Characterization of 95 novel microsatellite markers for Zhikong scallopChlamys farreriusing FIASCO-colony hybridization and EST database mining[J].Fish Sci, 74: 516-526.
Zhang YW, Zhang YP, Oliver AR. 2001. Microsatellites and its application[J].Zool Res,22(4): 315-320.[张云武, 张亚平. 2001. 微卫星及其应用. 动物学研究, 22(4): 315-320.]
Isolation of microsatellite markers forLateolabrax japonicusand polymorphic analysis
ZHAO Yan, JI Xiang-Shan, ZENG Yong-Qing, DING Lei, YANG Ping-Ping, WANG Hui*
(College of Animal Science and Technology,Shandong Agriculture University,Taian271018,China)
To investigate population structure and marker assisted breeding, fast isolation by AFLP of sequences containing repeats (FIASCO) and GenBank database mining were used to develop novel microsatellite markers for sea perch (Lateolabrax japonicus). Genomic DNA fragments containing SSR sequences were captured by hybridization to (GT)13biotin-labeled probe and were ligated to PMD18-T vector. Among 150 randomly chosen clones from the SSR-enriched library, 66 sequences contained microsatellite motif over five repeats. In addition, 540 cDNA sequences and 132 ESTs ofLateolabrax japonicuswere downloaded from GenBank and screened for di-, tri- and tetra-nucleotide repeats, while 22 sequences were found to contain microsatellites. As a result, 15 microsatellite loci were shown to be polymorphic in 30Lateolabrax japonicusindividuals, with the alleles ranging from two to ten, the observed heterozygosities from 0.6000 - 1.0000, and the expected heterozygosities from 0.5079 - 0.8890. Four loci (SP17, SP52, SP94 and SP468) were deviated from HWE in the sampled population after Bonferroni’s correction, and no linkage disequilibrium was found among all loci (P<0.003), whereas null alleles were detected at locus SP52 (P<0.05). Among 15 polymorphic loci, the PIC values, which can be used for related population genetics analysis, were all above 0.5, with the exception of SP17 and SP468.
FIASCO; Database mining;Lateolabrax japonicus; Microsatellite
Q95-33; Q959.483
A
0254-5853-(2011)05-0515-06
10.3724/SP.J.1141.2011.05515
2011-02-21; 接受日期: 2011-07-08
国家“863”计划项目(2008AA101010); 国家发改委生物育种高技术专项(2007249051); 山东省农业良种工程重大项目(2007LZ013); 博士后创新基金项目(200703044)
∗通讯作者(Corresponding author),王慧(1963 - ), 女, 教授, 博导,Tel: 13001771684; E-mail: wanghui2328@sdau.edu.cn
猜你喜欢
杂志排行
Zoological Research的其它文章
- 白眉山鹧鸪冬季觅食地选择
- Association of RELN promoter SNPs with schizophrenia in the Chinese population
- Decreased contrast sensitivity of visual cortical cells to visual stimuli accompanies a reduction of intracortical inhibition in old cats
- Complete mitochondrial genome of the laced fritillary Argyreus hyperbius (Lepidoptera: Nymphalidae)
- Possible genetic reproductive isolation between two tilapiine genera and species: Oreochromis niloticus and Sarotherodonmelanotheron
- A new blind loach species, Triplophysa huanjiangensis (Teleostei: Balitoridae), from Guangxi, China
