1种小麦白粉病菌DNA基因组的微量简捷提取方法
2011-09-25龚双军杨立军刘辉向立波喻大昭
龚双军,杨立军,刘辉,向立波,喻大昭*
(1.湖北省农科院植保土肥研究所湖北省重大农作物病虫草害重点实验室,湖北武汉430064;2.襄阳市植保站,湖北襄阳441000)
1种小麦白粉病菌DNA基因组的微量简捷提取方法
龚双军1,杨立军1,刘辉2,向立波1,喻大昭1*
(1.湖北省农科院植保土肥研究所湖北省重大农作物病虫草害重点实验室,湖北武汉430064;2.襄阳市植保站,湖北襄阳441000)
为了寻找适合小麦白粉菌基因组DNA微量提取的方法,分别采用改进破壁法,液氮研磨法和溶菌酶消化法进行破壁,提取专性寄生菌小麦白粉菌DNA。结果表明,用改进的破壁方法,仅用3~10 mg的分生孢子粉所获得DNA的收率为(12.23±3.46)~(40.32±5.67)ng/mg,且OD260/OD280比值为1.71~1.92之间,说明该破壁方法获得的DNA收率大且纯度高。通过PCR反应获得了良好的效果。同时该方法也适用于小麦条锈菌和大麦白粉菌专性寄生菌DNA的提取。
小麦白粉病菌;DNA微量提取
小麦白粉病是小麦重要病害之一,而小麦白粉病菌(Blumeria graminis f.sp.tritici)是一种活体专性寄生菌。可用于提取DNA的材料几乎仅为病叶表面的分生孢子,而分生孢子的繁殖收集耗时长,工作量大,成为进行群体研究的技术瓶颈。发展简便易行的高质量DNA提取方法是进行该病菌种群遗传多样性、分子抗药性监测以及病害流行预测等研究的重要保障。目前,真菌DNA的提取通常采用以冷冻干燥菌丝研磨法或酶解形成原生质球法破解细胞壁,然后以高浓度EDTA的SDS溶液裂解细胞膜,再以酚/氯仿/异戊醇或者氯仿/异戊醇抽提,乙醇沉淀,得到高质量DNA。一般耗时长,费用高[1-2]。以往对于小麦白粉病菌DNA提取通常和真菌DNA基因组提取方法类似。在对白粉病菌进行分子生物学研究中,在参考和使用不同的提取方法基础上[3-4]进行改进,探索并建立一套简便、易行、微量(3~5 mg分生孢子)的白粉病菌DNA的提取方法,同时也适合其他专性寄生菌。采用该方法获得高分子量DNA、纯度和浓度较高,PCR分析都获得良好的效果,完全适于进行各类分子生物学研究工作。
1 材料与方法
1.1材料
1.1.1 菌株培养及分生孢子的收集采用文献[5]方法进行菌种培养。温室隔离培养至小麦第一叶完全展开,剪取叶片中段3 cm长,叶片正面朝上放在含50 μg/mL苯骈咪唑的琼脂保鲜培养基上(100 mL蒸馏水+0.5 g琼脂+3 mL 0.2%的苯骈咪唑),在接种筒(23.5 cm×23.5 cm×100 cm)内对上述离体叶段接种预先繁殖好的新鲜分生孢子。把处理后培养皿中的叶段置于培养箱中培养((17±1)℃,18 h光照),待产生大量孢子时于超净工作台上收取孢子。将培养皿倒扣在硫酸纸上,轻轻用镊子敲打2~3次,收集到2 mL离心管中备用。大麦白粉菌(Blumeria graminis f.sp.horde)由华中农业大学董吾辈老师提供,小麦条锈菌(Puccinia striiformis f.sp.tritici)由中国农科院植保所提供。
1.1.2 试剂提取液为0.35 mol/L山梨醇,0.1 mol/L Tris,0.05 mol/L EDTA(pH 7.5),0.02 mol/L亚硫酸氢钠;核裂解液为0.2 mol/L Tris,0.05 mol/L EDTA,2.0 mol/L NaCl,2%(质量与体积比)CTAB。
1.2 方法
1.2.1 改进CTAB法称取白粉病菌的分生孢子粉3、5和10 mg,置于2 mL的离心管中,加入300 μL提取液,300 μL核裂解液,7粒直径为5 mm的玻璃珠(Sigma公司,货号18406);漩涡振荡器上震荡3、5、7和10 min后加入1 μL浓度为20 mg/mL的蛋白酶K;再加入120 μL 20%SDS;65℃水浴1 h;冷却后加入720 μL(氯仿/异戊醇,24∶1)抽提,上下温和颠倒20次;加1 μL RNAse(10 mg/mL),于37℃水浴10 min。平衡后12000 r/min离心10 min;取400 μL上清液;加入40 μL 3 mol/L NaAc (pH 8.0)和800 μL无水乙醇,沉淀1 h;12000 r/min离心10 min;弃上清,沉淀加600 μL 75%乙醇洗涤,将沉淀干燥。40 μL无菌水溶解;取1 μL于0.8%琼脂糖电泳检测,标准λ-HindⅢdigest DNA Marker 5 μL作对照。以标准浓度的DNA marker作对比,直观比较DNA浓度、DNA降解和RNA残存情况。用同样方法提取大麦白粉菌(B.graminis f.sp.horde)、小麦条锈菌(P.striiformis f.sp.tritici)2种专性寄生菌的DNA。
1.2.2 与其他破壁方法的比较分别以酶法[6]和液氮研磨法[7]提取总DNA。其中酶法步骤如下:用700 μL TE缓冲液重悬沉淀,加入300 μL溶菌酶(50 mg/mL),37℃温育1 h。加1 μL浓度为20 mg/mL蛋白酶K和60 μL 20%SDS,65℃温育30 min,12000 r/min离心10 min,上清液转移至另一离心管中,以酚-氯仿法抽提[8]。同时以3、5和10 mg的分生孢子粉震荡3 min作为对照,按照1.2.1方法进行提取DNA。
1.2.3 总DNA浓度、纯度和产量的测定用紫外吸收光谱法[9]定量测定产物DNA的浓度和纯度A260/A280比值,确定DNA的质量和产量。
1.2.4 PCR扩增对1.2.1方法提取的20个DNA样品扩增aox基因,扩增引物为AOX361F/ 589R,PCR反应的条件和体系参照文献[10]进行。
2 结果
2.1 分生孢子DNA的提取
提取的小麦白粉菌DNA在0.8%的琼脂糖上进行电泳检测见图1,总DNA大小都在23.1 kb以上,在所设定的3、5和10 mg孢子质量均可提取到DNA,且随着孢子质量和震荡时间的增加,DNA样品的浓度增高,根据点样量为1 μL的50 ng/μL λ-HindⅢdigest DNA Marker分子量标准估算出提取DNA的产量可稳定在15~40 ng/mg分生孢子。采用该方法用非常微量(3 mg)分生孢子震荡破壁10 min也可以提取到较好的DNA样品(图1),考虑到工作的效率建议震荡破壁时间为3 min。
2.2 与其他破壁方法的比较
采用液氮研磨和酶解破壁法与上述方法进行DNA提取效果比较。结果表明,液氮研磨的效果较上述方法相比,所获得DNA样品浓度与本方法所提取的样品DNA浓度基本一致,但所需要的孢子量较多(表1),然而本实验中采用酶解法未获得DNA,这可能与酶解破壁所需的时间以及酶的效率有一定的关系。
2.3 PCR扩增
如图2所示,采用该方法在所提取的20个DNA样品均可以扩增出相应的目的条带,说明采用该方法所提取的DNA完全可以进行PCR等常规的分子生物学试验。
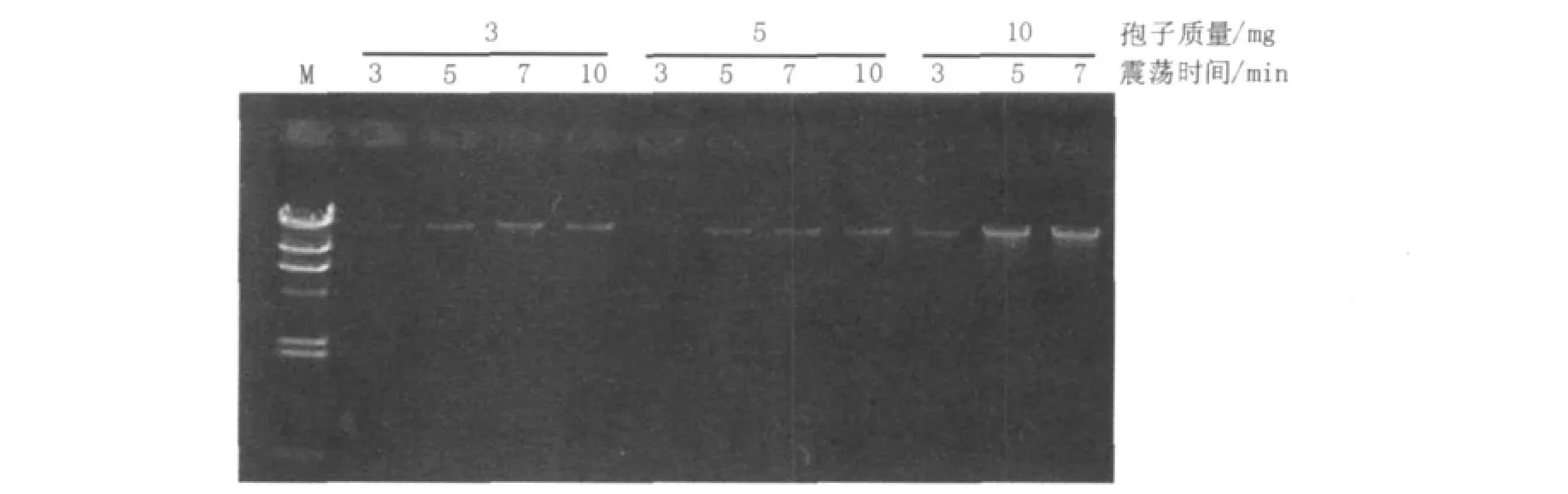
图1 改进的CTAB法提取小麦白粉病菌DNA琼脂糖凝胶电泳图aFig.1the effect of DNA extraction yields with modified method by agarose electrophoresis M:molecular size marker λ-HindⅢdigest DNA Marker

表1 比较不同的破壁提取法对专性寄生菌DNA浓度和纯度的影响Table 1Comparison of biotrophic filamentous DNA quantity and purity from different extraction methodsa
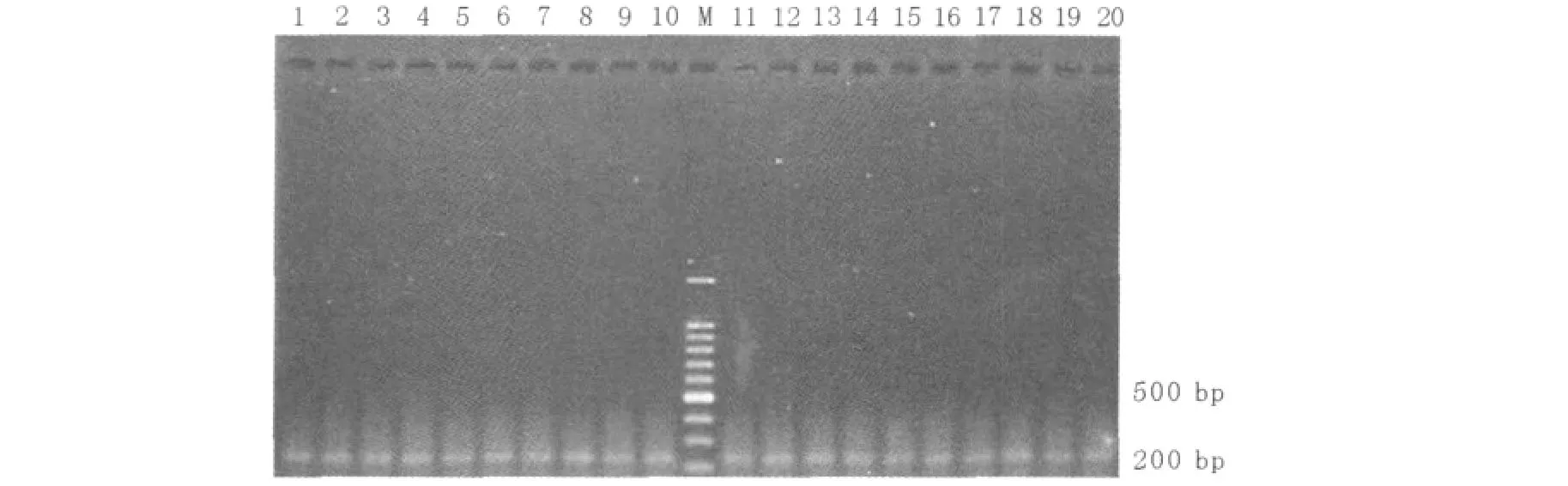
图2 不同DNA样本的AOX PCR产物琼脂糖凝胶电泳图Fig.2Agaros gel electrophoresls of AOX PCR products from different DNA samples 1~20:提取的DNA样品1~20 indicated the different DNA samples
3 讨论
用玻璃珠破碎细胞壁以及液氮研磨破壁法均能获得小麦白粉病菌基因组DNA,两者在DNA纯度和产量上相当,但采用改进CTAB法与传统液氮研磨破壁法的区别在于在离心管中加入玻璃珠,涡旋震荡破解细胞壁,仅需微量的分生孢子粉,克服了过去使用研钵研磨造成的样品浪费和污染。而对于专性寄生菌就大大节约了扩繁菌种时间,从而加快试验进度。同时,本方法尝试在灰葡萄孢(Botrytis cinerea)的孢子悬浮液中提取DNA并获得成功(数据未给出)。
在本实验中,涡旋混合器上充分混匀,是获得DNA收率的关键步骤。本研究表明,一般专性寄生菌的分生孢子量在10 mg左右涡旋震荡3 min即可获得较好的破壁效果,如果时间过长容易造成DNA降解,反之破壁不充分使核酸得不到充分的释放。以改进CTAB法获得高分子量DNA纯度高,可直接用PCR常规分子生物学工作,并获得了良好的试验结果。专性寄生菌DNA提取方法的改进为下一步开展小麦白粉病菌单核苷酸点突变(SNP)的种群研究提供帮助。
致谢:对在实验过程给予帮助的华中农业大学大学本科实习生韩容等同学表示感谢!
[1]Specht CA,Dirusso CC,Novontny CP,et al.A method for extracting high-molecular-weight deoxynucleic acid from fungi[J].Anal Biochem,1982,119(1):158-163.
[2]Carber R C,Yodar O C.Isolation of DNA from filamentous fungi and separation into nuclear,mitochondrial,ribosomal and plasmid components[J].Anal Biochem,1983,135(2):416-422.
[3]Ristaino J B,Madritch M,Trout C L,et al.PCR Amplification of ribosomal DNA for species identification in the plant pathogen genus Phytophthora[J].Appl Environ Microbio,1998,64 (3):948-954.
[4]Justesen AF,Ridout CJ,Hovmoller MS.The recent history of Puccinia striiformis f.sp.tritici in Denmark as revealed by disease incidence and AFLP markers[J].Plant Pathol,2002,51 (1):13-23.
[5]杨小军,倪汉文,杨立军.采用孢子萌发法测定化合物对白粉病菌的生物活性[J].植物保护,2007,33(1):75-77.
[6]Amann RI,Ludwing W,Schleifer K H,et al.Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J].FEMS Microbiol Review,1995,59(1):143-169.
[7]Edwards,K.,C.Johnstone,C.Thompson.A simple and rapid method for the preparation of genomic plant DNA for PCR analysis[J].Nucleic Acids Res,1991,19(6):1349.
[8]萨姆布鲁克J,弗里奇E F,曼妮阿蒂斯T著,金冬雁,黎盂枫译.分子克隆实验指南[M].北京:科学出版社,1999: 200-300.
[9]奥斯伯F,金斯顿R E,塞得曼J G,等著,颜子颖,王海林译.精编分子生物学实验指南[M].北京:科学出版社,1998:831-833.
[10]Parks R,Carbone I,Murphy Paul J.,et al.Population genetic A-nalysis of an eastern U.S.wheat powdery mildew population reveals geographic subdivision and recent common ancestry with U.K.and Israeli populations[J].Phytopath,2009,99(7): 840-849.
Simple and Rapid Method for DNA Genome Micro-Extraction from Wheat powdery mildew(Blumeria graminis f.sp.tritici)
GONG Shuang-jun1,YANG Li-jun1,LIU Hui2,XIANG Li-bo1,YU Da-zhao1
(1.Inst.for Plant Protect’n&Soil Sci.,Hubei Acad.of Agric.Sci.,Hubei Key Lab.of Crop Dis.,Insect Pests&Weeds Control,Wuhan 430064; 2.Plant Protection Station,Xiangyang City,Hubei Xiangyang 441000)
Three different DNA extraction methods for wheat powdery mildew(Blumeria graminis f.sp.tritici)were adopted.They were modified cell-wall breaking method,liquid nitrogen grinding method,and lysozyme digestion method.The results showed that the recovery rate of DNA extracted with modified cell-wall breaking method was (12.23±3.46)~(40.32±5.67)ng/mg using only 3~10 mg of condia,and OD260/OD280purity ratios were 1.71~1.92 indicated that cell-wall breaking method gained greater DNA yield and purity.PCR reactions obtained also good effects.At the same time,the method was also applicable to extract DNA from wheat stripe rust and barley powdery mildew and other DNAs from autoecious parasitic fungi.
wheat powdery mildew;DNA microextraction
S4
A
1005-7021(2011)01-0024-04
公益性行业(农业)科研专项(3-15);国家小麦产业体系项目(NYCYTX-03);公益性行业(农业)科研专项(200903035-09)
龚双军男,助理研究员。研究方向为小麦主要病害种群研究及化学防治。E-mail:gsj204@126.com
*通讯作者。Tel:027-87380681,E-mail:Dazhaoyu@china.com
2010-12-17;
2011-01-20
