MIR-122对IFN-α抗HCV效应的影响
2011-08-06李苏娟朱海红
李苏娟,陈 智,朱海红
(浙江大学医学院附属第一医院传染病诊治国家重点实验室,浙江杭州310003)
丙型肝炎病毒(HCV)是导致肝脏疾病的一个重要病原体,约80%的HCV感染者会发展为慢性肝炎[1]。目前,IFN-α 联合利巴韦林(RBV)是治疗慢性丙型肝炎(CHC)的标准方案,但仅有约50%患者能获得持续病毒学应答(SVR)[2],这与宿主、病毒和 IFN 等因素有关[3-4]。miR-122是一种肝脏特异性的miRNA,占肝脏总miRNA表达量的70%[5],与肝脏的发育、代谢及癌症有关[6-7]。近年来,Jopling等[8]发现miR-122促进HCV的复制,系第一个被证明与病毒复制成正相关的宿主miRNA。本研究旨在探讨miR-122是否影响IFN-α的抗HCV效应。
1 材料与方法
1.1 材料
1.1.1 质粒和细胞 含有HCV全长基因组的质粒pFL-Jc1由美国Apath公司惠赠,Huh7.5.1细胞来自美国斯克利普斯研究所Scott Forrest教授,由中国科学院上海巴斯德研究所钟劲教授提供。
1.1.2 主要试剂 DMEM培养基、FBS和Opti-MEM 购自Gibco公司,IFN-α购自PBL公司,Trizol和 LipofectamineTM2000 购自Invitrogen公司,限制性内切酶Xba、绿豆核酸酶、蛋白酶 K、DNA marker、逆转录试剂盒和SYBR Green Taq试剂盒均购自Takara公司,T7体外转录试剂盒(T7 RiboMAXTMExpress Large Scale RNA Production System)购自 Promega公司,RNeasy Mini Kits购自Qiagen公司。
1.1.3 引物 U6和miR-122的逆转录引物和上下游引物均购自广州锐博公司。GAPDH上游和下游引物分别为GAAGGTGAAGGTC GAGTC和GAAGATGGTGATGGGATTTC;HCV 5'UTR上游和下游引物分别为GCGTTAGTAT GAGTGTCGTG 和 TCGCAAGCACCCTATCAG,均由Invitrogen公司合成。
1.2 方法
1.2.1 HCV pFL-JC1的线性化 取质粒44 μl,加入1 μl限制性内切酶 Xba 及5 μl相应缓冲液,37℃孵育2 h,使质粒线性化,取2 μl反应液以1%琼脂糖凝胶电泳检测酶切消化完全。用酚氯仿法纯化DNA后,分别以绿豆核酸酶和蛋白酶K处理,再用酚氯仿法抽提纯化DNA,用12 μl水溶解 DNA,取1 μl跑电泳检测酶切消化后DNA片段的大小。
1.2.2 体外转录 以2 μg上述线性化的DNA为模板,用promega T7 RiboMAXTMRNA体外转录试剂盒对模板体外转录,反应体系为20 μl,37℃ 孵育 2 h。RNA 产物用 RNeasy Mini Kits纯化,最后用50 μl无RNA酶水洗涤,保存于-80℃。
1.2.3 细胞培养 Huh7.5.1细胞培养于含10%胎牛血清、100 U/ml青霉素和100 mg/ml链霉素的DMEM完全培养基,置于37℃、5%CO2培养箱孵育。
1.2.4 HCV细胞感染模型的建立 转染前1天,以4×105个/孔密度接种12孔细胞培养板,细胞融合度为70% ~80%。转染时,取1.5 μg HCV RNA转录体,用100 μl Opti-MEM无血清培养基稀释,轻轻混匀,室温放置5 min;取2 μl LipofectamineTM2000,用1 00 μl Opti-MEM 无血清培养基稀释,轻轻混匀,室温放置5 min。将稀释的HCV RNA和LipofectamineTM2000一起轻轻混合,室温静置20 min。将200 μl复合物加入12孔板的细胞中,轻轻混匀。6 h后换液,换为完全培养基。3天后,收集上清和未处理的Huh7.5.1细胞一起孵育3h,换为普通DMEM完全培养基,培养3天后即进行后续实验。
1.2.5 IFN-α处理和miR-122 mimics转染本实验中IFN-α的作用终浓度参照文献[9],为1 000 IU/ml,作用时间为48 h。miR-122 mimics的转染步骤同方法1.2.4,转染终浓度和时间分别为20 nmol/L、100 nmol/L和400 nmol/L和72 h。
1.2.6 HCV的逆转录和实时定量PCR 按Trizol法提取总RNA。基因组DNA去除反应包括5 ×gDNA Eraser缓冲液2 μl、gDNA Eraser 1 μl、无 RNA 酶水 3 μl和总 RNA 模板 4 μl,反应条件为42℃ 2 min。逆转录反应体系包括5× 反转录缓冲液 4 μl、反转录酶 1 μl、反转录引物混合液1 μl、无RNA 酶水4 μl和 DNA 去除反应液 10 μl,逆转录条件为 37℃ 15 min,85℃5 s。合成的cDNA作为实时定量PCR模板,反应体系包括2×SYBR Green Taq反应液10 μl、HCV或GAPDH的正反义引物各0.4 μl、Rox 0.4 μl,灭菌蒸馏水 4.8 μl和 cDNA 模板 4 μl。实时定量PCR反应条件为95℃ 30 s,共1个循环;95℃ 5 s,60℃ 34 s,共 40 个循环。IFN-α抑制率(%)=[(IFN-α处理前HCV RNA相对表达量-IFN-α处理后 HCV RNA相对表达量)/IFN-α处理前HCV RNA相对表达量]×100%,用来衡量IFN-α的抗病毒作用。
1.2.7 miRNA的逆转录和实时定量PCR 按Trizol法提取总RNA。逆转录反应体系包括5× 反转录缓冲液 2 μl、逆转录酶 0.5 μl、U6 或miR-122的特异性逆转录引物2 μl、无RNA酶水 3 μl和总 RNA 模板 2.5 μl,逆转录条件为42℃ 15 min,85℃ 5 s。合成的cDNA作为实时定量PCR模板,反应体系包括2×SYBR Green Taq反应液10 μl、miR-122或U6的正反义引物各 2 μl、Rox 0.4 μl,灭菌蒸馏水 1.6 μl 和cDNA模板4 μl,PCR反应条件同方法1.2.6。
1.2.8 统计学分析 用SPSS 11.5软件进行统计学分析,每个实验重复3次,数据以±s表示。多组均数间比较采用One-Way ANOVA,两组均数之间比较采用 student-t检验,P<0.05为差异有统计学意义。
2 结果
2.1 IFN-α对HCV RNA复制的影响 经IFN-α 处理 0.5 h、2 h、8 h、24 h、48 h 后,Huh7.5.1细胞内的HCV RNA分别降至原来的94%、98%、79%、54%、17%(见图1),与 IFN-α 作用呈时间依赖性,在48 h时降至最低,作为后续实验的作用时间。
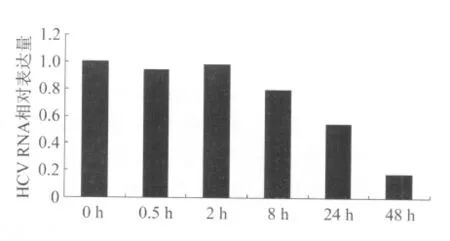
图1 感染HCV的Huh7.5.1细胞经1 000 IU/ml IFN-α处理不同时间后的HCV RNA相对表达量Fig.1 The relative expression levels of HCV RNA at different time points in Huh 7.5.1 cells infected with HCV after treating with 1 000 IU/ml IFN-α
2.2 miR-122对HCV复制的影响 Huh7.5.1细胞感染HCV 72 h后,转染终浓度为20 nmol/L、100 nmol/L 和400 nmol/L 的miR-122 mimics,72 h后检测细胞内的HCV RNA相对表达量分别为12.70±3.75、15.3±1.81和24.9±6.58,均明显高于对照组(P<0.05)。见图2。
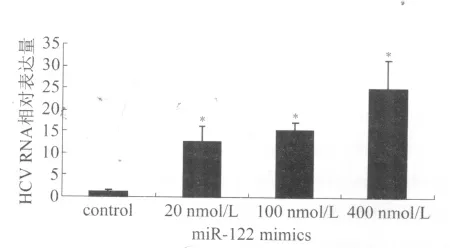
图2 感染HCV的Huh7.5.1细胞转染不同浓度miR-122 mimics后HCV RNA相对表达量Fig.2 The relative expression levels of HCV RNA in Huh7.5.1 cells infected with HCV after transfecting different concentrations of miR-122 mimics
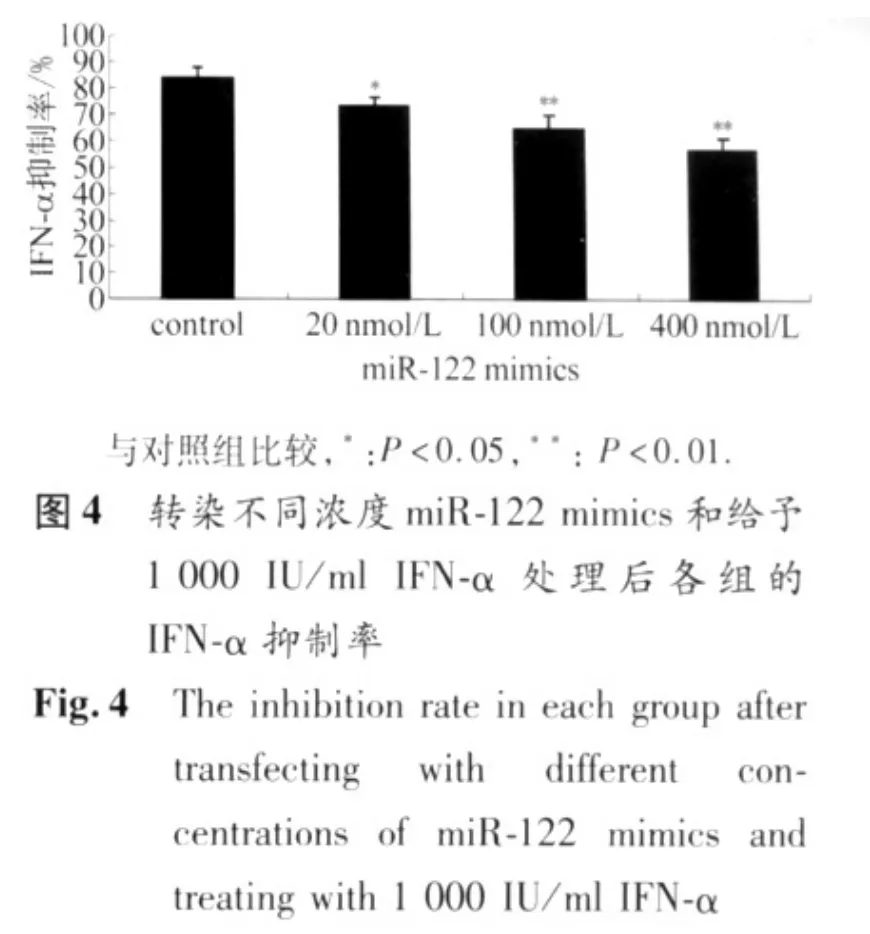
2.3 miR-122对IFN-α抗HCV效应的影响1 000 IU/ml IFN-α作用于转染miR-122 mimics 20 nmol/L、100 nmol/L、400 nmol/L 各组 48 h后,HCV RNA相对表达量分别为3.47±1.34、5.47±1.42、10.33±2.7,随 miR-122浓度增加而增高,并且后两组明显高于对照组(P<0.01),见图3。由于 IFN-α处理前,各组的HCV RNA水平不一样,我们用IFN-α抑制率来衡量IFN-α在各组的抗病毒作用。20 nmol/L组、100 nmol/L组和4 00nmol/L组的IFN-α抑制率分别为73.3% ±3.5%、64.67% ±5.5%和56.33% ±5.1%,明显低于对照组的84% ±4.5%(P<0.05或P<0.01),见图4。
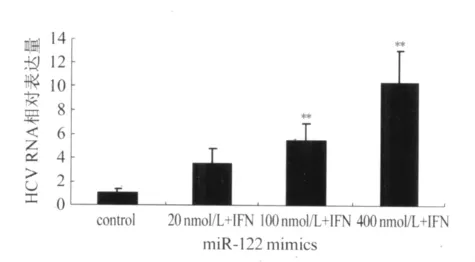
图3 转染不同浓度miR-122 mimics的Huh7.5.1细胞给予1 000 IU/ml IFN-α处理后细胞内的HCV RNA相对表达量Fig.3 The relative expression levels of HCV RNA in Huh7.5.1 cells transfected with different concen-trations of miR-122 mimics after treating with 1 000 IU/ml IFN-α
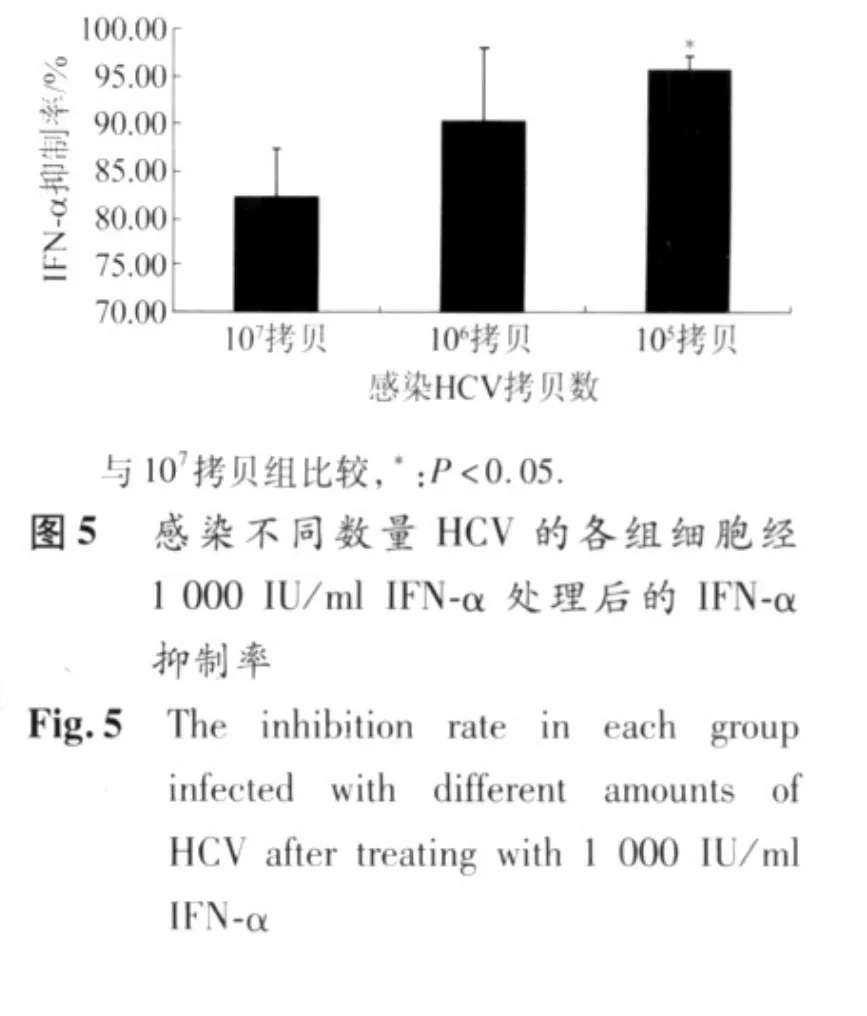
2.4 HCV载量对IFN-α抗病毒作用的影响由于还存在一个变量,即各组的HCV RNA表达量不同,接下来的实验来证明IFN-α抗HCV的作用是否与HCV的病毒载量有关。给Huh7.5.1 分别感染 107拷贝、106拷贝、105拷贝HCV,1 000 IU/ml IFN-α 作用 48 h 后各组的IFN-α抑制率分别为82.33% ±5%、90.33% ±7.6%和95% ±1.5%(图5),105拷贝组 IFN-α抑制率明显高于107拷贝组(P<0.05)。
3 讨论
由于缺乏有效的细胞模型和小动物模型,限制了对HCV生活周期和宿主-病毒之间相互作用的研究以及抗病毒药物和疫苗的研发。到目前为止,关于HCV的研究多仅限于HCV患者[10]和大猩猩[11]。随后在人肝源 Huh7 细胞株建立的HCV复制子系统使得对于HCV翻译和复制的研究成为可能[12-13]。然而,这些复制子并不能产生感染性的 HCV颗粒。Kato等[14-15]人从一名爆发性丙型肝炎患者体内分离出一株HCV全长基因克隆,以之构建的复制子能够在Huh7细胞及其他肝源性细胞和非肝源性细胞中有效的复制。接着,这个研究小组用体外转录的JFH-1 RNA转染Huh7,发现在上清分泌有感染性病毒颗粒。然而,在JFH-1 RNA转染的Huh7细胞中,病毒复制率和感染性差,并且将感染性上清孵育naïve Huh7细胞后,病毒不能复制传代。Blight等[16]人用 IFN-α长期处理含HCV复制子的Huh7细胞,消除HCV复制子,获得对HCV易感的Huh7.5细胞株。Zhong等[17]用 IFN-γ长期处理含有 I/5AGFP-6复制子的Huh7.5细胞,获得对HCV更易感的 Huh7.5.1细胞株。将 JFH-1转染Huh7.5.1细胞,能高效地分泌出感染性HCV颗粒,可重复感染Huh7.5.1细胞和黑猩猩。本研究中,我们利用高复制效能的JC-1株和易感性更高的Huh7.5.1细胞建立的HCV细胞感染模型,更加接近HCV的天然生活周期。由于临床上IFN-α为治疗HCV的主要药物,我们用IFN-α处理此细胞模型,观察其对HCV RNA复制的影响,结果显示,在IFN-α作用0.5h和2h时,HCV RNA没有明显下降,之后,在8h时才开始下降,24h已降至原来的一半,48h时降至17%,以此作为后续实验的时间点。
在本研究中,我们观察miR-122在细胞感染模型中对HCV复制的影响,结果表明,随着miR-122水平的增高,HCV RNA的相对表达量也明显升高,提示在感染模型中,miR-122促进HCV的复制,并且呈浓度依赖性。
我们观察miR-122对IFN-α抗HCV效应的影响,发现:随着miR-122水平的增高,IFN-α抑制率逐渐下降。由于除了miR-122这个变量外,还有一个变量——HCV载量,因此,我们给Huh7.5.1细胞感染不同载量的HCV后,给予IFN-α处理,观察HCV载量对IFN-α抑制率的影响。结果显示,HCV载量影响IFN-α抑制率,感染 HCV载量越多,IFN-α抑制率越低。由此推测,miR-122的表达可部分中和 IFN-α的抗HCV效应。
综上所述,本研究成功构建了HCV细胞感染模型,在此模型中miR-122促进HCV的复制。并且,进一步实验还发现,miR-122的表达可部分中和IFN-α的抗HCV效应。
[1]SKLAN E H,CHARUWORN P,PANG P S,et al.Mechanisms of HCV survival in the host[J].Nat Rev Gastroenterol Hepatol,2009,6(4):217-227.
[2]MODI A A,HOOFNAGLE J H,et al.New therapies for hepatitis C [J].Hepatology,2007,46:615-617.
[3]GE D,FELLAY J,THOMPSON A J,et al.Genetic variation in IL28B predicts hepatitis C treatmentinduced viral clearance [J].Nature,2009,461:399-401.
[4]TANG X P,QIAN K P,YUAN X Z,et al(唐小平,钱可平,袁小珍,等).Relationship between diversity of hepatitis C virus quasispecies and viremia,actinity of liver disease and response to interferon therapy [J].Zhonghua Shiyan He Linchuang Bingduxue Zazhi(中华实验和临床病毒学杂志),2002,16(2):129-132.(in Chinese)
[5]LAGOS-QUINTANA M,RAUHUT R,YALCIN A,et al.Identification of tissue-specific microRNAs from mouse [J].Curr Biol,2002,12(9):735-739.
[6]CHANG J,NICOLAS E,MARKS D,et al.miR-122,a mammalian liver-specific microRNA,is processed from hcr mRNA and may downregulate the high affinity cationic amino acid transporter CAT-1 [J].RNA Biol,2004,1(2):106-113.
[7]ESAU C,DAVIS S,MURRAY S F,et al.miR-122 regulation of lipid metabolism revealed by in vivo antisense targeting[J].Cell Metab,2006,3(2):87-98.
[8]JOPLING C L,SCHUTZ S,SARNOW P,et al.Position-dependent function for a tandem microRNA miR-122-binding site located in the hepatitis C virus RNA genome [J].Cell Host Microbe,2008,4(1):77-85.
[9]HELBIG K J,YIP E,MCCARTNEY E M,et al.A screening method foridentifying disruptionsin interferon signaling reveals HCV NS3/4a disrupts Stat-1 phosphorylation [J].Antiviral Res,2008,77:169-176.
[10]TAKAKI A,WIESE M,MAERTENS G,et al.Cellular immune responses persist and humoral responses decrease two decades after recovery from a single-source outbreak of hepatitis C [J].Nat Med,2000,6(5):578-582.
[11]LOGVINOFF C,MAJOR M E,OLDACH D,et al.Neutralizing antibody response during acute and chronic hepatitis C virus infection [J].Proc Natl Acad Sci USA,2004,101(27):10149-10154.
[12]BLIGHT K J,KOLYKHALOV A A,RICE C M,et al.Efficient initiation of HCV RNA replication in cell culture [J].Science,2000,290(5498):1972-1974.
[13]BUKH J,PIETSCHMANN T,LOHMANN V,et al.Mutationsthatpermitefficientreplication of hepatitis C virus RNA in Huh-7 cells prevent productive replication in chimpanzees[J].Proc Natl Acad Sci USA,2002,99(22):14416-14421.
[14]KATO T,FURUSAKA A,MIYAMOTO M,et al.Sequence analysis of hepatitis C virus isolated from a fulminant hepatitis patient[J].J Med Virol,2001,64(3):334-339.
[15]KATO T,DATE T,MIYAMOTO M,et al.Efficient replication of the genotype 2a hepatitis C virus subgenomicreplicon [J].Gastroenterology,2003,125(6):1808-1817.
[16]BLIGHT K J,MCKEATING J A,RICE C M,et al.Highly permissive cell lines for subgenomic and genomic hepatitis C virus RNA replication[J].J Virol,2002,76(24):13001-13014.
[17]ZHONG J,GASTAMINZA P,CHENG G,et al.Robust hepatitis C virus infection in vitro[J].Proc Natl Acad Sci USA,2005,102(26):9294-9299.
