印度洋深海沉积物中酵母菌IR08的分离与鉴定
2011-06-08赵昌会黄翔玲
赵昌会 ,孙 佳 ,黄翔玲
(1.湖南科技学院,湖南 永州 425100;2.国家海洋局第三海洋研究所,福建 厦门 361005;3.国家海洋局海洋生物遗传资源重点实验室,福建 厦门 361005)
海洋中酵母菌的含量比较低,一般情况下,远海的水体中酵母菌的浓度低于10 cells/dm3,而污染的沉积物中为100 cells/g以上,底质中酵母菌的含量受沉积物类型的影响比较大[1]。可通过浓缩水样或加富培养来分离酵母菌,但对于含量较少、生长较慢的酵母菌株,则需要运用特殊的富集法分离[2]。实验室环境与自然环境的差异,加上采样的困难,限制了人们对深海酵母菌的研究。Nagahama等[3-6]从1 000~11 000 m的深海样品中分离到多种酵母菌,其中一些菌株与已知菌株同源性差异较大,具有研发的新潜力。本文对分离自印度洋中脊底质的深海酵母IR08进行了研究。
1 材料和方法
1.1 材料
1.1.1 供试菌株 菌株IR08由黄翔玲同志采用PDA培养基从印度洋 IR-T3C1站(31.0877°S、59.1124°E)水深4 051 m 的洋底0~3 cm层底质样品中分离获得。
1.1.2 培养基 PDA培养基:马铃薯20%、葡萄糖2%、琼脂1.5%、pH 7.0(深海海水配制);同化碳源基础培养基:MgSO4·7H2O 0.05%、酵母膏 0.02%、(NH4)2SO40.5%、KH2PO40.1%(3.4%(W/V)NaCl溶液配制)。
1.2 方法
1.2.1 深海酵母的显微镜观察 挑取少量的新鲜酵母菌混匀于载玻片的无菌水中,盖上盖玻片,油镜下观察。
1.2.2 深海酵母菌的温度和盐度试验 用接种环挑取少量的酵母接于盐度分别为0、24、34、100的液体培养基中,分别在 4、10、25、35℃下培养(以不加菌液为对照),每隔24 h观察培养液混浊度的变化。
1.2.3 深海酵母菌的碳代谢试验 用接种环挑取少量酵母菌稀释于碳源利用鉴定液体基础培养基中,混匀,取0.4 cm3加到TH04酵母样真菌鉴定板上(杭州微生物试剂有限公司),35℃摇床培养5 d(100 r/min)。
1.2.4 深海酵母菌DNA的提取与扩增 以酵母染色体DNA的提取为参考[7]。
1.2.5 深海酵母菌IR8的鉴定 菌株的染色体DNA 采用特异引物[8]ITS5:5′-GGAAGTAAAAGTCG TAACAAGG-3′和 LR6:5′-CGCCAGTTCTGCTTA-3′,扩增26S rDNA的D1/D2区及ITS区在内的一段核糖体DNA。其PCR扩增反应系统中包括:DNA模板 50 ng、正向及反向引物(各 10-5mol)、Taq DNA聚合酶(4 U)、2.5 mmol/μdm3的 dNTP 5 μL、5 μL 的10×PCR 缓冲液、DDW 补至 50 μL,26S rRNA 基因扩增程序:95℃预热 4 min,94℃变性 1 min、52℃退火1 min、72℃延伸2 min,36个循环,最后 72℃ 10 min。对扩增的26S rDNA片段直接测序,测序引物选用F63、LR3;返回结果通过DNAMAN软件处理,以此为对比目标,通过FASTA网上分析搜索EMBL数据库,寻找同源性最高菌株,采用邻接法[7]构建系统发育树。
2 结果与分析
2.1 IR08的菌落和菌体形态
IR08菌落淡红色,圆形突起,较湿润,直径1~2 mm,易挑取。细胞卵圆形,单端芽殖(如图1)。
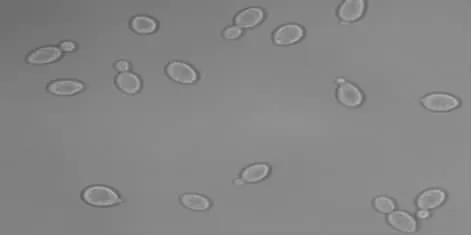
图1 IR08的细胞形态(100×)
2.2 不同温度和盐度对IR08生长的影响
IR08菌株受盐度的影响较小,在25~35℃生长良好,10℃以下生长缓慢(如表1)。
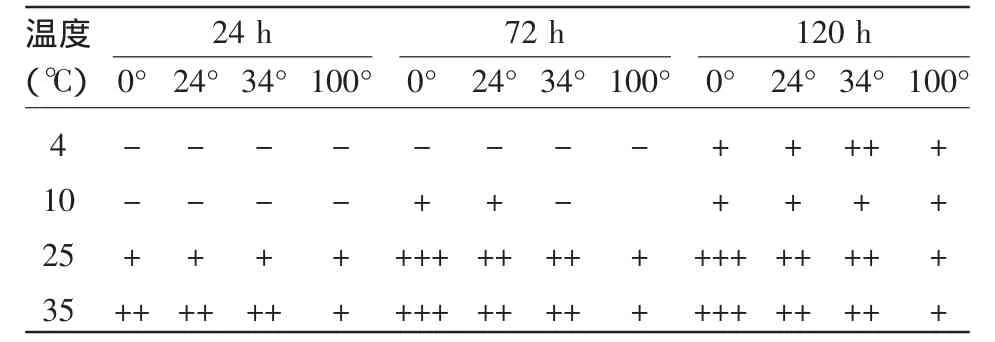
表1 酵母IR08在不同时间、盐度和温度下的生长状况
2.3 IR08的碳源利用特征
IR08除了能利用葡萄糖、蔗糖、麦芽糖及半乳糖外,还能利用尿素、纤维二糖、阿拉伯糖、棉子糖、肌醇和、松二糖、山梨醇等,但不能利用木醇、海藻糖、肌醇、木糖。
2.4 IR08菌株的鉴定
将26S rDNA的D1/D2区的序列测序,输入到EMBL数据库对比,得到rDNA分子同源性相似度最高为 Pichia guilliermondii-AY188372(98.8%),构建的系统发育树如图2。
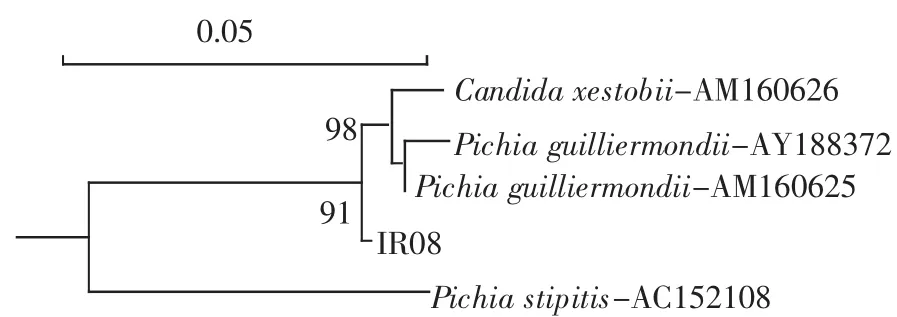
图2 IR08的系统发育树
3 讨论
酵母的鉴定主要借助于生理学特征,但其碳和氮的同化实验常表现易变或区别不明显,如红酵母属、掷孢酵母属、木森顿酵母属、锁掷孢酵母属等,需用ITS和5.8S DNA基因序列测定、18S rDNA及26S rDNA的转录间隔区的基因序列[1],这为发现新种提供了良好的途径。如Nagahama在深海底质中发现新种[6]。
季也蒙毕赤酵母能在含铬量较高的条件下生长,并将其吸收[9]、也可富集重金属铜[10]及防治果实产后的腐烂[11];分离自海洋的季也蒙毕赤酵母具有高产菊粉酶[12]等活性,深海分离的酵母菌IR08在环境修复及果实防腐等方面的价值有待于研究开发。致谢:
本研究在国家海洋局第三海洋研究所叶德赞研究员的实验室完成,对他在本研究中的指导和支持,谨致谢忱!
[1] Nagahama T.Yeast biodiversity in freshwater,marine and deepsea environments(Biodiversity and Ecophysiology of Yeasts)[J].Springer,2006,(3):242-262.
[2] Yanagida F,Kodama K,Shinohara T.Selection of marine yeast stock for making white wine[J].J Brew Soc Jpn,2002,97:150-161.
[3] Nagahama T,Hamamoto M,Nakase T,et al.Kluyveromyces nonfermentans sp.nov.,a new yeast species isolated from the deep sea[J].Int J Syst Bacteriol,1999,49:1899-1905.
[4] Nagahama T,Hamamoto M,Nakase T,et al.Distribution and identification of red yeasts in deep-sea environments around the northwest Pacific Ocean[J].Antonie Van Leeuwenhoek,2001,80:101-110.
[5] Nagahama T,Hamamoto M,Nakase T,et al.Cryptococcus surugaensis sp.nov.,a novel yeast species from sediment collected on the deep-sea floor of Suruga Bay [J].Int J Syst Evol Microbiol,2003,53:2095-2098.
[6] Nagahama T,Hamamoto M,Horikoshi K.Rhodotorula pacifica sp.nov.,a novel yeast species from sediment collected on the deepsea floor of the north-west Pacific Ocean[J].Int J Syst Evol Microbiol,2006,56:295-299.
[7] 骆祝华,王祥敏,黄翔玲,等.热带太平洋深海酵母菌的多样性与碳代谢特征研究.第三届海洋生物高技术论坛 [C].2005:417-423.
[8] Fell J W,Boekhout T,Fonseca A,et al.Biodiversity and systematics of basidiomycetous yeasts as determined by large-subunit rDNA D1/D2 domain sequence analysis[J].Int J Syst Evol Microbiol,2001,50:1351-1371.
[9] Ksheminska H,Jaglarz A,Fedorovych,et al.Bioremediation of chromium by the yeast Pichia guilliermondii:toxicity and accumulation of Cr(III)and Cr(VI)and the influence of riboflavin on Cr tolerance[J].Microbiological Research,2003,158(1):59-67.
[10] María-Isabel S,Luz B,Cristina A,et al.Feasibility of copper uptake by the yeast Pichia guilliermondii isolated from sewage sludge[J].Research in Microbiology,2002,153:173-180.
[11] Yan Z,Kang T,Sicong T,et al.A combination of heat treatment and Pichia guilliermondii prevents cherry tomato spoilage by fungi[J].International Journal of Food Microbiology,2010,137:106-100.
[12] Xinjun Y,Ning G,Zhenming C,et al.Inulinase overproduction by a mutant of the marine yeast Pichia guilliermondii using surface response methodology and inulin hydrolysis[J].Biochemical Engineering Journal,2009,43(3):266-271.
