两株猪圆环病毒2型的分离及基因组序列分析*
2011-05-31王文秀吕素芳沈志强
王文秀,吕素芳,朱 辉,管 宇,魏 凤,唐 娜,沈志强,*
(1.山东省滨州畜牧兽医研究院,山东滨州 256600;2.山东绿都生物科技有限公司,山东滨州 256600)
两株猪圆环病毒2型的分离及基因组序列分析*
王文秀1,吕素芳1,朱 辉2,管 宇1,魏 凤1,唐 娜1,沈志强1,2*
(1.山东省滨州畜牧兽医研究院,山东滨州 256600;2.山东绿都生物科技有限公司,山东滨州 256600)
为比较猪圆环病毒2型毒株的遗传变异特性,从2009年河南郑州和山东东营的猪场疑似猪断奶后多系统衰弱综合征(PMMS)病料中分离到2株病毒,经PCR检测初步鉴定为猪圆环病毒2型,并对病毒全基因组进行扩增,用DNA Star对序列进行比较分析。结果表明,分离到的河南郑州和山东东营PCV-2毒株全基因组长度均为1 767 bp,与国内外参考毒株核苷酸同源性为93.5%~99.6%,两毒株之间核苷酸同源性为95.9%,与PCV-1分离株核苷酸同源性为68.9%~69.3%。本研究中来自两个不同省份的2株PCV-2毒株均属于基因型PCV-2b,且两个PCV-2分离毒株在进化方面存在地域差异。
猪圆环病毒2型;分离;基因组;序列分析
*通讯作者
猪圆环病毒(Porcine circovirus,PCV)最早被视为一种细胞污染物,由 Tischer I等于 1974年在PK-15细胞系中首次发现[1-2]。1982年,Tischer I等用超速离心法提取病毒粒子,并用外切核酸酶S1鉴定核酸类型,用电镜观察核酸状态,首次揭示了猪圆环病毒是一种单链环状DNA病毒,并将其被命名为猪圆环病毒。猪圆环病毒2型(Porcine circovirus type 2,PCV-2)为猪断奶后多系统衰弱综合征(Postweaning multisystemic wasting syndrome,PMWS)的主要病原,1991年在加拿大首次被证实,随后在美洲、欧洲的许多国家和亚洲的一些国家和地区均有报道[3-5]。PCV-2又是引起猪的繁殖障碍、皮炎与肾病综合征和幼龄猪的先天性震颤等的重要致病因子[6],已成为影响养猪业发展的重要传染病病原之一。近年来,对PCV-2遗传变异情况的报道不断增多,PCV-2基因组多为1 767 bp或1 768 bp,而Dupont K等报道了基因组为1 766 bp的新病毒株[7-10]。为了解PCV-2流行毒株的遗传变异特性,本研究对2009年猪场分离到的2株PCV-2进行了全基因测定以及序列分析,为开展疫病流行病学、相关免疫学、遗传进化和疫病防控等研究奠定基础。
1 材料与方法
1.1 材料
1.1.1 病料与细胞 病料分别来自河南郑州、山东东营PMWS仔猪肺、脾和淋巴结,分别命名为HN01株、DY01株,无PCV-1和PCV-2污染的PK-15细胞由广东省农业科学院兽医研究所宋长绪研究员馈赠。
1.1.2 主要试剂、质粒与菌种 rTaq酶、dNTPs、DNA Marker、pMD 18-T Vector、Hind Ⅲ、BamHⅠ内切酶、蛋白酶K、氨苄青霉素等均为宝生物工程(大连)有限公司产品;DNA胶纯化回收试剂盒为百泰克生物技术有限公司产品,宿主菌DH5α由山东省滨州畜牧兽医研究院重点实验室保存;D-氨基葡萄糖为美国Alfa Aesar公司产品。
1.2 方法
1.2.1 引物设计 根据GenBank中发表的PCV-2基因序列(登录号为FJ544563),设计一对特异性引物:上游引物 P1:5′-GAACCGCGGGCTGGCTGAACT TT TGAAAGT-3′,下游引物 P2:5′-GCACCGCGGAAAT TTCTGACAAACGT TACA-3′, 引物由上海生工生物工程技术服务有限公司合成,上下游引物均含有SacⅡ酶切位点。
1.2.2 病料的PCR检测 取疑似PMMS病料充分研磨制成悬液,反复冻融3次,离心取上清200 μL,加入蛋白酶K至终浓度1 mg/L,加入终浓度为100 g/L的SDS,55℃水浴2 h,用苯酚-氯仿抽提,吸取水相加入1/10体积的3 mol/L的NaAc及2倍体积的无水乙醇沉淀1 h,于 4℃条件下 12 000 r/min离心 10 min,加超纯水悬浮沉淀,取 2 μL做PCR模板。
1.2.3 PCR扩增 采用引物对提取的DNA进行扩增,按照下列反应条件和循环参数进行:95℃变性5 min;94℃1 min,58℃1 min,72℃3 min,35个循环,72℃延伸10 min,最后4℃保存。
1.2.4 PCR扩增产物的克隆与酶切鉴定 PCR
产物在8 g/L琼脂糖电泳后,切下含有目的条带的琼脂块,用DNA胶纯化回收试剂盒回收凝胶中的DNA 。取 5 μL 胶回收产物与1 μL pMD 18-T Vector和4 μL SolutionⅠ在 16℃下连接过夜;按常规方法转化DH5α感受态细胞。挑取白色菌落接种LB液体培养基,37℃震摇过夜。以碱裂解法小量堤取质粒。用HindⅢ、BamHⅠ内切酶双酶切鉴定重组质粒。
1.2.5 测序鉴定与分析 将鉴定阳性质粒送至上海生工生物技术服务有限公司进行测序。应用DNA Star软件将获得的2个PCV-2毒株全基因序列及ORF1和ORF2基因序列分别与GenBank中登录的其他序列进行同源性比较分析,并对糖基化位点进行分析
1.2.6 病毒的培养增殖 将组织病料加入1 mL的PBS研磨匀浆,冻融3次,12 000 r/min离心5 min,取上清以0.22 μ m的细菌滤器过滤,加入青霉素(100单位/mL)与链霉素(100 mg/L),4℃作用过夜后与无PCV污染的PK-15细胞进行同步接种,培养1 d后弃上清,用300 mmol/L D-氨基葡萄糖于37℃处理30 min后弃去上清,用D-Hanks洗涤3遍,加入维持液继续培养72 h后收获病毒。以此法继续盲传15代,每3代提取接毒细胞的DNA模板,PCR检测。
2 结果
2.1 样品PCR检测结果
河南郑州和山东东营猪场的疑似PMWS仔猪病料PCR检测均扩增出约1 800 bp的目的片段(图1),与预期大小相符。将检测阳性的病料处理液接种到无PCV污染的PK-15,盲传3代后PCR检测仍为阳性,继续传代,每3代检测1次,直至第10代结果均为阳性。
2.2 测序、同源性比较及序列分析
用DNA Star软件对序列进行拼接和分析表明,分离到两株病毒全基因长度均为 1 767 bp,DY01毒株和HN01毒株的ORF2长度分别为702 bp和705 bp,且已录入GenBank,登录号分别为HM030908和GU574204。
全基因核苷酸同源性分析表明:本次分离毒株(HN01和DY毒株)与GenBank中的PCV-2参考毒株序列中同源性为93.5%~99.6%,与PCV-1(NC001792和 GU371908)毒株序列的同源性为68.9%~69.3%,HN01和DY01毒株相比较发现,HN01与其他毒株同源性相对较低,为 93.5%~95.9%,与东营毒株(DY01)同源性为95.9%,与北京毒株(EU921255、GQ449669-SD-09)同源性为95.9%和95.8%;与湖南毒株(FJ716704)、江苏毒株(FJ644559)同源性分别为95.4%和95.6%;与法国毒株(AY322002)、斯洛伐克毒株(EU545551)和中国台湾毒株(AF166528)同源性为95%~95.8%;与澳大利亚毒株(AY424401)、加拿大毒株(AF086834)、希腊毒株(DQ915586)和韩国毒株(FJ905471)同源性为93.5%~94.7%;其中国外毒株中,HN01株与法国和斯洛伐克毒株同源性相对较高,达95.8%和95.6%。而DY01毒株与其他毒株同源性相对较高,为94.8%~99.6%,同国外毒株同源性分析发现,与法国和斯洛伐克毒株同源性较高,分别为为98.6%和98.4%。本次分离的两个毒株HN01和DY毒株与国内外毒株同源性比较发现,HN01和DY毒株均与北京毒株(EU921255)同源性最高,分别为95.9%和99.6%,而与国外毒株同源性最高的均为法国和斯洛伐克毒株。此外,HN01和DY01毒株与两株PCV-1(美国毒株和天津毒株)同源性仅为68.9%~69.3%(图2)。

图1 PCV-2基因扩增产物电泳结果Fig.1 Electrophoresis of the PCR products of PCV-2 gene
将分离的HN01和DY01株的ORF1和ORF2核苷酸与其他毒株进行比较(表1和表2),结果分离株DY01的ORF1和ORF2与其他毒株均具有较高的同源性,与其他毒株 ORF1的同源性分别为97.5%~99.8%,与PCV-1 ORF1也具有较高同源性,为82.9%;与其他毒株ORF2的同源性为92%~99.6%,与 PCV-1 ORF2同源性为 68.2%;而HN01株的ORF1与其他毒株同源性为89.9%~94.2%,ORF2与其他毒株同源性为 89.9%~94.2%,与PCV-1的ORF1同源性为67.9%。两分离株之间的ORF1同源性为97.2%,ORF2同源性为94.2%。
进化树分析表明,PCV分为 PCV-1和PCV-2两大分支,PCV-2又大致分为2个亚分支,国外毒株(除欧洲外)和Taiwan毒株均位于a分支,而国内分离毒株与欧洲分离毒株均位于b分支,本次分离毒株与国内分离毒株亲缘关系较近,均属于b分支,DY毒株与北京分离毒株亲缘关系最近(图3)。
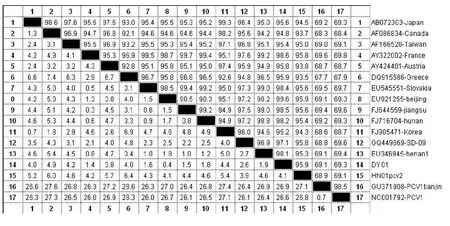
图2 分离毒株全基因核苷酸同源性分析Fig.2 Nucleotide homology comparison of genome DY01 and HN01 strains with other strains
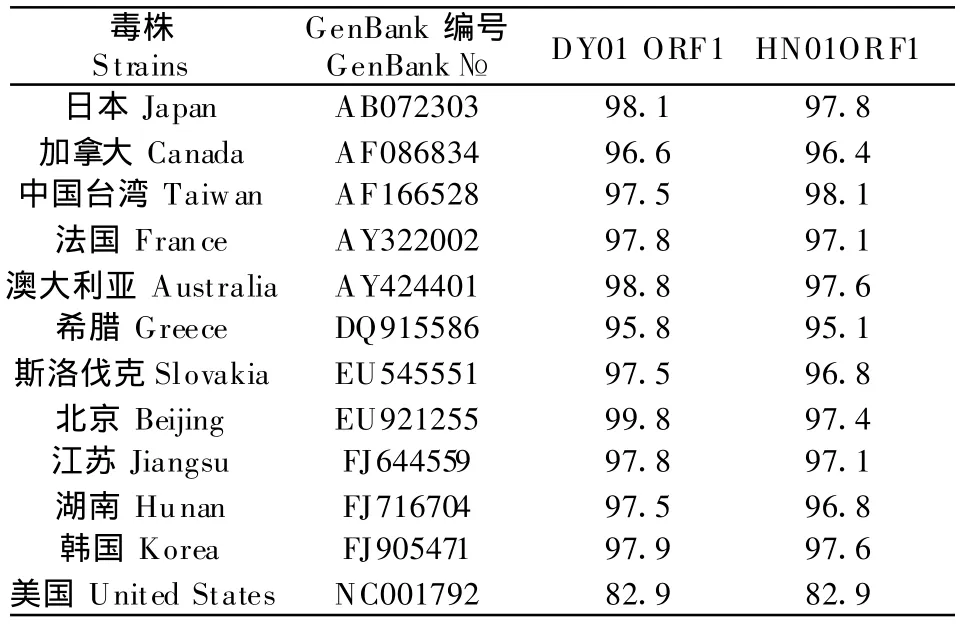
表1 PCV-2 HN和DY株与其他PCV的ORF1核苷酸同源性比较Table 1 Nucleotide homology comparison of ORF1 among PCV-2 HN01,DY01 strain and the other strains
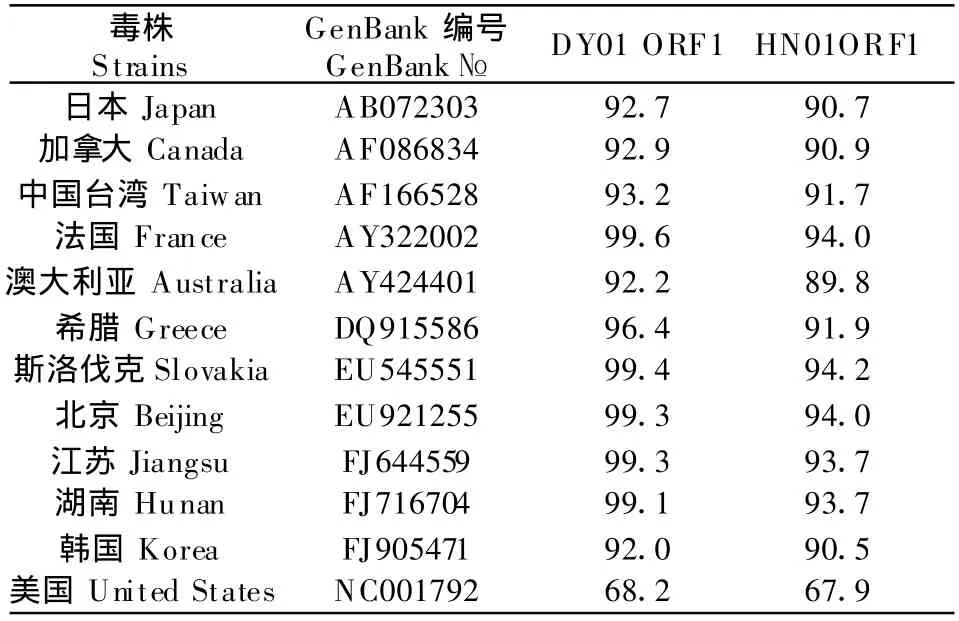
表2 PCV-2 HN和DY株与其他PCV的ORF2核苷酸同源性比较Table 2 Nucleotide homology comparison of ORF2 among PCV-2 HN01,DY01 strain and the other strains

图3 PCV-2分离株的系统发生进化树Fig.3 Phylogenic tree of PCV-2 isolates DY01 and HN01
3 讨论
PCV-2是引起PMWS的主要病因,对养猪业造成了巨大的经济损失。临床上出现的PMWS病例多为PRRSV、PPV等与PCV-2混合感染所致。诸多研究报道PCV-2基因序列在进化上比较保守,但引起的疾病却广泛多样,随着时间的推移,也逐渐发生了变异。
目前,有些学者根据链长度和ORF2内的特殊标记性结构将PCV-2分为两个主要亚型,即PCV-2a型和PCV-2b型。PCV-2a型和PCV-2b型序列相似性约95%;两种亚型的主要区别在ORF2基因,其核酸序列及肽序列的相似性约90%,含有特殊标记性结构PCV-2b型标记性结构为TCAAACCCCCG,PCV-2a型的标记性结构为ACCAACAAAAT很容易进行区分。另一个区别是 PCV-2b全长1 767 bp,而PCV-2a全长 1 768 bp[9,11]。有学者对 PCV-2a和PCV-2b群的毒力进行研究,结果表明两个亚群毒力没有明显差异[12]。然而也有调查研究显示PCV-2b群可引起猪PMWS,而PCV-2a多为带毒而不引起发病[13]。经过对国内外PCV-2分子流行病学分析发现,PCV-2基因型发生了由PCV-2a到PCV-2b的转换,以PCV-2b流行毒株为主[14]。
本研究2009年从来自河南郑州和山东东营疑似PMWS病料分离到2株PCV-2,通过PCR扩增获得了两株病毒株的全基因序列。经序列分析表明,分离的两株病毒株均属于基因型PCV-2b,且与国内外参考毒株核苷酸同源性为93.5%~99.6%,两毒株之间核苷酸同源性为95.9%,与PCV-1分离株核苷酸同源性为68.9%~69.3%。河南毒株HN01与参考序列同源性相对较低,为93.5%~95.9%,而山东分离毒株DY01与参考序列同源性较高,为94.8%~99.6%,亲缘性较近。两个分离毒株与国外毒株同源性比较发现,分离株与法国和斯洛伐克较其他国家毒株具有更高的同源性,表明他们可能来源于同一个祖先。
HN01毒株的ORF2基因含有705个核苷酸,而大多数PCV-2毒株的ORF2为702个核苷酸。从进化树中可看出,HN01在b分支中又自成独立的一支,与参考毒株亲缘关系较远;从序列同源性分析结果来看,HN01毒株全基因序列与参考序列同源性相对较低,其ORF2基因序列也与其他毒株存在较大差异。将分离毒株与参考毒株ORF2的氨基酸进行糖基化位点分析发现,DY01株与参考毒株均在131或132位有一个潜在的糖基化位点,而HN01毒株无糖基化位点,推测该毒株在进化过程中发生变异,导致潜在糖基化位点的缺失。ORF2基因是主要的结构蛋白基因,编码病毒的衣壳蛋白,是主要的免疫原性蛋白,该基因序列出现较大差异及糖基化位点的缺失提示,PCV-2毒株在进化过程中已经开始出现一定程度的变异,变异的发生是否会影响到抗原表位的抗原性或病毒的毒力,尚有待于进一步研究。
[1]Tischer I,Raseh R,T oehtermann G.Characterization of papovavirus-and pieornavirus-like particles in permanent pig kidney cell lines[J].Zentrabl Bakteriol Orig A,1974,226(2):153-167.
[2]T ischer I,Vettermann G W,Koch M A.A very small porcine virus with circular single-stranded DNA[J].Nature,1982,295:64-66.
[3]Allan G M,McNeilly E,Kennedy S,et al.PCV-2 associated PDNS in Northern Ireland in 1990[J].Vet Rec,2000,146(24):7l1-7l2.
[4]Mankertz A,Domingo M,Folch J M,et al.Characterization of PCV-2 isolates from Spain,Germany and France[J].Virus Res,2000,66(1):65-77.
[5]Onuk A,Abe K,T ogashi K,et al.Detection of porcine circovirus from lesions of a pig with wasting disease in Japan[J].J Vet Med Sci,1999,61(10):1119-1123.
[6]Kennedy S,Allan G,McNeilly F,et al.Procine circovirus infection in Northern Ireland[J].Vet Rec,1998,142:495-496.
[7]Dupont K,Nielsen E O,Bakbo P,et al.Genetic analysis of PCV-2 isolates from Danish archives and a current PMWS case control study supports a shift in genotypes with time[J]Vet Microbiol,2008,128:56-64.
[8]Choi K S,Chae J S.Genetic characterization of porcine circovirus type 2 in Republic of Korea[J].Res Vet Sci,2008,84:497-501.
[9]Olvera A,Cortey M,Segales J.Molecular evolution of porcine circovirus type 2 genomes:Phy logeny and clonality[J].Virology,2007,357:175-185.
[10]Shang S B,Jin Y L,Jiang X T,et al.Fine mapping of antigenic epitopes on capsid proteins of porcine circovirus and antigenic phenotype of po rcine circovirus type 2[J].Mol Immunol,2009,46(3):327-334.
[11]Cheung A K,Lager L M,Kohutyuk O I,et al.Detection of two porcine circovirus ty pe 2 genotypic groups in United States swine herds[J].Arch Virol,2007,152(5):1035-1044.
[12]Opriessnig T,Ramamoorthy S,Madson D M,et al.Differences in virulence among porcine circovirus type 2 isolates are unrelated to cluster type 2a or 2b and prior infection provides heterologous protection[J].J General Virol,2008,89(10):2482-2491.
[13]Wiederkehr D D,Sydler T,Buergi E,et al.A new emerging genotypesubgroup within PCV-2b dominatesthe PMWS epizooty in Switzerland[J].Vet Microbiol,2009,136(1/2):27-35.
[14]郭龙军,陆月华.我国部分地区猪圆环病毒 2型分离株的遗传变异分析[J].中国预防兽医学报,2009,31(11):856-859.
Isolation and Genomic Sequence Analysis of Two PCV-2
WANG Wen-xiu1,L¨U Su-fang1,ZHU Hui2,GUAN Yu1,WEI Feng1,TANG Na1,SHEN Zhi-qiang1,2
(1.Shandong Binzhou Animal Science&Veterinary Medicine Institute,Binzhou,Shandong,256600,China;2.Shandong Lvdu Bio-Sciences&Technology Co.Ltd,Binzhou,Shandong,256600,China)
Two porcine circovirus type 2(PCV-2),strains DY01 and HN01 were isolated from highly pathogenic porcine reproductive and respiratory syndrome virus-infected samples.The PCV-2 isolates were identified by PCR method.The complete genomes of these PCV-2 isolates were sequenced and their molecular variations analyzed using DNAStar software.Comparison analysis of the complete genomic sequences of two PCV-2 isolates indicated that they were closely related to each other(95.9%)and to other PCV-2 strains in GenBank(93.5%-99.6%).There was 68.9%to 69.3%sequence identity among PCV-2 and PCV-1 isolate genes.Phylogenetic analysis revealed that the two isolates belonged to genotype PCV-2b.This two PCV-2 strains isolated from Henan and Shandong provinces were both belong to PCV-2b type,and this two PCV-2 strains have area lvariation on gene evolution.
Porcine circovirus type 2;isolation;genome;sequence analysis
S852.659.2
A
1007-5038(2011)09-0005-05
2011-05-11
山东省自主创新成果转化重大专项计划(2008ZHZX1A1103)
王文秀(1978-),女,新疆伊宁人,博士,助理研究员,主要从事分子病原学与细胞生物学研究。
