双齿围沙蚕CYP4基因的克隆及序列分析
2011-03-20周一兵陈雪杨大佐万良王斌王丽丽孙静波
周一兵,陈雪,杨大佐,万良,王斌,王丽丽,孙静波
(1.大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,辽宁大连116023;2.国家海洋环境监测中心,辽宁大连116023;3.宝生物工程(大连)有限公司,辽宁大连116600)
双齿围沙蚕CYP4基因的克隆及序列分析
周一兵1,陈雪1,杨大佐1,万良1,王斌1,王丽丽2,孙静波3
(1.大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,辽宁大连116023;2.国家海洋环境监测中心,辽宁大连116023;3.宝生物工程(大连)有限公司,辽宁大连116600)
根据已知的绿沙蚕Nereis virens细胞色素氧化酶CYP342A1保守区设计引物,采用RACE技术首次从双齿围沙蚕Perinereis aibuhitensis体壁肌肉中克隆出细胞色素氧化酶CYP4基因全长序列。分析结果表明,该序列全长为1 857 bp,5′端非翻译区为186 bp,3′端非翻译区为225 bp,阅读框为1 446 bp,编码为481个氨基酸。第422~431氨基酸序列符合P450所具有的结构保守共有序列FxxGxxxCxG,第319~322氨基酸序列为K螺旋特征基序ExxR区,第282~294氨基酸序列含有CYP4家族特征序列EVDTFMFEGHDTT。经Blast与GenBank中已知物种的氨基酸序列进行同源性比对,CYP4与多毛类绿沙蚕Nereis virens CYP4BB1、CYP342A1(AY453408)氨基酸同源性为73%、36%,与多毛类小头虫Capitella capitata CYP4AT1 (AY574044)同源性为39%。据此推断,双齿围沙蚕CYP4基因属于CYP4B亚家族。
双齿围沙蚕;CYP4;RACE;生物信息学分析
细胞色素P450酶系(CYP酶)是存在于所有生物体中的超基因家族,其中CYP4是细胞色素P450酶系中的一个重要家族。研究表明,CYP4与多种生物代谢、解毒和抗药性机制相关[1-4]。细胞色素氧化酶P450家族是多毛类体内生物转化的第一阶段代谢酶,在外源性化合物代谢中起着极其重要的作用,这与脊椎动物体内的生物转化相似[5]。有报道指出,无脊椎动物亦具有AhR的同构物,并具有前者的典型特征,但其对芳香族化合物尚缺乏高特异性的结合能力,这与脊椎动物明显不同[4]。关于细胞色素P450基因特性的研究,无论是对其基因转录调控还是对其特定的生化活性在海洋无脊椎动物尤其是多毛类沙蚕的报道都是十分有限的。因此,应用分子生物学方法确定和研究海洋无脊椎动物CYP酶系组成的多样性及其功能特征,既能克服方法学上存在的问题,又可有效地揭示海洋无脊椎动物CYP酶系不同家族之间结构多样性的特征。迄今为止,在多毛类中已克隆出4种P450基因,包括2004年克隆出绿沙蚕Nereis virens CYP4家族(CYP342A1)及其亚家族成员(CYP4BB1)[4],还有2005年公布的小头虫Capitella capitate CYP4AT1和CYP331[6]。
双齿围沙蚕Perinereis aibuhitensis广泛分布于沿海潮间带及河口滩地,既是重要的经济多毛类动物,又是外源性化学物质生物转化机制中重要的决定性物种。因此,克隆沙蚕CYP4基因,研究其表达与调控规律,既有助于揭示沙蚕对芳香族化合物的代谢特征和解毒机制,也可为建立以沙蚕健康评价为基础的海洋沉积环境早期污染生态风险评价的基因诊断方法提供依据。本研究中,作者采用RTPCR和RACE法,首次获得完整的双齿围沙蚕CYP4基因序列,以期为今后进一步研究该基因在受到有机污染物胁迫下的表达规律奠定基础。
1 材料与方法
1.1 材料
研究用双齿围沙蚕采自辽宁省盘锦双台子河口,挑选3尾体质量为1.5~2.0 g的个体,将其放置在加冰的泡沫保温箱内运送到大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室暂养,每天更换一次海水。试验所用海水取自大连市
黑石礁海区,经沉淀、砂滤后贮存备用。试验海水中Cu(Ⅱ)、Hg(Ⅱ)、Zn(Ⅱ)、Pb(Ⅱ)和Cd(Ⅱ)的背景浓度分别为0.38、0.01、75、0.27、0.06 μg/L,盐度为31~32,pH为8.25± 0.10,水温为(18±0.5)℃。
1.2 方法
1.2.1 总RNA的提取 研钵使用液氮预冷,取0.1 g双齿围沙蚕体壁肌肉,迅速投入预冷研钵中,加入适量液氮并研磨成粉末状,再按照RNAiso Pius(购自TAKARA公司)说明书提取总RNA,最终溶于DEPC处理水中。经15 g/L琼脂糖凝胶电泳检验总RNA的完整性,使用紫外分光光度计测定其浓度。于-80℃下保存,备用。
1.2.2 引物设计 根据近源物种Nereis virens细胞色素氧化酶CYP342A1 CDs(登录号AY453408)找到保守区域设计一对简并引物,以双齿围沙蚕的总RNA为模板,反转录扩增出特异性片断,并对其进行序列测定和比对。根据得到的CYP4基因片段设计3′RACE和5′RACE引物。
1.2.3 cDNA第一链合成及特异性片段扩增 用已提取的双齿围沙蚕总RNA为模板进行反转录,按照Takara RNA PCR Kit(AMV)Ver.3.0试剂盒(购自TAKARA公司)说明合成cDNA第一链及PCR反应。cDNA第一链合成反应条件:30℃下反应10 min,42℃下反应30 min,99℃下反应5 min,5℃下反应5 min。特异性片段PCR反应条件:94℃下预变性2 min;94℃下变性30 s,47℃下退火30 s,72℃下延伸30 s,共进行32个循环;72℃下再延伸10 min。
取5 μL PCR产物于10 g/L琼脂糖凝胶电泳检验PCR扩增效果后,再用EZ Spin Column DNA Gel Extraction Kit切胶回收试剂盒(购自上海生工有限公司)切胶回收。回收后的产物与TaKaRa pMDTM19-T Vector载体连接进行TA克隆,热转化至E.coli Competent Cells JM109中,涂布于Amp/X-gal/IPTG/LB平板,37℃下过夜培养。挑选独立5株阳性菌斑加入至LB液体培养基(含Amp)中培养16 h,送由上海生工有限公司测序。
1.2.4 RACE扩增 使用Clontech SMARTTMRACE cDNA Amplification Kit(购自Clontech公司)试剂盒,按照其说明书进行3′RACE扩增。3′RACE PCR反应条件:94℃下变性30 s,72℃下延伸3 min,共进行5个循环;94℃下变性30 s,70℃下退火30 s,72℃下延伸3 min,共进行5个循环; 94℃下变性30 s,68℃下退火30 s,72℃下延伸3 min,共进行25个循环。
使用TaKaRa LA Taq®with GC Buffer、TaKaRa 5′-Full RACE Kit(购自TAKARA公司)试剂盒,按照其说明书对总RNA进行去磷酸化处理、“去帽子”反应,并与5′RACE Adaptor连接后,反转录合成5′RACE的cDNA,再进行PCR扩增。5′RACE PCR反应条件:94℃下预变性3 min;94℃下变性30 s,53℃下退火30 s,72℃下延伸1 min,共进行35个循环;72℃下再延伸10 min。
序列测定由TAKARA公司完成,应用DNA Star软件将得到的序列拼接成完整的cDNA序列。
1.2.5 序列分析 将推导的氨基酸序列使用Blast进行同源性比对,用DNA Star预测等电点、相对分子质量,进行TMHMM跨膜区分析、Motif Scan氨基酸MOTIF分析、PSORT功能位点分析,用SignalP预测信号肽,再进行ExPASy-ProtScale疏水性分析、SOMPA蛋白二级结构预测、Clustal W氨基酸序列多重比对,并使用Mega 4.0软件构建同源关系进化树。
2 结果与分析
2.1 特异性片段扩增及RACE扩增
提取的总RNA经15 g/L琼脂糖电泳检查,可清晰地看到5S、18S和28S核糖体RNA条带(图1 -a)。结果显示,OD260nm/OD280nm值为1.86~1.88,RNA完整性和均一性好,无降解现象。
根据近源物种Nereis virens CDs找保守区域设计引物,经PCR产物割胶后克隆测序得到442 bp的特异性片段条带(图1-b)。通过优化PCR反应条件,用3′RACE PCR扩增得到371 bp核苷酸序列(图1-c),用5′RACE PCR扩增获得一段1 044 bp核苷酸序列(图1-d)。经DNA Star序列拼接,得到1 857 bp完整的双齿围沙蚕CYP4 cDNA全序列,并提交GenBank,登录号为HM126463。
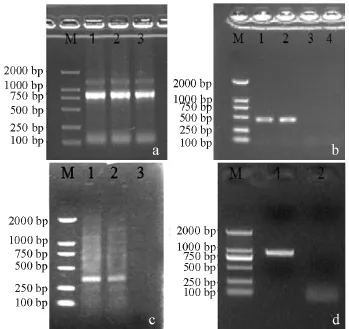
图1 双齿围沙蚕RNA提取结果及CYP4 PCR扩增产物电泳图Fig.1 The total RNA and PCR amplified product of CYP4 from P.aibuhitensis
2.2 序列分析
序列分析结果显示,双齿围沙蚕CYP4基因为P450家族的新成员,其cDNA包括5′非翻译区186 bp,3′非翻译区225 bp,开放阅读框为1 446 bp,编码为481个氨基酸(图2)。CYP4基因的理论等电点为7.25,相对分子质量为55 726.61。对SignalP信号肽和信号肽位点的分析结果显示,双齿围沙蚕CYP4蛋白没有明显的信号肽位点,可能为非分泌蛋白。采用TMHMM对CYP4氨基酸序列的跨膜区进行分析,CYP4蛋白无明显的跨膜区,推测其可能是可溶性蛋白。根据ProtScale预测结果,CYP4基因编码的氨基酸序列疏水性最大为2.70,最小值为-3.40,均值为-0.86。整个多肽链中大多数氨基酸的分值较低,亲水性氨基酸多于疏水性氨基酸,据此推断该蛋白为亲水性的。用SOMPA预测双齿围沙蚕CYP4基因编码的蛋白二级结构中含α-螺旋为43.66%,β转角为5.82%,延伸链为13.10%,无规则卷曲为37.42%。由CYP4 cDNA推导的氨基酸序列中,第282~294氨基酸序列含有CYP4家族特征序列EVDTFMFEGHDTT,其中M由L代替,而谷氨酸残基(E)只存在于CYP4家族中;第319~322氨基酸序列为K螺旋特征基序ExxR区;血红素结合区保守序列为FxxGxxxCxG,第422~431氨基酸序列符合P450所具有结构保守的血红素结合区共有序列。
将双齿围沙蚕CYP4序列提交到NCBI,使用在线Blast进行同源性比较。结果表明,CYP4基因氨基酸序列与多毛类绿沙蚕Nereis virens CYP4BB1、CYP342A1的同源性分别为73%、36%,与多毛类小头虫Capitella capitata CYP4AT1的同源性为39%。与软体动物袖扣海兔螺Cyphoma gibbosum CYP4BK1、菲律宾蛤仔Venerupisphilippinarum CYP4、栉孔扇贝Chlamys farreri CYP4,环节动物囊舌虫Saccoglossus kowalevskii CYP4-like,两栖动物非洲爪蟾Xenopus(Silurana)tropicalis CYP4B1-like以及哺乳动物人Homo sapiens CYP4F、苏门达腊猩猩Pongo abelii CYP4F12、褐家鼠Rattus norvegicus CYP4F1等推导的氨基酸序列同源性为42%~51%。将双齿围沙蚕CYP4氨基酸序列与Gen-Bank数据库进行比较,经Clustal W进行同源性比对分析,结果见图3。应用NJ法构建的同源关系树,结果见图4,其中Bootstraping值为1000。综上所述,推断双齿围沙蚕CYP4基因为CYP4B亚家族新成员。
3 讨论
本试验中采用RACE扩增方法得到多毛类双齿围沙蚕CYP4cDNA全长序列。该序列全长为1 857 bp,开放阅读框为1 446 bp,编码为481个氨基酸。以往的研究表明,疏水螺旋I区(EVDTFMFEGHDTT)、K螺旋区(ExxR)和血红素结合区(FxxGxxxCxG)普遍存在于P450家族中,这些被认为是鉴定P450家族的特征序列[7-9]。本试验中由所获得的CYP4 cDNA推导的氨基酸序列,发现第282~294氨基酸序列具有疏水螺旋I区,其中M由L代替;第319~322氨基酸序列存在K螺旋特征基序ExxR区;CYP4第422~431氨基酸序列符合P450所具有的结构保守共有序列血红素结合区。保守区非功能的个别氨基酸残基变异属于正常现象,并不影响该保守区域的鉴定。

图2 双齿围沙蚕CYP4编码基因全长cDNA及推导的氨基酸序列Fig.2 Nucleotide and deduced amino acid sequence of Perinereis aibuhitensis CYP4

图3 双齿围沙蚕CYP4氨基酸序列与其它动物CYP4基因家族推导的氨基酸序列的比对分析(由Clustal W软件分析获得)Fig.3 The alignment of deduced protein sequence of CYP4 with seleceted amino acid sequences was performed using Clustal W
细胞色素P450是一个成员众多的超基因家族,基于PCR的分子克隆技术可以有效地鉴定其家族基因组成的多样性,近年来已确定了一系列CYP酶系的新基因。Rewitz等[4]最先从多毛类绿沙蚕中
鉴定出两种具有一定分化的CYP cDNA序列。分析表明,它们均属于CYP4家族,其最大的相似性表现为均具有谷氨酸残基(E),被认为这是CYP4家族的典型特征。另外,根据国际细胞色素P450命名委员会命名规则,如果CYP氨基酸序列与CYP4酶系的同源性>40%,视为CYP4同一家族;在此基础上,相同氨基酸序列的同源性>55%,便可认定其属于CYP4家族中的亚家族成员[10]。迄今为止,在多毛类中已克隆出5种P450基因,如Rewitz等[4]从绿沙蚕肠道中分离出CYP4家族CYP342A1及其亚家族成员CYP4BB1,二者的氨基酸序列与哺乳动物CYP4F酶具有极高的同源性,分别达到61%和58%。环节动物的P450酶基因主要是从肠道微粒体中分离出来,而本试验中首次从双齿围沙蚕体壁肌肉中获得CYP4基因,说明该基因存在部位具有广泛性。本试验中得到的CYP4 cDNA序列全长编码为481个氨基酸,根据同源关系分析,双齿围沙蚕CYP4与绿沙蚕Nereis virens CYP4BB1的同源性最高为73%;与软体动物Cyphoma gibbosum CYP4BK1同源性为48%;与两栖动物Xenopus(Silurana)tropicalis CYP4B1同源性为43%;与哺乳动物的CYP4F亚家族的同源性为42%~47%。据此可推断,本试验中由双齿围沙蚕所得到的CYP4基因属于CYP4B亚家族成员,同时同源关系树也表明多毛类CYP4家族更接近CYP4F亚家族,这与Rewitz等[4]得到的结论是一致的。
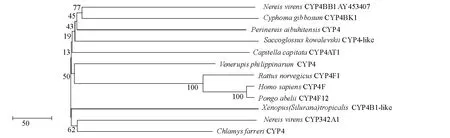
图4 根据CYP4氨基酸序列构建的同源关系树Fig.4 Phylogenetic neighbour-joining tree of the 481 amino acid sequences of CYP4 with the corresponding regions of 11 representatives from other species
目前,关于脊椎动物中CYP酶对污染物代谢、去除作用的研究较为详尽,但对无脊椎动物尤其对海洋无脊椎动物的了解还十分有限。有一种观点认为,CYP酶系是海洋多毛类对多环芳烃(PAH)生物转化代谢途径中的重要酶类,与脊椎动物相似,PAH亦可能通过激活沙蚕体内相关受体,进而诱导CYP基因表达[10]。海洋多毛类绿沙蚕及小头虫在受到持久性有机污染物多环芳烃等胁迫下,其P450基因表达量提高2~3倍[4,6]。关于其它海洋无脊椎动物细胞色素P450基因表达水平也有类似的报道[11-15]。这表明无脊椎动物的细胞色素P450基因转录水平稍高于脊椎动物,但是多环芳烃与芳香烃受体(AhR)相互作用导致CYP1A基因转录水平的上涨是原来的10~100倍[16]。无脊椎动物的细胞色素P450基因转录、翻译的基本分子机制尚未阐明,但P450基因的表达调控有可能与脊椎动物类似,无脊椎动物也具有受体和相应的表达调控原理,如金属反应转录因子(MTF1)[17]和维甲类X受体(RXR)[18]与脊椎动物类似。本研究中利用RT-PCR和RACE技术对双齿围沙蚕CYP4基因进行了克隆,获得了1 857 bp的双齿围沙蚕CYP4 cDNA序列,既可揭示以沙蚕为代表的多毛类动物CYP4不同亚家族结构的多样性,又为进一步了解CYP4基因在沙蚕中的本底表达以及污染物暴露条件下的表达量变化奠定了基础。
[1] Simpson A E C M.The cytochrome P450 4(CYP4)family[J]. General Pharmacology,1997,28:351-359.
[2] 冷欣夫,邱星辉.细胞色素P450酶系的结构、功能与应用前景[M].北京:科学出版社,2001.
[3] Parmentier J H,Lavrentyev E N,Falck J R,et al.Evaluation of cytochrome P450 4 family as mediator of phospholipase dectivation in aortic vascular smooth muscle cells[J].Life Sciences,2005,77 (9):1015-1029.
[4] Rewitz K,Kjellerup C,Jogensen A,et al.Identification of two Nereis virens(Annelida:Polychaeta)cytochromes P450 and induction by xenobiotics[J].Comparative Biochemistry and Physiology Part C:Toxicology&Pharmacology,2004,138:89-96.
[5] Jogensen A,Giessing A M B,Rasmussen L J,et al.Biotransformation of the polycyclic aromatic hydrocarbon pyrene in the marine polychaete Nereis virens[J].Environmental Toxicology and Chemistry,2005,24:2796-2805.
[6] Li B,Bisgaard H C,Forbes V E.Identification and expression of two novel Cytochrome P450 genes,belonging to CYP4 and a new CYP331 family,in the polychaete Capitella capitata sp.[J].Biochemical and Biophysical Research Communications,2004,325: 510-517.
[7] Bradfield J Y,Lee Y H,Keeley L L.Cytochrome P450 family 4 in a cockroach:molecular cloning and regulation by hypertrehaolosemic hormone[J].Proceedings of the National Academy of Sciences of the United States of America,1991,88(10):4558-4562.
[8] Feyereisen R.Insect cytochrome P450[M]//Comprehensive Molecular Insect Science.Amsterdam:Elsevier,2005.
[9] Jiang H B,Wang J J,Liu G Y,et al.Molecular cloning and sequence analysis of a novel P40 gene encoding CYP345D3 from the red flour beetle,Tribolium castaneum[J].Journal of Insect Science,2008,8:1-7.
[10] Jogensen A,Anders M B,Giessing C,et al.Biotransformation of polycyclic aromatic hydrocarbons in marine polychaetes[J].Marine Environmental Research,2008,65(2):171-186.
[11] Vigano L,Arillo A,Faugl C,et al.Biomarkers of exposure and effect in flounder(Platichehys flesus)exposed to sediment of the Adriatic sea[J].Marine Polution Bulletin,2001,42(10):887-894.
[12] Livingstone D R.The fate of organic xenobiotics in aquatic ecosystems:quantitative and qualitative differences in biotransformation by invertebrates and fish[J].Comparative Biochemistry and Physiology—Part A:Molecular&Integrative Physiology,1998,120(1): 43-49.
[13] Livingstone D R,Martinez P G,Michel X,et al.Oxyradical production as a pollution-medicated mechanism of toxicity in the common mussel,Mytilus edulis L.,and other molluscs[J].Functional Ecology,1990,4:415-424.
[14] Snyder M J.Cytochrome P450 enzymes belonging to the CYP4 family from marine invertebrates[J].Biochemical and Biophysical Research Communications,1998,249:187-190.
[15] Snyder M J.Cytochrome P450 enzymes in aquatic invertebrates: recent advances and future directions[J].Aquatic Toxicology, 2000,48:529-547.
[16] Rewitz K,Styrishave B,Andersen O.CYP330A1 and CYP4C39 enzymes in the shore crab Carcinus maenas:sequence and expression regulation by ecdysteroids and xenobiotics[J].Biochemical and Biophysical Research Communications,2003,310:252-260.
[17] Zhang B,Egli D,Georgiev O,et al.The Drosophila homolog of mammalian zinc finger factor MTF-1 activates transcription in response to heavy metals[J].Molecular and Cellular Biology,2001, 21:4505-4514.
[18] Wu X,Hopkins P M,Palli S R,et al.Crustacean retinoid-X receptor isoforms:distinctive DNA binding and receptor-receptor interaction with a cognate ecdysteroid receptor[J].Molecular and Cellular Biology,2004,218:21-38.
Cloning and sequence analysis of cytochrome CYP4 family in polychaete Perinereis aibuhitensis
ZHOU Yi-bing1,CHEN Xue1,YANG Da-zuo1,WAN Liang1,WANG Bin1,WANG LI-li2,SUN Jing-bo3
(1.Key Laboratory of Marine Bio-resources Restoration and Habitat Reparation in Liaoning Province,Dalian Ocean University,Dalian 116023,China; 2.National Marine Environmental Monitoring Center,Dalian 116023,China,3.Takara Biotechnology(Dalian)Co,LTD,Dalian 116600,China)
In the present study,the partial cDNA of CYP4 was amplified from the total RNA of epidermis muscle of the polychaete,Perinereis aibuhitensis,by reverse transcription with the primers based upon the CYP342A1 from Nereis virens.Then other primers were designed according to the partial gene of Perinereis aibuhitensis CYP4,and two fragments about 371 bp and 1044 bp were obtained by using 3′RACE and 5′RACE.Sequence analysis revealed a 1857 bp cDNA containing the 186 bp 5′-untranslated region,225 bp 3′-untranslated regions and 1 446 bp open reading frame encoding 481 amino acid.The identity rate of deduced amino acid sequence of CYP4 was subjected to Blast searching in NCBI.The results showed that the CYP4 shared 73%identity with CYP4BB1,36%identity with CYP342A1 of Nereis virens and 39%identity with CYP4AT1 of Capitella capitate.The deduced amino acid sequence of CYP4 contained the conserved motifs of P450 family(FxxGxxxCxG)and had K-helix motifs of P450 family(ExxR).In addition,it has the characteristic sequences of CYP4 family(EVDTFMFEGHDTT).So it is inferred that the cDNA sequence might belong to the subfamily of CYP4B.
Perinereis aibuhitensis;CYP4;RACE;sequence analysis
Q78
A
2095-1388(2011)06-0507-07
2011-02-08
国家“863”计划项目(2006AA10Z410);国家海洋公益性行业科研专项(200805069);国家自然科学基金资助项目(30901107,41106115)
周一兵(1957-),男,教授。E-mail:ybzhou@dlou.edu.cn
