植物乳杆菌中胆盐水解酶结构基因的克隆与序列分析
2011-03-13李蓉于长青
李蓉,于长青
(黑龙江八一农垦大学食品学院,大庆163319)
胆盐水解酶(bile salt hydrolase,BSH)是由bsh基因编码的一种胞内酶[1-2],是微生物在生长和繁殖过程中所产生的代谢产物,微酸环境下的最适pH值一般在5和6之间,对氧气不敏感,该酶已经从多种微生物中得到了分离和鉴定[3-4]。目前,发现的产胆盐水解酶的菌株都是革兰氏阳性菌,其中包括部分的益生菌株,如:乳酸菌、双歧杆菌等该酶主要由乳酸菌产生。BSH可能对胆固醇和氨基酸基团(甘氨酸/牛磺黄氨酸)都具有识别性,有文献报道BSH酶能识别胆酸酯基团。Moser和Savage[5]的研究发现JCM1069布氏乳杆菌表达牛磺酸脱氧胆汁酸水解酶但不表达牛磺酸胆汁酸水解酶。BSH能水解结合态牛磺酸胆盐和甘氨酸胆盐,将其转变成氨基酸和游离胆酸。
血清中高水平的胆固醇是诱发高血压、冠心病等众多心血管疾病的重要因素[6-7],所以如何控制血清胆固醇水平已经受到越来越广泛的关注。药物治疗存在药费昂贵,毒副作用较大、治疗效果不明显等缺陷。目前,口服益生菌是研究的热点,它的治疗效果明显,能够显著地降低血清胆固醇的含量,对高血脂的防治有积极的作用[8-9]。BSH酶能水解结合态牛磺酸胆盐和甘氨酸胆盐,转变成氨基酸和游离胆酸,作用后产生的去结合型胆盐比结合型胆盐重吸收效率低,致使大量的去结合型胆盐从粪便中排出。去结合型胆盐的大量流失使得体内需要消耗更多的胆固醇重新合成胆酸盐来弥补损失,从而逐步使血清胆固醇水平降低[10-11]去结合胆盐能够降低血清中胆固醇的含量,主要通过以下两个途径:一是通过合成胆酸来补充那些随粪便排出的胆酸来增加胆固醇的需求量;二是降低了胆固醇的溶解度,减少了肠腔对胆固醇的吸收,使胆固醇与脱结合胆盐一同沉淀下来。
BSH是绝大部分乳杆菌和大量益生菌产生的胞内酶,目前有关胆盐水解酶的研究报道相对较少,国内的研究主要集中在降胆固醇机理、高产BSH酶优良菌株的筛选、BSH酶的纯化等方面;而国外也则正在进行有关BSH酶微生物分子生物学等方面的研究,但还至今未见有突破性进展。研究主要将植物乳杆菌中的BSH结构基因进行克隆,为BSH基因的功能及基因工程的进一步研究奠定基础。
1 实验材料与方法
1.1 菌种及质粒
植物乳杆菌Lp M1-UVS29、E.coliDH5α由黑龙江八一农垦大学食品学院保存;pMD18-T载体购自宝生物工程(大连)有限公司。
1.2 试剂
DNA Ladder 2000、rTaqDNA聚合酶、NcoⅠ和Hind III等限制性内切酶及T4 DNA连接酶均购自TaKaRa公司;质粒小量提取试剂盒和DNA凝胶回收纯化试剂盒均购自博大泰克公司;琼脂粉、琼脂糖、氨卞青霉素(Amp)、胰蛋白胨、酵母提取物等购自上海生工公司;SDS购于Sigma公司。
1.3 引物的设计与合成
参照Genbank中已报道的胆盐水解酶全长基因序列(EU477374),利用Primer 5软件设计了一对扩增BSH酶编码区序列的特异性引物,引入酶切位点Hind III/NcoI,引物由上海生工生物工程有限公司合成。上游引物:5'AAT CCA TGG ATG TGT ACT GCC ATA ACT TAT C 3';下游引物:5'AAT GAG CTC TAT TAG TTA ACTGCA TAG TAT TGTG 3'。
1.4 BSH基因的PCR扩增
采用CTAB法[12]提取植物乳杆菌基因组DNA,以DNA为模板,进行PCR扩增BSH结构基因,PCR反应体系为2.5μL 10×PCR Buffer,2μL dNTPs,1μL模板DNA,上下游引物分别为0.5μL,0.2μL TaqDNA聚合酶,最后加ddH2O至终体积为25μL。PCR反应条件为:95℃预变性5 min,94℃变性50 s,48℃退火50 s,72℃延伸1 min,经过30个循环,72℃再延伸10min[12]。
1.5 目的基因的克隆及测序
利用凝胶回收试剂盒纯化回收PCR产物,与pMD18-T载体连接,16℃过夜。将连接产物转化至感受态E.coliDH5α,37°培养12~16 h。经蓝白筛选获得阳性重组子命名为pMD18-BSH;进行质粒小量提取,经PCR(反应体系和反应条件同上)以及双酶切鉴定,筛选阳性重组子,最后送至上海生工生物工程技术服务公司进行测序,将获得DNA序列与Genbank数据库中公布的已知功能基因进行序列同源性比较以鉴定和获得该DNA序列的功能信息。
2 结果与分析
2.1 PCR扩增BSH结构基因
取5μL PCR扩增BSH结构基因的产物经1%琼脂糖凝胶电泳分析,在接近1 000 bp处可见特异性目的条带,见图1。
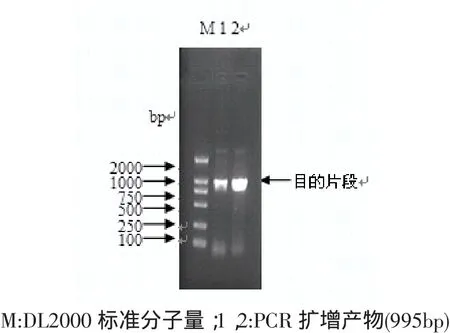
图1 BSH基因PCR扩增产物电泳图Fig.1 PCR productof BSH gene
2.2 重组克隆质粒的鉴定
重组克隆质粒pMD18-BSH的PCR产物和双酶切产物经1%琼脂糖凝胶电泳分析,在接近1 000 bp处可见特异性目的基因条带,见图2。

图2 重组克隆质粒pMD-BSH的PCR鉴定Fig.2 The PCR identity of pMD18-/BSH clone vector
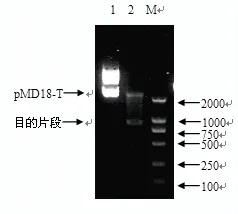
图3 重组克隆质粒pMD-BSH的酶切鉴定M:DL2000:标准分子量;1:pMD18-BSH;2:pMD18-BSH(BamH I/Sal I)Fig.3 Restrictionmap of recombinant plasmid pMD18-BSH
2.3 测序比对结果
使用DNAMAN软件,将测序的核苷酸序列与Genebank已公布的序列(EU477374)进行比对,结果显示两者同源性高达99.3%。由于菌株的特异性和遗传密码子的简并性,所以核苷酸序列有个别差异。见图4。
3 结论
研究采用PCR技术从植物乳杆菌Lp M1-UVS29基因组中得到了BSH基因片段,通过克隆、测序,得到其核苷酸序列,为995 bp。对其核苷酸序列进行生物信息学分析,发现BSH基因的核苷酸序列与Genbank(EU477374)有较高的同源性。同源性高达99.3%,证明成功克隆了BSH结构基因。

图4 测序的核苷酸序列与Genebank(EU477374)比对Fig.4 The comparison between the predicted amino acid sequence and the EU477374
[1]李贵节.降胆固醇乳杆菌的综合评价及胆盐水解酶活性研究[D].上海:上海交通大学,2008.
[2]牛治霞,刘恩梅.益生菌中胆盐水解酶(BSHs)研究进展[J].中国乳品工业,2007,35(9):35-40.
[3]Binder H.J,B.Filburn,M.Floch.Bile acid inhibition of intestinal anaerobic organism[J].Am.J.Clin.Nutr,1975,28(10):119-125.
[4]KIM G B,LEE B H Biochemical and Molecular Insights Into Bile Salt Hydrolase in the Gastrointestinal Microflora a Review Asian-Aust[J].Anita Sci,2005,18:1505-1512.
[5]MOSER SA,SAVAGE D C.Bile Salt Hydrolase Activity and Resistance to To city of Conjugated Bile Salts are Unrelated Properties in Lactobacilli[J].Appl Environ Microbiol,2001,67:3476-3480.
[6]Jean-pierregrill,etal.Purification and characterization of conjugated bile salt hydrolase from bifidobacterium longum BB536[J].Applied and Envi-ronmental Microbiology, 1995,3(7):2577-2582.
[7]Kocubinski G,Perez P,Antoni G.Screening of Bile Resistance and Bile Peicipitation in Lactic Acidacteria and Bifidobacteria[J].Food Prot,1999,5(62):905-912.
[8]王玉文,王延祥,李平兰.胆盐水解酶的分离纯化与部分特性研究[J].2007,5(7):35-41.
[9]PEREIRA D I,GIBSON G R.Effects of Consumption of Probiotics and Prebiotics on Serum Lipid Levels in Humans[J].Crit Rev Biochem Mol Biol,2002,37:259-281.
[10]Binder H.J,B.Filbum,M.Floch.Bile acid inhibition of intestinal anaerobic organism[J].Am.j.Clin.Nutr,1975,28 (10):119-125.
[11]Coleman JP,Hudson LL.Cloning and characterization of a conjugated bile acid hydrolase gene from Clostridium perfringens.Appl[J].Environ.Microbiol,1995,61:2518-2520.
[12]萨姆布鲁克J,拉塞尔DW,黄培堂.分子克隆实验指南[M].3版.北京:科学出版社,2003.
