鲍曼不动杆菌整合酶基因测定及可变区基因盒结构分析*
2011-02-28马全玲武大伟魏殿军张坚磊胡静仪
马全玲 武大伟 魏殿军 张坚磊 胡静仪
整合子(integron)是上世纪80年代在对耐药质粒和转座子进行系统研究的过程中被发现的。常见的耐药整合子分为5类,其中Ⅰ、Ⅱ、Ⅲ类相关研究较多。整合子本身无法进行转移,但常常出现在转座子及接合性质粒上,这种情况提高了耐药基因盒扩散的能力[1]。本研究通过设计Ⅰ、Ⅱ、Ⅲ类整合酶基因的特异性引物,采用PCR和DNA序列分析方法测定本地区鲍曼不动杆菌整合子存在的状况及类型,结合已检测的耐药基因,应用多基因聚类分析的方法对临床分离株进行亲缘性分析,以了解其在天津地区的流行情况。
1 材料与方法
1.1 材料 2008年2月—2009年2月收集分离自天津医科大学第二医院、天津市第一中心医院及天津市环湖医院的多重耐药鲍曼不动杆菌共55株。采用K-B法测定14种抗菌药物的敏感性,判定标准按照美国临床实验室国家标准化委员会(CLSI)2007年的标准。医院内感染诊断标准参照卫生部2001年《医院感染管理规范》诊断标准,对3种及3种以上不同种类抗菌药物耐药的菌株判定为多重耐药株。标准菌株为大肠埃希菌ATCC 25922和铜绿假单胞菌ATCC 27853。
1.2 模板DNA的提取 按文献[2]操作。整合酶基因及可变区引物的合成由上海生工生物工程技术服务有限公司完成,各种靶基因引物序列和目的产物长度,见表1。

表1 整合酶基因及可变区引物序列
1.3 整合酶基因及整合子可变区的PCR扩增 (1)整合酶基因的PCR扩增:对55株多重耐药株分别进行Ⅰ、Ⅱ、Ⅲ类整合酶基因的PCR扩增。Ⅰ类和Ⅱ类、Ⅲ类整合酶基因检测的反应条件相同,参照参考文献[3]。(2)整合酶基因阳性株整合子可变区的限制性片段长度多态性(RFLP)-PCR扩增:反应体系(50 μL)为One shot LA PCRTMMix 25 μL,上下游引物(25 pmol)各1 μL,模板DNA 3 μL,剩余体积用灭菌去离子水补足,94℃5 min,94℃45 s,47℃45 s,72℃1 min,72℃10 min,进行35次循环。对可变区的扩增产物用内切酶HinfⅠ进行RFLP分型。
1.4 观察指标 PCR产物经琼脂糖凝胶电泳后用紫外透射仪进行初步分析,阳性结果用凝胶成像系统拍照。把整合子可变区扩增片段大小相同且RFLP显示同一带型的可变区片段认为具有相同的DNA序列[4]。PCR产物的纯化、克隆均按试剂盒说明操作后,将连有目的基因的重组质粒送往上海英骏公司进行正反向测序,在GenBank上进行BLAST分析后确定可变区含有的基因盒类型。
1.5 多基因聚类分析菌株间亲缘性(Average法) 参照参考文献[5],用1来代表特异性基因扩增的片段条带存在,用0来代表特异性基因扩增的片段条带不存在,把55株多重耐药鲍曼不动杆菌已检测过的各种基因检测数据收集起来组成一个原始数据矩阵,采用SPSS 10.0软件进行聚类分析以获得菌株间亲缘性分析结果。
2 结果
2.1 整合酶基因和可变区的检测 55株多重耐药鲍曼不动杆菌共检出47株含有Ⅰ类整合酶基因,在所入选的药物中,整合酶基因阳性株耐药率明显高于整合酶基因阴性株,见表1。其中47株Ⅰ类整合酶基因阳性株中有23株检出可变区结构,未检出Ⅱ、Ⅲ类整合酶基因,可变区的PCR产物共有2个片段长度:1 500 bp(4株)和2 000 bp(19株),见图1、2。对其用HinfⅠ进行酶切后,发现1 500 bp可变区RFLP显示同一带型;2 000 bp可变区RFLP也显示同一带型,见图3、4。
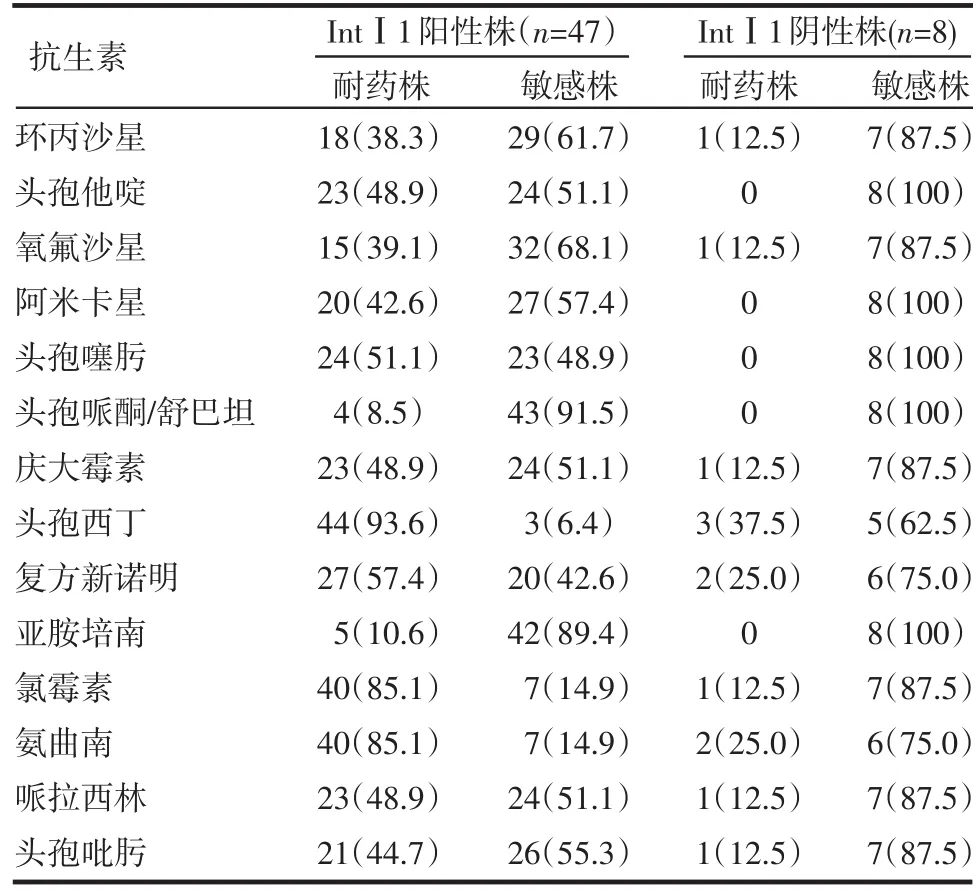
表2 Ⅰ类整合子阳性株与阴性株耐药率比较 株(%)

图1 1 500 bp可变区PCR产物电泳结果

图3 1 500 bp可变区RFLP电泳结果
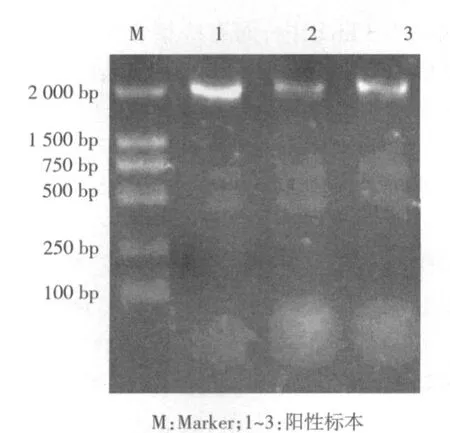
图2 2 000 bp可变区PCR产物电泳结果

图4 2 000 bp可变区RFLP电泳结果
2.2 可变区PCR产物测序 BLSAT网上比对分析结果显示,1 500 bp可变区含有arr-3和aacA4 2个基因盒;2 000 bp可变区含有aacA4、catB8及aadA1 3个基因盒。
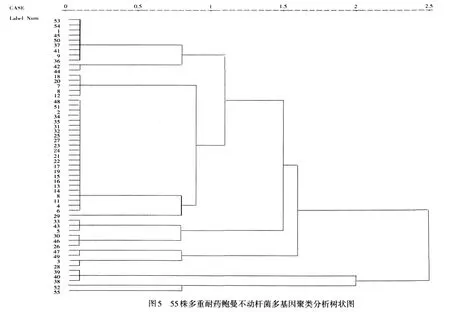
2.3 多基因聚类分析菌株间亲缘性 见图5。55株多重耐药鲍曼不动杆菌中主要存在3种克隆株,Ⅰ:53、54、1、45、50、37、41、9及36号菌株均携带TEM、ADC、intⅠ1基因;Ⅱ:18、20、7、10及12号菌株均携带ADC基因;Ⅲ:48、51、2、34、35、31、32、25、27、23、14、21、22、17、19、15、16、13、14、8、11、4及6号菌株均携带ADC和intⅠ1基因,39、40、38号菌株携带TEM、ADC、intⅠ1、OXA-23及IMP-2基因,30、46、26号菌株携带intⅠ1基因,33、43、5号菌株未发现携带本实验所研究的基因。其中,Ⅰ和Ⅱ组有交叉均含有ADC基因,Ⅰ和Ⅲ也有交叉均含有intⅠ1基因等,它们之间具有亲缘关系。
3 讨论
整合子由1个编码整合酶的intⅠ基因、2个基因重组位点attI和attC、启动子和基因盒构成,目前已知的2组整合子包括:耐药整合子(resistant-integrons, RI)和超级整合子(super-integrons,SI)。耐药整合子位于转座子、质粒及染色体,几乎所有编码抗微生物耐药的耐药基因均来自耐药整合子。根据整合酶基因序列不同,耐药整合子可分为5类,Ⅰ类整合子主要存在于Tn21及与Tn21类似转座子上,Ⅱ类整合子常与Tn7相关,Ⅲ类整合子仅在少数肠杆菌科如沙雷氏菌中可检出。有报道Ⅳ、Ⅴ类整合子在其他菌中存在[1]。本研究结果显示,47株鲍曼不动杆菌Ⅰ类整合酶基因扩增阳性,阳性率为85.5%(47/55),未测到Ⅱ、Ⅲ类整合酶基因,与Hall等[6]报道在不动杆菌中Ⅰ类整合子最常见相符。
整合子可变区基因盒由耐药基因和attC(59 bp)组成,在整合子中已发现100多种基因盒[7],编码的耐药基因覆盖了大多数目前正在使用的抗菌药物。本研究对整合酶基因阳性株进行可变区PCR扩增,结果显示有23株可变区阳性,共检测出2种Ⅰ类整合子系统,片段长度为1 500 bp和2 000 bp。其中4株出现1 500 bp条带,测序结果为arr-3,aacA4;19株出现2 000 bp条带,测序结果为aacA4,catB8,aa⁃dA1,它们分别是氨基糖苷类、氯霉素、利福平类耐药的基因,其中aacA4,catB8,aadA1这一基因盒组合在国内外均有报道[8]。另外,本研究中整合子可变区并未检测出磺胺类耐药基因,但药敏结果却显示整合子与该类抗生素耐药的有关,考虑原因可能是由于整合子阳性菌对磺胺类药物的耐药还有另外的影响因素,即在Ⅰ类整合子的3′保守区含有一个磺胺类耐药基(sul1),故携带Ⅰ类整合子的菌株就可介导对磺胺类抗菌药物的耐药。
本研究中未发现喹诺酮类抗菌药物的耐药基因位于整合子可变区中,但药敏表型上整合子阳性株耐药率比阴性株耐药率显著增高,可能是与细菌染色体DNA的基因突变有关。本研究结果显示,同一类耐药基因aacA4可在不同菌株间检出,表明在药物持续选择压力下,不仅耐药基因盒上耐药基因本身发生变异,也促使整合子整合其他耐药基因,耐药基因盒在不同菌株间移动,造成细菌耐药性的变化,甚至造成多重耐药。此前笔者曾预测整合子上存在OXA-23或IMP-1、IMP-2基因,但结果显示该类基因未处于整合子上,考虑正是这2类基因未能快速大范围扩散的原因。
本研究结果显示,整合子可变区的PCR扩增阳性率(23/55,41.8%)没有整合酶基因PCR扩增阳性率高(47/55,85.5%),有24株未能扩增出可变区结构,与国外相关研究报道相似[9],可能原因是基因盒片段过长不能有效扩增,常规PCR扩增有效的产物大小应在2~3 kb以内,而有些整合子可变区含多个耐药基因盒超过有效扩增范围,且若一株菌内同时存在2个以上大小片段不等的整合子结构,则PCR扩增时可能仅扩出小片段整合子;另外,若整合子3-CS引物相应序列缺失造成无法扩增,也可造成假阴性结果。
本研究结果表明,携带3种基因、携带1种基因及携带2种基因的3种鲍曼不动杆菌为流行株(3个克隆),它们均携带intⅠ1基因,提示intⅠ1可与其他耐药基因同时存在一个流行株中;另外,这些菌株均已被公认为院内感染菌株,亦提示本研究中流行株的出现是由于院内感染所致。同时,每个克隆所含菌株耐药表型不尽相同,且它们分离自不同医院,表明克隆株在医院间存在交叉传播,需要临床医生和微生物工作者给予足够重视。
[1]陶陶,姜海洋,付建平,等.整合子-基因盒系统与细菌耐药性[J].医学综述,2010,16(11):1620-1622.
[2]Khalaf NG,Eletreby MM,Hanson ND.Characterization of CTX-M ESBLs in Enterobacter cloacae,Escherichia coli and Klebsiella pneumoniae clinical isolates from Cairo,Egypt[J].BMC Infectious Diseases,2009,4(9):84-86.
[3]Gaur A,Prakash P,Anupurba S,et al.Possible role of integrase gene polymerase chain reaction as an epidemiological marker:study of multidrug-resistant Acinetobacter baumannii isolated from noso⁃comial infections[J].Int J Antimicrob Agents,2007,29(4):446-450.
[4]Gu B,Tong MQ,Zhao WJ,et al.Prevalence and characterization of classⅠintegron among Pseudomonas aeruginosa and Acinetobacter baumannii isolates from patients in Nanjing,China[J].J Clin Micro⁃biol,2007,45(1):241-243.
[5]凌月明,陈金玉,刘秀华,等.多药耐药铜绿假单胞菌耐药机制与菌株亲缘性分析[J].中华医院感染学杂志,2009,19(23):3168-3171.
[6]Hall RM,Collis CM.Mobile gene cassettes and integrons:capture and spread of genes by site-specific recombination[J].Mol Micro⁃biol,1995,15(4):593-600.
[7]Rowe-Magnus DA.Integrase-directed recovery of functional genes from genomic libraries[J].Nucleic Acids Res,2009,37(17):e118.
[8]Lee YT,Huang LY,Chen TL,et al.Gene cassette arrays,antibiotic susceptibilities,and clinical characteristics of Acinetobacter bau⁃mannii bacteremic strains harboring class 1 Integorns[J].J Microbiol Immunol Infect,2009,42(3):210-219.
[9]Turton JF,Kaufmann ME,Glover J,et al.Detection and Typing of Integorns in epidemic Strains of Acinetobacter b aumannii Found in the United Kingdom[J].J Clin Microbiol,2005,43(7):3074-3082.
