重组真核质粒pERFP-C1-hTudor-SN-SN(1~4)的构建和表达*
2011-02-28朱梦瑜高星杰钱宝鑫王保亚
付 晓 朱梦瑜 高星杰 钱宝鑫 王保亚 赵 虹 葛 林 杨 洁
人类Tudor-SN(tudor staphylococcal nuclease)蛋白,又称为p100蛋白、SND1(staphylococcal nuclease domain containing 1),首次作为EB病毒细胞核抗原2(Epstein-Barr virus nuclear protein 2,EBNA2)的转录调控激活因子被发现[1]。Callebaut等[2]运用疏水簇结构分析(hydrophobic cluster analysis,HCA)发现人类Tudor-SN蛋白由N端4个重复葡萄球菌核酸酶样(staphylococcal nucleases-like,SN-like)的SN(1~4)结构域及C端的Tudor-SN 5(TSN)结构域组成。本研究采用能够表达红色荧光蛋白(red fluorescent protein,RFP)的pERFP-C1质粒载体,构建pER⁃FP-C1-hTudor-SN-SN(1~4)重组质粒,在真核细胞内表达带红色荧光标签的Tudor-SN蛋白SN(1~4)各功能片段,从而为深入研究Tudor-SN蛋白SN结构域的生物学功能提供便利工具。
1 材料与方法
1.1 材料 真核表达质粒pSG5-Tudor-SN-flag、pERFP-C1-Tudor-SN,大肠杆菌DH5α及HeLa细胞均为本实验室保存。限制性内切酶EcoRⅠ、BamHⅠ及T4 DNA连接酶均购自Fer⁃mentas公司,Taq酶购自北京全式金生物技术有限公司,T/A载体(pTZ57R/T)购自大连Takara生物工程公司,B型小量DNA快速回收试剂盒购自北京博大泰克生物基因技术有限公司,质粒快速小提试剂盒购自北京索来宝科技有限公司,去内毒素质粒提取试剂盒购自Omega公司,Lipofectamine 2000购自Invitrogen公司。倒置荧光显微镜购自日本Olym⁃pus公司,显微数码摄影机购自日本Nikon公司,引物由北京六合通公司合成,基因测序工作由北京天一辉远生物科技有限公司完成。
1.2 方法
1.2.1 目的片段的获取 参照质粒快速小提试剂盒内说明书提取pSG5-Tudor-SN-flag质粒DNA。根据Tudor-SN的SN(1~4)结构域序列设计含有EcoRⅠ和BamHⅠ限制性内切酶位点的引物序列,见表1。以pSG5-Tudor-SN-flag质粒DNA为模板,PCR扩增SN(1~4)各片段,条件为95℃预变性5 min,95℃30 s,55℃45 s,72℃3 min共33个循环,72℃延伸10 min。将PCR产物进行琼脂糖凝胶电泳鉴定,紫外灯下切割含有目的条带的凝胶块,利用凝胶回收试剂盒进行目的片段回收,在T4 DNA连接酶作用下,与T/A载体22℃连接过夜。连接产物转化感受态细菌DH5α,在含有氨苄的LB平板上筛选克隆。挑取阳性克隆,摇菌扩增并提取质粒。以EcoRⅠ和BamHⅠ进行双酶切,凝胶回收试剂盒回收并纯化各目的片段。

表1 Tudor-SN的SN(1~4)功能片段引物序列
1.2.2 线性pERFP-C1的获取 采用pERFP-C1质粒载体,见图1。以质粒快速小提试剂盒提取pERFP-C1-Tudor-SN质粒DNA,采用EcoRⅠ、BamHⅠ双酶切pERFP-C1-Tudor-SN质粒,完整切下线性pERFP-C1质粒载体,琼脂糖电泳。凝胶回收试剂盒回收得到约4 700 bp线性pERFP-C1。
1.2.3 重组真核表达质粒pERFP-C1-Tudor-SN-SN(1~4)的构建 在T4 DNA连接酶作用下,将线性pERFP-C1质粒载体与具有相同黏性末端的Tudor-SN各功能片段SN(1~4)进行22℃连接过夜。连接产物转化感受态细菌大肠杆菌DH5α,在含有卡那霉素的LB平板上筛选克隆。挑取阳性克隆,摇菌扩增并提取重组质粒。

图1 pERFP-C1质粒图谱
1.2.4 单、双酶切及基因测序鉴定 用EcoRⅠ和BamHⅠ对重组质粒pERFP-C1-Tudor-SN-SN(1~4)进行单、双酶切,并以1.5%的琼脂糖凝胶电泳验证片段的大小。同时,对重组质粒进行基因测序鉴定。
1.2.5 HeLa细胞的转染及荧光显微镜观察 以去内毒素质粒提取试剂盒提取无内毒素重组质粒。HeLa细胞接种至10 mm培养皿中,置于含10%胎牛血清的DMEM高糖培养基,37℃、5%的CO2培养箱中培养,至细胞80%汇合时,依据产品说明书进行脂质体瞬时转染。转染后48 h,在倒置荧光显微镜下观察pERFP-C1-Tudor-SN-SN(1~4)的表达并以显微数码摄影机拍照。
2 结果
2.1 PCR扩增目的片段 以pSG5-Tudor-SN-flag为模板进行PCR扩增,再以1.5%的琼脂糖凝胶电泳鉴定PCR产物,结果在500 bp左右观察到与目的片段长度相符的荧光条带,见图2。
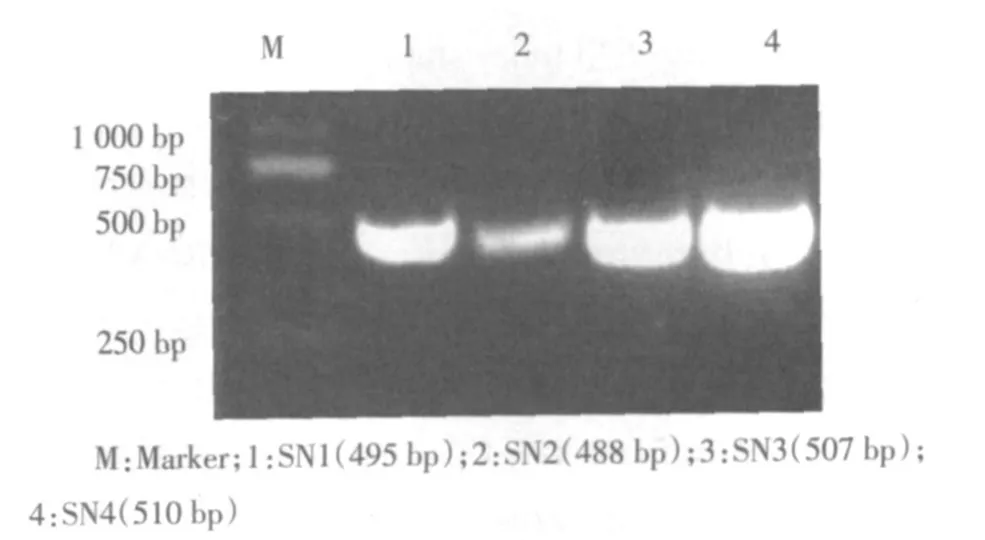
图2 目的片段SN(1~4)的获取
2.2 线性pERFP-C1质粒载体的获取 采用EcoRⅠ、BamHⅠ双酶切pERFP-C1-Tudor-SN质粒,切下完整的线性pERFP-C1质粒载体,经1%琼脂糖凝胶电泳,在4 700 bp左右出现条带,见图3,纯化回收后用于后续的连接反应。
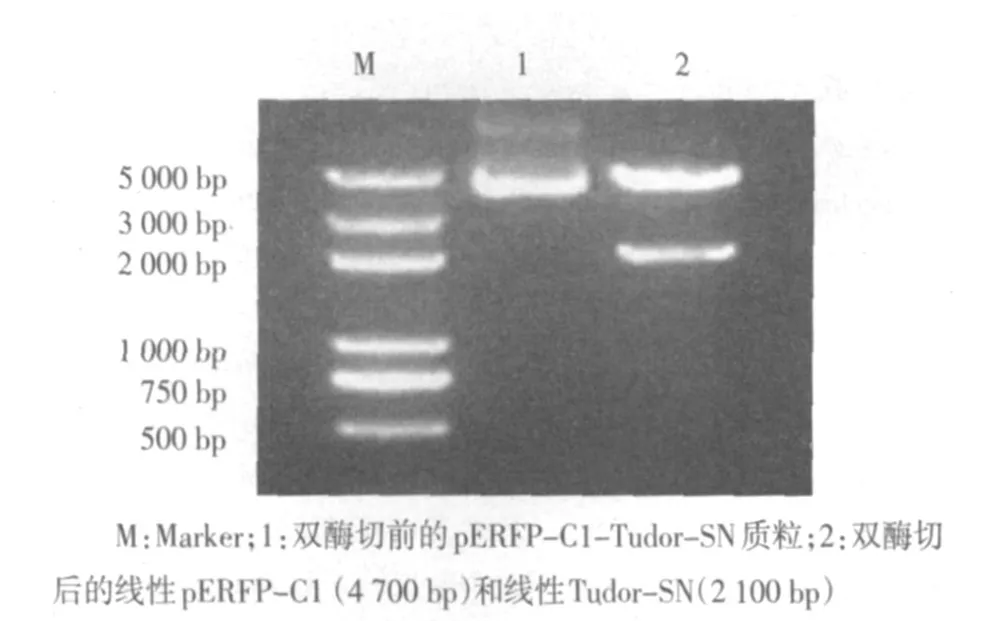
图3 线性pERFP-C1质粒载体的获取
2.3 重组真核表达质粒pERFP-C1-Tudor-SN-SN(1~4)的鉴定
2.3.1 单、双酶切及测序鉴定 将构建的重组质粒分别进行EcoRⅠ和BamHⅠ的单、双酶切,产生的条带大小与预期相符,见图4,重组质粒基因测序结果正确,证明Tudor-SN-SN(1~4)各功能片段已成功插入到pERFP-C1的多克隆位点上。
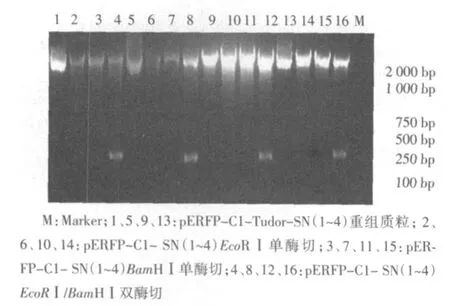
图4 pERFP-C1-Tudor-SN-SN(1~4)的单、双酶切鉴定图
2.3.2 荧光显微镜下观察重组质粒的表达 在10 mm培养皿上接种HeLa细胞,脂质体瞬时转染pER⁃FP-C1-Tudor-SN-SN(1~4),培养48 h后,荧光显微镜下可见红色荧光蛋白RFP-Tudor-SN-SN(1~4)的表达,见图5。
3 讨论
本研究所采用的pERFP-C1是将pEGFP-C1载体中编码绿色荧光蛋白(green fluorescent protein,GFP)的基因替换为樱桃红色荧光蛋白(cher⁃ry-RFP)基因后得到的一种真核质粒载体,其多克隆位点(multiple cloning site,MCS)位于RFP序列和SV40 polyA之间。插入到MCS的目的基因如果与RFP基因的阅读框一致,就可以与RFP的C末端融合表达,且目的蛋白的生物学活性通常不受影响。重组真核红色荧光质粒转染入细胞后并不干扰细胞的生长,还可根据红色荧光信号来定位示踪目的蛋白,故该质粒可用于活细胞状态下目的基因表达的观察、质粒转染效率的鉴定及蛋白间结合关系的研究等。
图5 荧光显微镜下观察重组质粒的表达(×40)
真核基因表达是一个精细复杂的过程,包括基因转录、pre-mRNA剪接、mRNA翻译等步骤。基因转录和pre-mRNA剪接并非2个独立的的过程,常通过某些特定蛋白的作用而紧密相联[3]。人类Tu⁃dor-SN蛋白就是一种可偶联调控转录和剪接过程的多功能蛋白。研究发现,Tudor-SN蛋白作为一种转录共激活因子,借助其N端重复的SN结构域与c-Myb[4],STAT6[5],STAT5[6]等多种启动子特异转录因子结合,在基因的转录调控中发挥重要作用。本研究将Tudor-SN蛋白的SN(1~4)基因序列成功插入到pERFP-C1质粒中,构建针对SN结构域的红色荧光重组质粒,其可与特定转录因子的绿色荧光重组质粒共同转染入真核细胞,通过共聚焦显微镜技术进行共定位分析,将有助于进一步研究Tudor-SN蛋白不同SN片段在基因转录调控的作用。
葡萄球菌核酸酶又称为耐热核酸酶,是一种Ca2+依赖酶,可水解单/双链DNA和RNA的5-磷酸二酯键,生成3-单核苷酸和多核苷酸,其催化能力主要依赖于其氨基酸序列中的2个活化位点[7]。人类Tudor-SN蛋白的SN结构域虽与葡萄球菌核酸酶相似,却未发现此活化位点,推测其可能没有催化DNA的能力[8]。然而,Tudor-SN蛋白SN结构中的第一个亚结构域(98个氨基酸)却含有可识别多种核苷酸的保守OB折叠结构(oligonucleotide/oligosac⁃charide-binding fold,寡聚核苷/寡聚糖结合折叠)[2],提示Tudor-SN蛋白的SN结构域虽没有核酸酶的催化位点,但仍有可能与核酸结合。本研究成功进行了Tudor-SN蛋白不同SN结构域的红色荧光标记,有助于深入探讨该多功能蛋白在基因转录调控、核酸代谢及相关疾病发生中的作用。
[1]TongX,Drapkin R,Yalamanchili R,et al.The Epstein-Barr virus nuclear protein 2 acidic domain forms a complex with a novel cellu⁃lar coactivator that can interact with TFIIE[J].Mol Cell Biol,1995, 15(9):4735-4744.
[2]Callebaut I,Mornon JP.The human EBNA-2 coactivator p100:mul⁃tidomaian organization and relationship to the staphylococcal nucle⁃ase fold and to the tudor protein involved in Drosophila melanogas⁃ter development[J].Biochem J,1997,321(Pt1):125-132.
[3]Kornblihtt AR,de la Mata M,Fededa JP,et al.Multiple links be⁃tween transcription and splicing[J].RNA,2004,10(10):1489-1498. [4]Leverson JD,Koskinen PJ,Orrico FC,et al.Pim-1 kinase and p100 cooperate to enhance c-Myb activity[J].Mol Cell,1998,2(4): 417-425.
[5]Yang J,Aittomaki S,Pesu M,et al.Identification of p100 as a coacti⁃vator for STAT6 that bridges STAT6 with RNA polymeraseⅡ[J].EM⁃BO J,2002,21(18):4950-4958.
[6]Paukku K,Yang J,Silvennoinen O.Tudor and nuclease-like do⁃mains containing protein p100 function as coactivators for signal transducer and activator of transcription 5[J].Mol Endocrinol,2003, 17(9):1805-1814.
[7]HynesTR,Fox RO.The crystal structure of Staphylococcal nuclease refined at 1.7 A resolution[J].Proteins Struct Funct Genet,1991,10 (2):92-105.
[8]Ponting CP.P100,a transcriptional coactivator,is a human homo⁃logue of staphylococcal nuclease[J].Protein Sci,1997,6(2): 459-463.
