SUMO基因的内在原核启动子活性及其在大肠杆菌蛋白表达系统中的应用
2011-02-10亓燕红邹竹荣邹华英范云六张春义
亓燕红,邹竹荣,邹华英,范云六,张春义
1 山东农业大学农学院,泰安 271000
2 云南师范大学生命科学学院,昆明 650092
3 云南省农业科学院 院办档案科,昆明 650231
4 中国农业科学院生物技术研究所,北京 100081
在真核生物泛素家族成员中,SUMO (Small ubiquitin-related modifier,小泛素相关修饰物) 具有多种重要的生物学功能。SUMO化修饰介导靶蛋白分子定位和功能调节,在转录调控、核质转运、信号转导、DNA修复和细胞周期调控等方面均发挥着重要作用[1-2]。
近年来,SUMO作为N端融合标签在大肠杆菌表达系统中得到越来越多的应用[3-8]。SUMO标签不仅能有效提高目标蛋白的表达水平和可溶性[5],而且整体作为一个空间结构上的识别位点,被其特异性蛋白酶 Ulp1从融合表达蛋白上定位 (SUMO末端二甘氨酸,-GG基序的后随肽键上) 切除[3,9],不存在任何非特异性酶切,目标蛋白上可以不留任何残基,这对生产具有天然氨基酸序列的重组蛋白极其适用。另外,Ulp1不仅酶切效率高,而且在较广范围的pH和温度条件下都具有活性[3],这相比其他切除标签、依赖识别序列的工具蛋白酶 (如Factor Xa、烟草蚀刻病毒蛋白酶 TEV、肠激酶 EK) 都具有显著的综合优势。类似的泛素 (Ubiquitin) 融合系统虽然具备 SUMO融合系统的诸多优点[3,10],但切除泛素标签的去泛素化酶DUBs不稳定,难以生产和廉价制备[3];而且大肠杆菌中存在一种内源性泛素蛋白酶 ElaD,特异性切割泛素融合蛋白,使得泛素标签作用失去意义[11]。因此,SUMO融合技术已成为目前大肠杆菌重组蛋白表达和纯化的重要手段。
尽管如此,SUMO融合系统在载体构建效率和蛋白可溶性等方面仍有待改进。在本实验中,我们发现酿酒酵母SUMO基因Smt3(Sm) 具有组成型原核启动子活性。利用此特性,结合引入Sm 3′末端StuⅠ位点以及His标签和超酸增溶标签,构建了基于 Sm’-LacZα融合基因的系列通用克隆表达载体。通过多个基因克隆和表达实验,结果表明在改良的SUMO融合系统中实现了基因表达载体的快速构建和蛋白高可溶性表达,一同建立的共表达载体系统还可以用作研究细胞内蛋白间的相互作用。
1 材料与方法
1.1 材料
大肠杆菌菌株 DH5α、BL21(DE3) 以及pUC19、pET32a(+)、pACYC184和pEGFP-N1等质粒为本实验室保存,本工作中所建载体以外的其他载体均为本实验室先前构建和保存。限制性内切酶、pfu DNA聚合酶、T4 DNA连接酶和 DNA marker购自MBI Fermentas公司;DNA快速纯化回收试剂盒和蛋白分子量marker购自北京鼎国生物技术有限公司;氨苄青霉素 (Amp)、氯霉素 (Cm)、异丙基-β-D-硫代半乳糖苷 (IPTG) 为Genview公司产品;其他生化试剂均为国产分析纯。本工作中所有引物 (表1) 的合成以及DNA序列分析均由上海生工生物工程技术服务有限公司完成。

表1 本研究中所用引物Table 1 Primers used in this study
1.2 方法
1.2.1 载体的构建
所用基因操作方法均参照分子克隆实验手册[12]。载体构建如结果中详述,并通过DNA测序验证。
1.2.2 原核启动子预测分析
在NCBI网站上查询并提取各物种SUMO基因的 mRNA序列,分别对其基因编码区序列 (CDS)通过软莓网站 (www.softberry.com) 提供的原核启动子预测程序 BPROM 和设定的参数 (LDF≥0.2指含有启动子) 进行分析,得到预测原核启动子的强度 (LDF值)、−10和−35元件等信息,并通过EXCEL软件整理分析。
1.2.3 蛋白表达及SDS-PAGE分析
将表达载体转化至大肠杆菌BL21(DE3) 中,挑取单菌落37 ℃培养过夜。然后按照4%量接种到含相应抗生素浓度的LB液体培养基中 (对pET载体单独表达使用100 mg/L Amp,对pAY载体单独表达使用34 mg/L Cm;对pET和pAY载体共表达使用100 mg/L Amp和34 mg/L Cm),37 ℃培养至OD600达0.6,加入IPTG至终浓度1 mmol/L,37 ℃诱导表达4 h。收集10 mL的诱导表达细菌培养液,离心后加入3 mL PBS缓冲液悬浮,并用超声波裂解。取细胞裂解物20 µL,15 000 r/min、4 ℃离心5 min,分为上清 (S) 和沉淀 (P),用12% SDS-PAGE进行电泳检测,并用凝胶成像系统生成图形文件。
1.2.4 蛋白体外酶切反应
将SUMO融合蛋白的pET载体表达细菌与酵母Ulp1蛋白酶的载体pET(Ulp1) 表达细菌分别按上述方法制备得到裂解物上清。两者上清一般按体积比20∶1混匀,放置 25 ℃反应 3 h,然后用 12% SDS-PAGE分析反应情况。
2 结果
2.1 酵母SMUO基因Smt3具有组成型原核启动子活性
利用引物 SmNdFw/SmStRv扩增酿酒酵母SUMO基因 Smt3(Sm) 的 DNA片段平末端克隆到pUC19的HincⅡ(GTCGAC) 位点,转化大肠杆菌DH5α,发现在X-gal+IPTG的LB平板上长出来的菌落几乎全都为蓝色。经PCR鉴定Sm的重组克隆,命名为pUC(Sm-LacZα)。

图1 质粒pUC19(Sm-LacZα) 中SUMO基因Smt3与LacZα融合的序列分析Fig. 1 Sequence analysis of the fusion gene of Smt3 and LacZα in plasmid pUC19 (Sm-LacZα).
通过测序分析,发现Sm插入 (图1,两竖箭头之间) 打断了pUC19上LacZα的翻译阅读框,即由核糖体结合位点 RBS(lac) [AGGA]介导的翻译 (图1)。但是,Sm的反向引物SmStRv在其3′末端 (即 -GG基序密码子处) 引入 StuⅠ位点 (AGGCCT)产生了一个经典的RBS,即RBS(sm) [GGAGG],同时该引物 5′端附加保护碱基对应的互补碱基 (-AT 3′) 与克隆位点HincⅡ的平末端 (5′ GAC-) 融合产生一个ATG密码子,与RBS(Sm) 的间隙长为7 bp,这些刚好为其下游LacZα表达提供了有效的翻译元件 (图 1),从而导致蓝色菌落形成。不过,这种翻译模板除了由Lac启动子 (Plac) (图1) 在IPTG诱导下产生的Sm-LacZα转录产物,会不会在Sm上存在其他启动子产生新的转录产物并由 RBS(sm) 介导翻译LacZα呢?
通过对 Sm(306 bp) 进行启动子预测分析,发现 Sm存在依赖σ70的原核启动子 Psm,包含−35(Psm)、−10(Psm) 元件,分别为 TTAAGA、TATTAGAAT (其后面6 bp为核心序列) (图1),可能引导下游LacZα表达。由于受固有Lac启动子的转录干扰,这种预测需要进一步证实。
随后以 pUC(Sm-LacZα) 为模板,利用引物SmNdFw和 LZXhRv,扩增出 Sm-LacZα片段,经NdeⅠ和 XhoⅠ酶切,与同样酶切的 pET32a(+) 连接,转化大肠杆菌DH5α,发现在X-gal+IPTG的LB平板上也能长出蓝色菌落,而且经鉴定全都为阳性克隆,取名pET(Sm-LacZα)。由于pET32a(+) 使用依赖噬菌体 T7 RNA聚合酶的 T7启动子 (后随LacO序列),DH5α和载体自身不表达T7 RNA聚合酶,因而即便存在 IPTG也不会由 T7启动子转录Sm-LacZα,那么这个导致蓝色菌落形成的LacZα表达唯独可能由Sm启动子Psm的作用所致。
另外,以pEGFP-N1为模板,利用引物GFPfw和GFPrv扩增GFP (绿色荧光蛋白基因) 编码区片段,分别平端插入到 pET(Sm-LacZα) 的 StuⅠ和Ecl136Ⅱ位点 (图1),转化大肠杆菌DH5α,通过蓝白斑筛选和菌落 PCR 鉴定正确阳性克隆pET(Sm-GFP) stu和pET(Sm-GFP) ecl。GFP片段在两个位点的插入都导致其后面的LacZα不表达,形成白色菌落。分别挑取 2种白菌落划线在普通 LB平板上培养,发现pET(Sm-GFP) ecl的菌落逐渐变成绿色,而pET(Sm-GFP) stu的菌落颜色则没什么变化。通过序列分析,GFP在两个位点的插入都与其5′端Sm形成一个正确阅读框的融合基因,而且都有可能由Sm启动子引导GFP转录物产生。但是,在StuⅠ位点插入导致RBS (sm) (图1) 与GFP起始密码子ATG紧密相邻,GFP不能在此处翻译;而在Ecl136 II位点插入则可以通过RBS (sm) 和其后的ATG翻译出GFP蛋白并累积,使pET(Sm-GFP) ecl菌落逐渐变成绿色。这表明在诱导物IPTG不存在的情况下,Sm启动子能够组成型发挥转录活性,叠加在 StuⅠ位点的 RBS(sm) 是其下游基因翻译的重要功能元件。总之,上述结果表明酵母SMUO基因Smt3具有明显的组成型原核启动子活性。
2.2 不同物种SUMO基因的原核启动子预测分析
SMUO是真核生物中广泛存在的一类蛋白,酿酒酵母SUMO基因Smt3具有原核启动子活性,我们推测其他物种的SUMO基因可能具有同样特性。
通过软莓程序BPROM对NCBI核酸数据库中几乎所有的SUMO基因编码区 (CDS) 进行启动子预测分析,发现121个被测对象中约90%都具有原核启动子活性 (LDF≥0.2),在不同范围 LDF值的SUMO CDS的比例依次为3% (>5)、4% (4~5)、9% (3~4)、19% (2~3)、38% (1~2) 和17% (0.2~1) (图2)。进一步将不同SUMO CDS按物种划分成4类:真菌 (Fungi)、植物 (Plant) (绝大多数为被子植物)、无脊椎动物 (Invertebrate) 和脊椎动物(Vertebrate),然后分类统计它们在不同LDF范围所占比例。结果显示,真菌和植物中 85%的 SUMO CDS具有原核启动子活性,而在脊椎和无脊椎动物中这个比例高达95%。LDF>3的SUMO CDS在各类物种所占比例分别为30% (真菌)、18% (植物)、27% (无脊椎动物) 和2% (脊椎动物),说明除脊椎动物外其他物种的 SUMO CDS有较大比例具有较高的原核启动子活性,尤其在真菌和无脊椎动物中表现更为突出,LDF>4的比例分别为19%和16% (图2)。
2.3 基于Sm’-LacZα融合基因的通用克隆表达载体构建及其特性分析
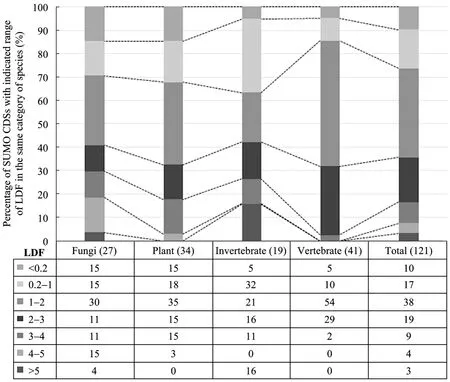
图2 含某指定范围预测启动子LDF值的SUMO基因CDS在同类物种中所占比例Fig. 2 Percentages of SUMO CDSs with indicated range of LDFs of the predicted promoters in the same category of species.
以 pET(Sm-LacZα) 为模板,使用引物对HSNdFw/SmNsRv和LZNsFw/LZXhRv,通过引物重叠延伸法[13]合成新的融合基因片段 Sm’-LacZα。通过NdeⅠ和XhoⅠ酶切,与同样酶切的pET32a(+) 连接,构建成pET(Sm’-LacZα) (图3A)。Sm’是由引物HSNdFw使Sm 5′端加上了一个His标签序列 (H6),有利于表达蛋白纯化;同时在 Sm’-LacZα融合处又引入了NcoⅠ和SalⅠ/HincⅡ位点,使得该处多克隆位点 (MCS) 数目更多,便于目的基因 (如TEV) 多种形式克隆和表达。在Sm 3′末端 -GG基序密码子处,引入了一个StuⅠ位点下划线代表-GG基序密码子,代表Ulp1切点;大写序列 AGGCCT代表 StuⅠ位点,代表平末端切点) (图1、图3A),该酶切平末端5′-Sm’ ggAGG-3′能与任何靶蛋白编码序列 target的 PCR产物 5′-N target-3′ (在target 5′端仅需通过PCR引物添加任何一种碱基N,表1) 连接,即可得到正确阅读框的Sm’-target融合基因 (即5′-Sm’ ggAGGNtarget-3′),融合表达蛋白刚好在-GG基序 (由ggAGGN 编码)后被 Ulp1酶切开,不会残留任何氨基酸在靶蛋白的N端。由于Sm标签还只能相对有限地提高靶蛋白的可溶性[7-8],我们分别将超酸增溶标签 Msb (E. coli msyB)、Yd (E. coli yjgD)、Od (E. coli rpoD的N端结构域)[7-8]的PCR产物用NdeⅠ酶切,然后插入到pET(Sm’-LacZα) 的NdeⅠ位点 (图3A),构建成通用增溶表达载体 pET(MsbSm’-LacZα)、pET(YdSm’-LacZα) 和pET(OdSm’-LacZα),超酸标签Msb、Yd、Od与Sm’分别形成复合超酸增溶标签MsbSm’、YdSm’和OdSm’。由于Sm原核启动子活性,所有这些载体的大肠杆菌 DH5α转化菌落在X-gal+IPTG平板上都呈现蓝色。总之,这些载体组成的改良SUMO融合系统理论上不但通过蓝白斑筛选非常便于目的基因PCR产物的快速克隆和蛋白可溶性表达,而且尤其适合含天然氨基酸序列靶蛋白的重组生产。
此外,用引物PayFw和PayRv扩增低拷贝型质粒 pACYC184[14],得到去除四环素抗性基因的大片段,然后用EcoR V酶切,与上面Sm’-LacZα融合基因 PCR片段连接,转化大肠杆菌 DH5α,通过蓝白斑筛选和菌落 PCR鉴定构建了重组质粒pAY(Sm’-LacZα) (图 3B)。与载体 pET(Sm’-LacZα)一样,它也可以通过蓝白斑筛选用作目的基因片段(如Ulp1) 的快速克隆和表达。由于pAY(Sm’-LacZα)派生载体含pACYC184的复制起始区p15A,与其他类型如pET(Sm’-LacZα) 派生载体 (含 ColEI复制起始区) 相容,两者可在同一细菌中共表达,可以用于两个 (或几个) 表达靶蛋白的相互作用分析。

图3 载体pET(Sm’-LacZα) (A) 和pAY(Sm’-LacZα) (B) 示意图Fig. 3 Diagrams of vectors pET(Sm’-LacZα) (A) and pAY(Sm’-LacZα) (B).
2.4 基于 pET(Sm’-LacZα) 系列的基因表达载体构建及其蛋白表达分析
根据上述 pET(Sm’-LacZα) 系列载体的特性,分别利用引物对 GfpFw/GfpRv、Et1Fw/Et1Rv、TevFw/TevRv、EkFw/EkRv从pET(GFP)、pET(Et1)、 pET(EK)、pET(TEV) 扩增出靶蛋白 GFP、Et1 (TEL-SAM,转录抑制因子TEL的N端SAM结构域)、EK、TEV的基因片段,插入到载体的StuⅠ位点,通过蓝白斑筛选和菌落PCR鉴定,依次得到重组质粒 pET(Sm’-GFP)、pET(MsbSm’-GFP)、 pET(Sm’-Et1)、pET(YdSm’-Et1)、pET(OdSm’-Et1)、pET(Sm’-EK)、pET(MsbSm’-EK)、pET(YdSm’-EK)、pET(OdSm’-EK)、pET(Sm’-TEV)、pET(MsbSm’-TEV)、pET(YdSm’-TEV) 和pET(OdSm’-TEV)。
通过 IPTG诱导,这些载体都获得了很好地表达,融合表达蛋白的可溶性与其对应的非融合表达靶蛋白相比都不同程度地获得了提高。其中,Sm’ 能轻微增强蛋白可溶性,但其各种复合超酸增溶标签(MsbSm’、YdSm’、OdSm’) 的增溶效果则极其显著(图4),这与我们先前工作报道的结论一致[7-8]。
将每个高可溶性表达融合蛋白的细菌裂解物上清分别与表达 Ulp1酶的细菌裂解物上清进行体外反应,经SDS-PAGE分析,发现这些含Sm的融合蛋白都能与Ulp1很好地反应,Sm’及其各种复合标签 (MsbSm’、YdSm’、OdSm’) 都能有效地从融合蛋白上切离,并得到相应的靶蛋白产物 (GFP、Et1、EK、TEV) (图5),这说明Sm的N端附加蛋白序列并不影响 Ulp1对它的特异性酶切,而且也证实Ulp1酶的反应条件非常温和[3,8],在细菌 PBS裂解上清中都有很好的活性。这种SUMO/Ulp1体外反应实验可以用来分析不同标签对靶蛋白的增溶作用机理[8]。另外,这类融合蛋白表达产物如果通过 His标签亲和层析纯化,然后与纯化的 Ulp1酶体外反应,将能够有效地制备各种所需的靶蛋白 (尤其是含天然氨基酸序列的功能蛋白),与我们以前报道的脂联素结果一致[15]。
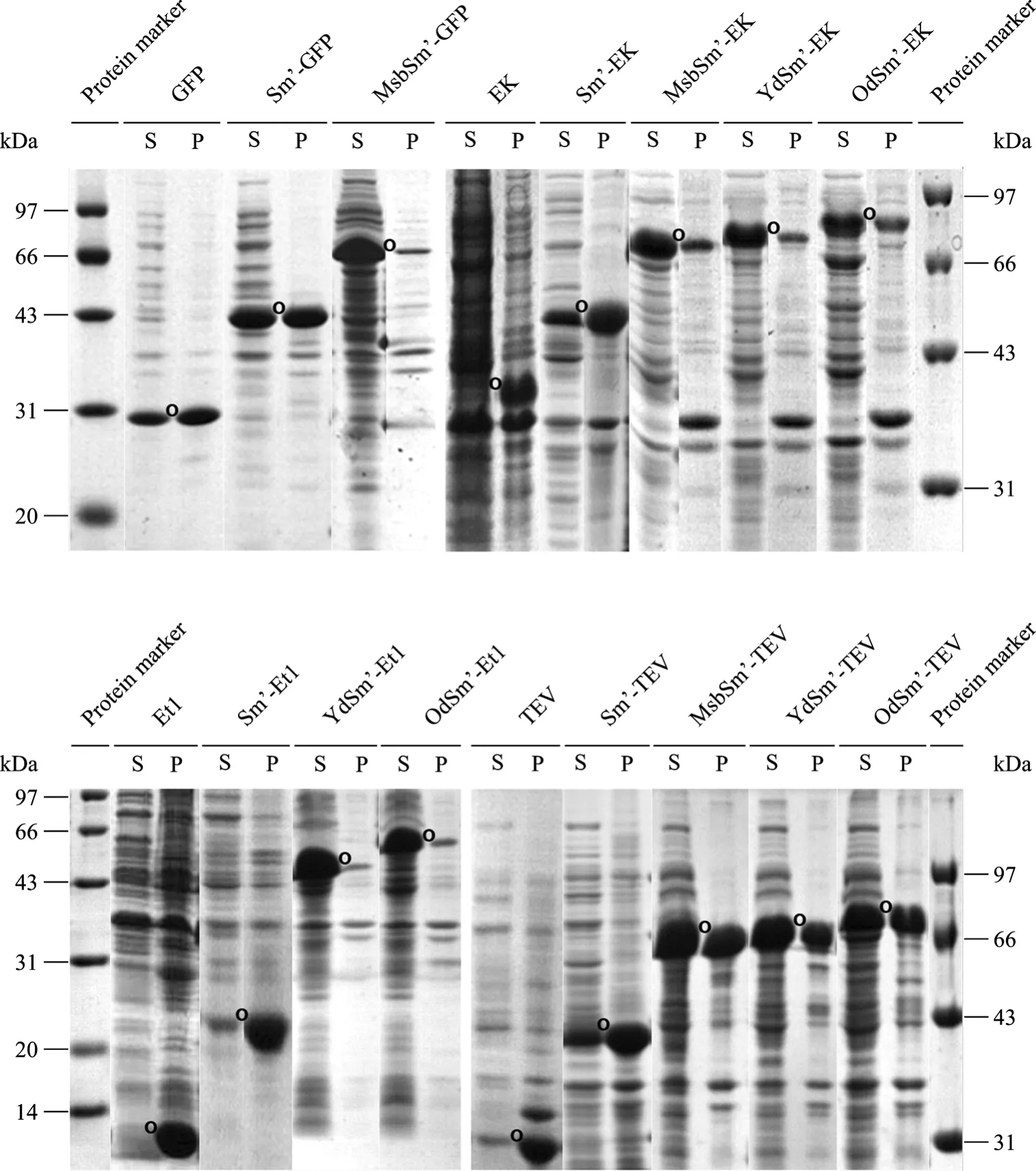
图4 SDS-PAGE分析各种表达载体在大肠杆菌BL21(DE3) 中的表达Fig. 4 Expression of various constructs in E. coli BL21(DE3) analyzed by SDS-PAGE. O indicates the expressed target protein in non-fusion or fusion form; S: supernatant of the cell lysate; P: pellet of the cell lysate.
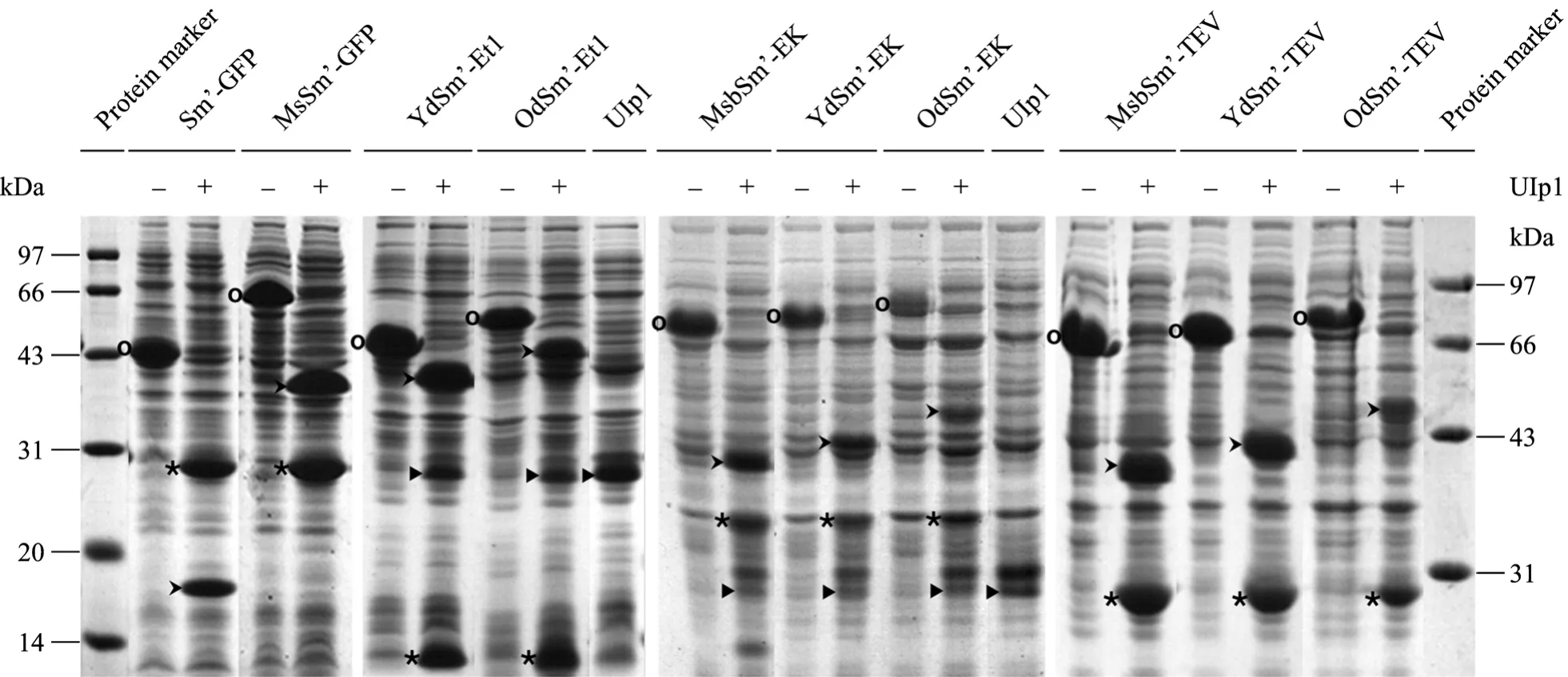
图5 SDS-PAGE分析Sm融合蛋白与Ulp1表达细菌的裂解物上清体外酶切反应Fig. 5 In vitro enzymatic reactions of the supernatants of cell lysates of Sm fusion proteins and Ulp1, analyzed by SDS-PAGE. Oindicates Sm fusion protein; * indicates target protein; > indicates Sm’ or its combinatorial tag (e.g. YdSm’); indicates Ulp1 overused in some reactions.
2.5 基于 pAY(Sm’-LacZα) 的基因表达载体构建及其蛋白表达分析
基于上述 pAY(Sm’-LacZα) 的特性,根据需要可以快速简便地将不同目的基因片段克隆到该质粒的MCS上 (图3B),通过蓝白斑筛选和菌落PCR鉴定得到重组质粒。
当需要诱导型表达时,可以将目的基因表达盒 (诱导型启动子+目的基因+转录终止子) 插入到MCS上的任何单一酶切位点。本实验利用引物对Pt7Fw/Tt7Rv,分别以pET(Et1)、pET(Et1-GFP)、pET(YA)、pET(MST) 为模板,扩增其对应基因表达盒片段Pt7_Et1_Tt7、Pt7_Et1-GFP_Tt7、Pt7_YA_Tt7、Pt7_MST_Tt7 (Pt7为t7启动子,Tt7为t7转录终止子,位于pET载体上,图3A)。将这些PCR片段分别平末端克隆在pAY(Sm’-LacZα) 的MCS内HincⅡ位点 (可以选择其他平末端酶位点如 StuⅠ、SmaⅠ和Ecl136Ⅱ),构建成载体pAY(Et1)、pAY(Et1-GFP)、pAY(YA) 和pAY(MST)。如同pET系列载体一样,它们都是受IPTG诱导表达。在不加IPTG的情况下,Pt7不会发生转录,同时由插入片段上游Sm启动子Psm起始的转录遇到 Pt7后 LacO序列时也会被阻断,因而没有目的基因表达产物;当IPTG诱导时,这些pAY载体都能很好地表达 (图6)。由于pAY载体与 pET载体使用不同的复制起始区 (p15A、ColEI),因而能够相容于同一细菌中共表达。分别将pET(EY)/pAY(Et1)、 pET(EY)/pAY(Et1-GFP)、pET(Trx-Ab)/pAY(YA)、pET(Ab-GFP)/pAY(YA)、pET(TEV)/pAY(MST) 进行共表达,发现IPTG诱导后所有目的基因都不同程度地获得高水平表达 (图6),这种共表达实验可以用来研究两 (几) 个关联蛋白在细胞内的相互作用。此外,所有pAY载体在任何情况下都有一条明显的 Cm抗性基因 (CmR) 表达条带 (图6),这可能与CmR上游组成型启动子活性密切相关。
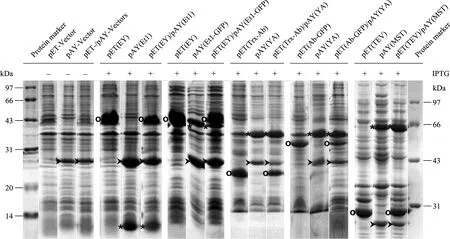
图6 SDS-PAGE分析派生于pAY(Sm’-LacZα) 的pAY系列载体与其相关pET载体的共表达Fig. 6 Co-expressions of pAY (Sm’-LacZα)-derived pAY vectors and their correlated pET vectors, analyzed by SDS-PAGE. O indicates the recombinant protein expressed from pET vector; * indicates the recombinant protein expressed from pAY vector; > indicates the protein product of CmR (chloramphenicol acetyltransferase).
当需要组成型表达时,可以将目的基因编码区插入在pAY(Sm’-LacZα) MCS的NcoⅠ后面任何单一酶切位点 (图3B),并使基因翻译阅读框从NcoⅠ上的 ATG密码子开始,这样目的基因就可以由Sm启动子Psm组成型转录,并由RBS(sm) 和其后NcoⅠ上 ATG密码子介导翻译 (图1)。在一些细菌体内共表达分析系统中,某种蛋白 (如工具酶) 并不需要高水平的表达,这样有必要选择普通启动子而不是强启动子如 Pt7。基于此,我们利用引物对Ulp1Fw/ Ulp1Rv从 pET(Ulp1) 模板中扩增出 Ulp1片段,插入到pAY(Sm’-LacZα) 的HincⅡ位点,构建了 Ulp1组成型表达载体 pAY(c-Ulp1) (c-代表由Sm启动子介导的组成型表达),并与上述含Sm标签的pET系列载体如pET(YdSm’-Et1) 共表达,体内分析标签对靶蛋白的作用,得到的结果 (未显示) 与我们先前报道的一致[8]。
3 讨论
近年来,SUMO融合技术在大肠杆菌表达系统中得到越来越多的应用,但仍处在不断的改进中[3-8]。在本实验中,我们发现酵母SUMO基因Smt3 (Sm)具有组成型原核启动子活性,包含明显的−35、−10特征序列 (图1)。而且,通过软莓BPROM程序预测发现,大多数物种SUMO基因编码区普遍都具有依赖σ70的原核启动子,但活性存在较大差异 (图2),这种现象可能与SUMO基因作为直向同源基因(Ortholog) 在不同类物种的进化程度相关。
利用SUMO标签本身特点及Sm内含原核启动子特性,通过在Sm 3′末端 (-GG基序密码子处) 引入StuⅠ位点和进一步在Sm的5′端附加His标签和超酸增溶标签 (Msb、Yd、Od),我们构建了基于Sm’-LacZα融合基因和 pET32a(+) 背景的系列通用克隆表达载体 (图3A),并由多个基因克隆和表达实验得到检验 (图 4、5)。由此组成的改良 SUMO融合系统集成了诸多特性,如通过蓝白斑筛选快速构建基因表达载体、SUMO和超酸增溶标签促进蛋白可溶性表达、SUMO/Ulp1反应系统制备任何含天然氨基酸序列的靶蛋白、利用His标签纯化表达蛋白、以及pET系统固有的蛋白高水平表达等等,因此可以成为大肠杆菌重组蛋白表达的有力工具;与另一改良的SUMO融合系统[6]相比,在某些方面可能更具优势。
另外,我们还构建了基于Sm’-LacZα融合基因、派生于质粒pACYC184 (低拷贝、含p15A复制起始区) 的通用克隆表达载体pAY(Sm’-LacZα) (图3B)。通过蓝白斑筛选鉴定,由此按需构建的pAY系列目的基因表达载体可以进行单独表达或与其他含不同复制起始区 (如ColEI) 的质粒表达载体如pET系列共表达 (图 6)、组成型或诱导型表达,以用于蛋白研究的不同用途如分析细胞内蛋白间的相互作用关系[8]等。
[1] Hay RT. SUMO: a history of modification. Mol Cell, 2005, 18(1): 1−12.
[2] Johnson ES. Protein modification by SUMO. Annu Rev Biochem, 2004, 73: 355−382.
[3] Malakhov MP, Mattern MR, Malakhova OA, et al. SUMO fusions and SUMO-specific protease for efficient expression and purification of proteins. J Struct Funct Genomics, 2004, 5(1/2): 75−86.
[4] Butt TR, Edavettal SC, Hall JP, et al. SUMO fusion technology for difficult-to-express proteins. Protein Expr Purif, 2005, 43(1): 1−9.
[5] Marblestone JG, Edavettal SC, Lim Y, et al. Comparison of SUMO fusion technology with traditional gene fusion systems: Enhanced expression and solubility with SUMO. Protein Sci, 2006, 15(1): 182−189.
[6] Lee CD, Sun HC, Hu SM, et al. An improved SUMO fusion protein system for effective production of native proteins. Protein Sci, 2008, 17(7): 1241−1248.
[7] Su Y, Zou ZR, Feng SY, et al. The acidity of protein fusion partners predominantly determines the efficacy to improve the solubility of the target proteins expressed in Escherichia coli. J Biotech, 2007, 129(3): 373−382.
[8] Zou ZR, Cao LJ, Zhou P, et al. Hyper-acidic protein fusion partners improve solubility and assist correct folding of recombinant proteins expressed in Escherichia coli. J Biotech, 2008, 135(4): 333−339.
[9] Mossessova E, Lima CD. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Mol Cell, 2000, 5(5): 865−876.
[10] Catanzariti AM, Soboleva TA, Jans DA, et al. An efficient system for high-level expression and easy purification of authentic recombinant proteins. Protein Sci, 2004, 13(5): 1331−1339.
[11] Catic A, Misaghi S, Korbel GA, et al. ElaD, a deubiquitinating protease expressed by E. coli. PLoS ONE, 2007, 2(4): e381.
[12] Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual. 3rd Ed. New York: Cold Spring Harbor Laboratory Press, 2001.
[13] Horton RM, Hunt HD, Ho SN, et al. Engineering hybrid genes without the use of restriction enzymes: gene splicing by overlap extension. Gene, 1989, 77(1): 61−68.
[14] Rose RE. The nucleotide sequence of pACYC184. Nucleic Acids Res, 1988, 16(1): 355.
[15] Zhou P, Feng SY, Zou ZR, et al. Soluble expression of human adiponectin in Escherichia coli. J Sichuan Univ: Nat Sci Ed, 2010, 47(1): 161−166.周培, 封淑颖, 邹竹荣, 等. 人脂联素在大肠杆菌中的可溶性表达. 四川大学学报:自然科学版, 2010, 47(1): 161−166.
