海藻工具酶——褐藻胶裂解酶研究进展
2011-02-10李丽妍管华诗江晓路郝剑君
李丽妍,管华诗,江晓路,郝剑君
1 中国海洋大学医药学院,青岛 266003
2 中国海洋大学食品科学与工程学院,青岛 266003
海藻工具酶
——褐藻胶裂解酶研究进展
李丽妍1,管华诗1,江晓路2,郝剑君2
1 中国海洋大学医药学院,青岛 266003
2 中国海洋大学食品科学与工程学院,青岛 266003
从海洋生物中筛选提取有价值的酶类,开发海洋多糖降解产物,已成为海洋生物资源开发的一个重要方面。因此,近年来对于海藻工具酶之一的褐藻胶裂解酶及其降解产物——褐藻寡糖的研究日益受到人们的普遍关注。从褐藻胶裂解酶的来源、分类、底物专一性、作用方式及结构与机理研究、酶活力测定和酶学性质等方面,结合本课题组的研究工作综述近十年来有关褐藻胶裂解酶的研究进展。
褐藻胶,褐藻胶裂解酶,聚甘露糖醛酸,聚古洛糖醛酸,寡糖
1 褐藻胶裂解酶的分类
褐藻胶裂解酶按其降解褐藻多糖作用方式的不同在EC (Enzyme classification) 数据库中被分为多聚α-L-1,4-古洛糖醛酸裂解酶 (EC 4.2.2.11) 和多聚β-D-1,4-甘露糖醛酸裂解酶 (EC 4.2.2.3)。它们分别以β消除方式作用于含有古洛糖和甘露糖的 C-O糖苷键,形成在非还原端 C4,5间具有不饱和双键的4-deoxy-L-erythro-hex-4-enopyranosyluronic acid结构,该寡糖在230~240 nm下有强烈的吸收峰[2]。
为了更加详细地描述酶的分类地位,CAZy (Carbohydrate-active enzymes) 数据库将EC4.2.2的酶根据其初级结构不同细化为 22个多糖裂解酶(Polysaccharide lyase,PL) 家族,按照酶的氨基酸序列相似性加以分类,以反映各个酶的结构特征。褐藻胶裂解酶被划分在PL-5、PL-7和PL-15家族中,其中大多数的褐藻胶裂解酶被列于 PL-5和PL-7中。PL-5属α/α barrel结构,目前共有40个酶;PL-7属β-jelly roll结构,共84个酶,其中82个酶来自细菌,2个酶来自真核生物;个别的褐藻胶裂解酶被列于 PL-15家族,例如来自鞘氨醇单胞菌Sphingomonas sp. A1的alginate lyase A1-Ⅳ和重组酶A1-Ⅳ’,而alginate lyase A1-Ⅰ、A1-Ⅱ属于PL-7家族,A1-Ⅲ属于PL-5家族[3]。大多数PL-5和PL-7家族的褐藻胶裂解酶分别降解 pM (Polymannuronic acid) 和 pG (Polyguluronic acid) 片段,而对于PL-15,则两种情况均存在[4]。
按其分子量大小不同也可将褐藻胶裂解酶分为3类[3]:第1类分子量在20~35 kDa之间(Mr 30 000类),第2类分子量约为40 kDa (Mr 40 000类),第3类分子量约60 kDa (Mr 60 000类)。
2 褐藻胶裂解酶的来源
褐藻胶裂解酶主要来源于海洋藻类、海洋软体动物、棘皮动物等体内和多种微生物 (包括海洋细菌、陆生真菌和极少数的病毒),其中细菌类是褐藻胶裂解酶研究最为广泛的来源,如假单胞菌(Pseudomonas sp. QD03[5]、Pseudomonas sp. F6[6]、Pseudomonas fluorescens[7]、Pseudomonas aeruginose CF1/M1[8]、Pseudomonas sp. Os-ALG-9[9]、Pseudomonas syringae[10])、弧菌 Vibrio sp. O2[11]、固氮菌Azotobacter vinelandii[12]、克雷伯氏菌 Klebsiella pneumoniae[13]、黄杆菌Flavobacterium sp. LXA[14]、棒杆菌 Corynebacterium sp. ALY-1[15]、芽胞杆菌Bacillus sp. ATB-1015[16]、类单胞菌Alteromonas sp. No. 272[17]、交替假单胞菌 (Pseudoaltermonas elyakovii[18],Pseudoalteromonas sp. CY24[19])、肠杆菌 Enterobacter-Cloacae M-1[20]、鞘氨醇单胞菌Sphingomonas sp. strain A1[3]、链霉菌 (Streptomyces sp. A5[21],Streptomyces sp. ALG-5[22]) 和Agarivorans sp. JAM-Alm[23]等。
在海洋软体动物和棘皮动物体内也存在褐藻胶裂解酶。2009年,Hata等[24]对分离自一些腹足类软体动物肝胰腺的褐藻胶裂解酶的基本性质进行了比较研究,分离自古腹足目Archeogastropoda的皱纹盘鲍 Haliotis discus hannai、纽西兰鲍螺 H. iris和粗瘤黑钟螺Omphalius rusticus的单一酶的分子量分别为28、34、34 kDa,而分离自中腹足目的滨螺L. brevicula的3个酶分子量分别为35、32、 28 kDa。这些酶依据其底物特异性被确认为polyM裂解酶 (EC 4.2.2.3)。Suda等[25]1999年在小球藻病毒 Chlorella virvus中也发现了褐藻胶裂解酶的基因。
3 底物专一性
褐藻胶是由α-L-古洛糖醛酸和它的异构体β-D-甘露糖醛酸以1,4糖苷键连接成的线性多糖,包括3种可能的排列方式:多聚古洛糖醛酸 (poly-α 1,4-L-guluronate,pG)、多聚甘露糖醛酸 (poly-β1,4-D-mannuronate,pM) 和不规则杂合片段pMG。褐藻胶的结构组成、褐藻胶裂解酶的作用方式和作用位点见图1。依据褐藻胶裂解酶在褐藻胶pM或pG处的断裂方式不同分为pM裂解酶和pG裂解酶。褐藻胶裂合酶底物专一性包括pG、pM和pMG。褐藻胶的来源通常影响它们的特异性。来自软体动物的褐藻胶裂合酶和来自几种海洋细菌的细胞结合的褐藻胶裂合酶具有 pM底物特异性,而那些发现于细菌培养液的胞外酶则具有pG底物特异性。底物特异性与裂解酶产生菌的来源环境有关,或者可以追溯到褐藻胶的种类,例如能降解细菌来源的褐藻胶多为pM特异性。大多数的裂解酶均具有内切酶活性,但有一些酶是外切酶,从多糖的末端切除单体或二聚体寡糖,如来自Sphingomonas sp. A1的A1-IV[4]。
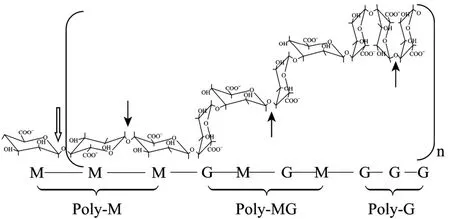
图1 褐藻胶裂解酶酶切位点及作用方式示意图Fig. 1 Profile of degradation position and mode of action of alginate lyase on alginate acid. The place hollow arrow showed the degradation position of exo-alginate lyase. The places solid arrows showed the degradation positions of endo-alginate lyase on pM, pMG and pG, respectively.
4 褐藻胶裂解酶作用方式及机理
褐藻胶裂解酶以β-消除方式催化降解褐藻胶,在C4,5之间形成不饱和双键,并产生一个非还原末端 为 4-deoxy-L-erythro-hex-4-enopyranosyluronic acid的结构。Gacesa[26]假设了褐藻胶裂解酶的催化机理为3步反应,包括:(a) 由盐桥中和以移除羧基离子上的负电荷;(b) 吸引 C5位上的质子;(c) 电子从羧基基团上转移并在C4,5之间形成双键,导致4-O-糖苷键的β消除反应。
在研究众多的褐藻胶裂解酶中,有关其降解机制研究最为全面和深入的是鞘氨醇单胞菌Sphingomonas sp. A1产褐藻胶裂解酶。1993年,Yonemoto等[27]首次报道了菌株Sphingomonas sp. A1产来自同一前体蛋白经翻译后自动加工的 3种褐藻胶裂解酶A1-Ⅰ(66 kDa)、A1-Ⅱ(25 kDa) 和A1-Ⅲ(40 kDa)。1998年Hashimoto等[28]确定A1-Ⅲ为内切酶,对 pM和杂合的褐藻胶片段区域有降解能力,作用于四糖,产生二糖和三糖,并于次年得到该酶的晶体结构[29]。A1-Ⅱ具有pG特异性,并释放三糖和四糖,A1-Ⅰ具有pM和pG特异性,产生二糖和三糖产物。2003年 Miyake等[30]在研究褐藻胶裂解酶降解褐藻胶机理的过程中发现了产自该菌的寡糖裂解酶 (A1-Ⅳ) (85 kDa) 及其基因,该酶在前3种酶将褐藻胶降解为二糖和三糖片段后,以外切形式将寡糖彻底切割为单糖,因此在褐藻胶的彻底降解过程中扮演了重要的角色。该酶基因位于编码前 3种内切酶的前体蛋白基因和ABC载体基因的下游,编码A1-Ⅰ、A1-Ⅱ和A1-Ⅲ共同前体蛋白的基因,编码 A1-Ⅳ酶的基因及载体基因形成了基因簇。为了确定酶催化反应的关键氨基酸,2001年Yoon等[31]对 A1-Ⅲ酶的 His残基进行定点突变研究,证实His192确为活性中心的关键氨基酸之一。2005年A1-Ⅳ与其同源蛋白 A1-Ⅳ’ (90 kDa) 均被划分在PL15家族中[32]。同年,Yamasaki等[33]得到了A1-Ⅱ和重组酶A1-Ⅱ’的晶体结构。2008年,Ogura等[34]报道了A1-Ⅱ’经定点突变和晶体结构分析等推断的该酶的催化中心,确定了His191、Tyr284和Gln189为活性中心关键氨基酸并推断它们在催化中的作用方式。该研究小组主要对该菌所产褐藻胶裂解酶的结构和降解过程进行了研究,通过酶或酶与底物复合物的晶体结构的研究理解褐藻胶裂解酶的作用方式。迄今为止已获得褐藻胶裂解酶A1-Ⅱ、A1-Ⅱ’、A1-Ⅲ和 A1-Ⅳ’的十几种单体和复合物的晶体结构并保存在Protein Data Bank (PDB) 中。
5 褐藻胶裂解酶酶活的测定方法
目前,褐藻胶裂解酶的酶活力测定方法主要有:硫代巴士酸法 (Thiobarbituric acid assay)[35]、紫外吸收法 (Ultroviolet absorption assay)[35]、粘度法(Viscometry assay)[36]和还原糖法 (Reducing sugar assay)[35,37]。硫代巴比妥酸法 (Thiobarbituric acid assay,TBA) 是基于酶解产生的不饱和糖醛酸可以和高碘酸反应生成 β-甲酰丙酮酸,此中间产物再与硫代巴比妥酸反应的阳性产物可以在 548 nm下比色定量。紫外吸收法是基于酶解产生的不饱和糖醛酸在230~240 nm处有较高的吸收峰,这种方法比较灵敏,是目前褐藻胶裂解酶酶活测定的主要方法。粘度法的应用是由于褐藻酸、琼胶等多糖的胶体性质,通过粘度计测定其流体的运动性能的变化判断酶解程度,但这种方法可重复性差,操作繁琐。常用的还原糖法包括DNS法和Nelson-Somogyi法。DNS试剂 (3,5-二硝基水杨酸) 与含有自由醛基和酮基的还原糖共热反应,生成3-氨基-5-硝基水杨酸,在520 nm波长处有最大光吸收,利用比色法测定其还原糖的生成量就可测定酶的活力。而 Nelson-Somogyi法是指酶解产生的还原性寡糖在碱性溶液中将铜试剂中的二价铜离子还原成一价铜离子,中间产物氧化亚铜可与砷钼酸试剂生成蓝色的砷钼蓝溶液,该溶液在一定波长下的吸光值与还原糖的浓度成正相关,从而确定酶的活力。
6 褐藻胶裂解酶的酶学性质及酶系
近年来多种细菌来源的褐藻胶裂解酶[3,5,6,8-10,13,17-18,20,22-24,36,38-47]得到提纯并对其酶学性质进行了研究,现按照分子量、最适温度、最适pH值、等电点和底物特异性等列于表1中。由表可知,按照分子量划分褐藻胶裂解酶的方法,第1类酶多表现为pG裂解特异性,第3类酶则表现出pM裂解特异性或具有多种底物专一性。
本研究小组从海藻中筛选到一株褐藻胶降解菌,经生理生化和分子水平鉴定,该菌株被命名为荧光假单胞菌Pseudomonas fluorescens HZJ216。分离纯化得到 3种褐藻胶裂解酶,其部分酶学性质列于表1中。金属离子Na+、K+、Mg2+等均对3种酶的活力有促进作用,Fe2+、Fe3+、Ba2+和Zn2+等离子则显示不同程度的抑制作用。经优化后发酵液酶活可达1 120 U/mg (紫外吸收法),是制备褐藻胶裂解酶制剂比较理想的候选酶。
职业技术竞赛是体现学生能力的重要途径,也是体现教学效果的重要方式。环境类专业学生拟参加全国职业技术大赛,须进行第一轮的理论考试、第二轮的实操考核、第三轮的小组选拔和第四轮的心理测试,最终选定2名参赛选手进行重点培育和2名备选选手跟踪培养,参赛选手的选拔过程见图1。这种选拔方式虽然能够体现强者更强的用人思路,但却忽略了教育的普惠性[6]。为此,在设置专业实训上,以参赛学生带动和指导未参赛学生的形式,在3周的《环境监测与治理综合实训》课程时间内把平台操作与计算推广到全年级学生,从而惠及全年级学生,让所有学生公平地享受教育资源。这既是教育的初衷,也是“以赛带教、以赛促教”的重要体现。
2008 年 Ma等[18]报道了交替假单胞菌Pseudoalteromonas elyakovii产褐藻胶裂解酶在大肠杆菌中的表达,该重组酶的部分酶学性质见表1,该酶在工程菌大肠杆菌中的表达水平高于 P. elyakovii 39.6倍,粗酶液酶活力为310 U/mg (还原糖法测定)。经Sephadex G-75和DEAE-FF纯化后的酶活力为1 009 U/mg。来自Pseudoalteromonas sp. CY24[19]的褐藻胶裂解酶在pH 10.6范围内表现稳定的活性,0.1 mol/L NaCl可使酶表现最高酶活。Na+、K+、Ca2+和Mg2+可以提高酶活水平。以上两种产自交替假单胞菌属的褐藻胶裂解酶均具有广泛的底物特异性。
链霉菌 Streptomyces sp. A5[21]产褐藻胶裂解酶经硫酸铵分级沉淀、DEAE-cellulose色谱和Sephadex G-100色谱纯化,得到比活力为101.6 U/µg的纯化酶,可被0.05 mol/L SDS和2 mmol/L Hg2+、Cu2+和Fe3+抑制,但EDTA却能提高该酶的活力水平。该酶制备的褐藻胶寡糖片段具有促进香蕉苗根部生长的作用。Kim等[22]2009年报道了海洋细菌Streptomycessp. ALG-5的重组褐藻胶裂解酶,经Ni-Sepharose亲和色谱纯化得到该重组酶,并对其降解产物的二糖、三糖、四糖和五糖纯化及质谱分析。这 2种产自链霉菌属的酶具有相同的pG降解专一性。
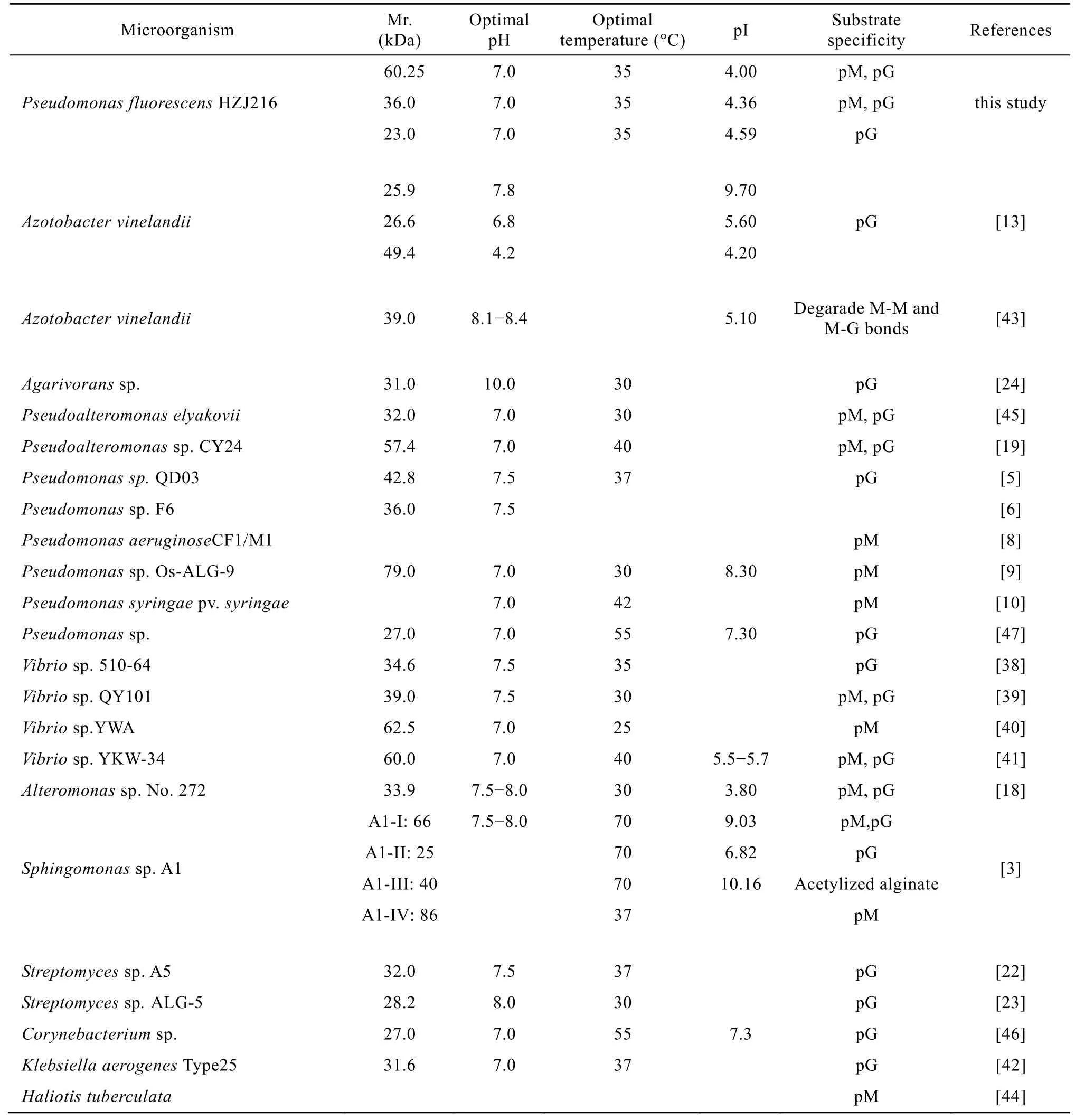
表1 不同来源的褐藻胶裂解酶的部分酶学性质Table 1 Characterization of alginate lyases from different microorganisms
Song等[39]2003年报道了弧菌Vibrio sp. QY101产褐藻胶裂解酶,经阴离子交换色谱和凝胶色谱分离纯化得到39 kDa的单一酶,该酶在0~30 ℃、pH 6.5~8.5范围内酶活稳定,0.5 mol/L NaCl、1.0 mmol/L Ca2+或者5.0 mmol/L Mn2+可以促进酶活的增加,而5.0 mmol/L Ni2+、1.0 mmol/L Fe2+或1.0 mmol/L EDTA抑制酶活。底物特异性研究初步表明该酶作用于pM和pG底物,具有广泛的底物特异性,但是没有有关酶活力的报道。2006年该研究小组[5]报道了来自Pseudomonas sp. QD03编码褐藻胶裂解酶的基因 algL在 pET24a(+)/E. coli BL21中表达一种新的高活性降解细菌褐藻胶的裂解酶,基本酶学性质见表1。AlgL以褐藻酸钠为底物时酶活为188.5 U/mg,来自铜绿假单胞菌P. aeruginosa FRD1的乙酰化褐藻胶为底物时酶活为158.3 U/mg,M 片段为底物时为 197.9 U/mg。而来自 Vibrio sp. QY101的AlyVI却表现出pG特异性,对pM及乙酰化的褐藻胶没有作用。此外,P. aeruginosa 产M特异性裂解酶对乙酰化程度达到 10%的褐藻胶没有活性,而Pseudomonas sp. QD03产AlgL对54%乙酰化的褐藻胶显示高活性,这一活性与对 M片段的活性相当,文章提示该酶可作为治疗铜绿假单胞菌感染的囊中纤维化病的一种辅助治疗药物。该研究小组在 2009 年[19]又报道了海洋细菌Pseudoalteromonas sp. CY24中褐藻胶裂解酶编码基因 alyPI在大肠杆菌中的表达,AlyPI经 Ni-Sepharose亲和色谱纯化,确定其分子量为60 kDa,基本酶学性质见表1。Na+、K+、Mn2+、Ca2+和Fe3+等离子的存在可以促进酶活。AlyPI酶表现为pM、pG和褐藻胶降解能力,具有广泛的底物特异性。
2006−2007年,分离自弧菌Vibrio sp. 510-64[38]、Vibrio sp. YWA[40]和Vibrio sp. YKW-34[41]的褐藻胶裂解酶相继被报道,部分酶学性质见表1。
Vibrio sp. 510-64产褐藻胶裂解酶发酵液酶活力可达41 EU/mg (还原糖法),纯化后酶活力为1 666.67 U/mg (紫外法)。研究表明,50 mmol/L的Na+、Mg2+、Ca2+、Li+和 K+可提高该酶的酶活。Vibrio sp. YWA 产褐藻胶裂解酶的酶活力较低,仅为5.6 U/mg。Vibrio sp. YKW-34产褐藻胶裂解酶具有Na+-K+依赖性,该酶的发酵液酶活为148 U/mg,纯化酶酶活力达3 731 U/mg。
迄今为止,大约有20个来自细菌的编码褐藻胶裂解酶的基因被克隆和测序。来自Sphingomonas sp. A1[4,27]的 A1-I 至 A1-IV、 Pseudomonas sp. OS-ALG-9[9]、丁香假单胞菌 Pseudomonas syringae pv. syringae[10]、Pseudoalteromonas elyakovi[18]、Streptomyces sp. ALG-5[22]和P. elyakovii IAM[23]等的酶基因均表达于大肠杆菌细胞中。其中 A1-IV在大肠杆菌中的表达量为8.6 U/L (培养液),是原始菌株表达水平的 270倍。A1-I在大肠杆菌中的表达量为3 500 U/L,是在Sphingomonas sp. A1中的10倍。2007年发现P. elyakovii IAM产褐藻胶裂解酶在大肠杆菌中的表达量可因钙离子的调节而提高[45]。
7 褐藻胶裂解酶的应用及前景
褐藻胶裂解酶因其降解褐藻胶获得具有生物活性的寡糖而备受人们的青睐,来自P. elyakovii的褐藻胶裂解就已成功应用于对于特殊食品产品有特殊作用的褐藻寡糖的生产[45]。近些年除了对其降解产物的活性研究以外,其应用研究也包括作为海藻解壁酶获得原生质体、辅助提取海藻DNA、降解海藻废物并再利用[46-48]等。
目前已有各种来源的 50多种褐藻胶裂解酶被分离并得到纯化。有关褐藻胶裂解酶早期的工作主要描述其特性并通过酶的特异性应用于褐藻胶多糖结构分析研究。近年来的研究重点转向酶的构效关系研究和降解产物的生物学研究。同时分子生物学技术的发展使得多种褐藻胶裂解酶基因得到克隆、测序并在工程菌中过量表达,这些研究工作都将为实现褐藻胶裂解酶向商业酶转化并大量应用于工业中奠定重要的研究基础,而大量的褐藻胶裂解酶或酶与底物复合物的晶体结构的获得将为人们不断深入认识褐藻胶裂解酶的作用机制并利用其特性在药物分子设计的进一步应用中奠定基础。
[1] Kaiser P, Leborgne L, Hardisso C, et al. An exocellular alginate-lyase isolated from Clostridium alginolyticum cultures. C R Acad Sci D, 1968, 267 (3): 376−379.
[2] Wong TY, Preston LA, Schiller NL. Alginate lyase: review of major sources and enzyme characteristics, structurefunction analysis, biological roles, and applications. Annu Rev Microbiol, 2000, 54: 289−340.
[3] Miyake O, Ochiai A, Hashimoto W, et al. Origin and diversity of alginate lyases of families PL-5 and-7 in Sphingomonas sp. strain A1. J Bacteriol, 2004, 186 (9): 2891−2896.
[4] Miyake O, Hashimoto W, Murata K. An exotype alginate lyase in Sphingomonas sp. A: overexpression in Escherichia coli, purification, and characterization of alginate lyse IV (A1-IV). Protein Expr Purif, 2003, 29(1): 33−41.
[5] Xiao L, Han F, Yang Z, et al. A novel alginate lyase with high activity on acetylated alginate of Pseudomonas aeruginosa FRD1 from Pseudomonas sp. QD03. World J Microbiol Biotechnol, 2006, 22: 81−88.
[6] Miyazaki M, Obata J, Iwamoto Y, et al. Calcium-sensitive extracellular poly (α-L-guluronate) lyase from a marine bacterium Pseudomonas sp. strain F6: purification and some properties. Fisheries Sci, 2001, 67(5): 956−964.
[7] Bakkevig K, Sletta H, Gimmestad M, et al. Role of the Pseudomonas fluorescens alginate lyase (AlgL) in clearing the periplasm of alginates not exported to the extracellular environment. J Bacteriol, 2005, 187(24): 8375−8384.
[8] Rehm BHA. Alginate lyase from Pseudomonas aeruginosa CF1/M1 prefers the hexameric oligomannuronate as substrate. FEMS Microbiol Lett, 1998, 165(1): 175−180.
[9] Kraiwattanapong J, Motomura K, Ooi T, et al. Characterization of alginate lyase (ALYII) from Pseudomonas sp. OS-ALG-9 expressed in recombinant Escherichia coli. World J Microbiol Biotechnol, 1999, 15(1): 105−109.
[10] Preston LA, Wong TY, Bender CL, et al. Characterization of alginate lyase from Pseudomonas syringae pv. syringae. J Bacteriol, 2000, 182(21): 6268−6271.
[11] Kawamoto H, Horibe A, Miki Y, et al. Cloning and sequencing analysis of alginate lyase genes from the marine bacterium Vibrio sp. O2. Mar Biotechnol, 2006, 8(5): 481−490.
[12] Gimmestad M, Ertesvag H, Heggeset TMB, et al. Characterization of three new Azotobacter vinelandii alginate lyases, one of which is involved in cyst germination. J Bacteriol, 2009, 191(15): 4845−4853.
[13] Østgaard K, Knutsena SH, Dyrset N, et al. M. Production and characterization of guluronate lyase from Klebsiella pneumoniae for applications in seaweed biotechnology. Enzyme Microb Technol, 1993, 15(9): 756−763.
[14] An QD, Zhang GL, Wu HT, et al. Alginate-deriving oligosaccharide production by alginase from newly isolated Flavobacterium sp. LXA and its potential application in protection against pathogens. J Appl Microbiol, 2009, 106(1): 161−170.
[15] Matsubara Y, Kawada R, Iwasaki K, et al. Cloning and sequence analysis of a gene (aly PG) encoding poly (alpha-L-guluronate) lyase from Corynebacterium sp. strain ALY-1. J Biosci Bioeng, 2000, 89(2): 199−202.
[16] Nakagawa A, Ozaki T, Chubachi K, et al. An effective method for isolating alginate lyase-producing Bacillus sp. ATB-1015 strain and purification and characterization of the lyase. J Appl Microbiol, 1998, 84(3): 328−335.
[17] Iwamoto Y, Araki R, Iriyama KI, et al. Purification and characterization of bifunctional alginate lyase from Alteromonas sp. No. 272 and its action on saturated oligomeric substrates. Biosci Biotechnol Biochem, 2001, 65(1): 133−142.
[18] Ma LY, Chi ZM, Li J, et al. Overexpression of alginate lyase of Pseudoaltetmonas elyakovii in Escherichia coli, purification, and characterization of the recombinant alginate lyase. World J Microbiol Biotechnol, 2008, 24(1): 89−96.
[19] Duan GF, Han F, Yu WG. Cloning, sequence analysis, and expression of gene alyPI encoding an alginate lyase from marine bacterium Pseudoalteromonas sp. CY24. Can J Microbiol, 2009, 55: 1113−1118.
[20] Shimokawa T, Yoshida S, Kusakabe I, et al. Some properties and action mode of (1→4)-α-L-guluronan lyase from Enterobacter cloacae M-1. Carbohydr Res, 1997, 304(2), 125−132.
[21] Cao LX, Xie LJ, Xue XL, et al. Purification and characterization of alginate lyase from Streptomyces species strain A5 isolated from banana rhizosphere. J Agric Food Chem, 2007, 55(13), 5113−5117.
[22] Kim DE, Lee EY, Kim HS. Cloning and characterization of alginate lyase from a marine bacterium Streptomyces sp. ALG-5. Mar Biotechnol, 2009, 11(1): 10−16.
[23] Kobayashi T, Uchimura K, Miyazaki M, et al. A new high-alkaline alginate lyase from a deep-sea bacterium Agarivorans sp.. Extremophiles, 2009, 13(1): 121−129.
[24] Hata M, Kumagai Y, Rahman MM, et al. Comparative study on general properties of alginate lyases from some marine gastropod mollusks. Fisheries Sci, 2009, 75(3): 755−763.
[25] Suda K, Tanji Y, Hori K, et al. Evidence for a novel Chlorella virus-enconded alginate lyase. FEMS Microbiol Lett, 1999, 180(1): 45−53.
[26] Gacesa P. Enzymic degradation of alginates. Int J Biochem, 1992, 24: 545−552.
[27] Yonemoto Y, Tanaka H, Hisano T, et al. Bacterial alginate lyase gene: nucleotide sequence and molecular route for generation o alginate lyase species. J Ferment Bioeng, 1993, 75(5): 336−342.
[28] Hashimoto W, Okamoto M, Hisano T, et al. Sphingomonas sp. A1 lyase active on both poly-β-D-mannuronate and heteropolymeric regions in alginate. J Ferment Bioeng, 1998, 86(2): 236−238.
[29] Yoon H J, Mikami B, Hashimoto W, et al. Crystal structure of alginate lyase A1-III from Sphingomonas species A1 at 1.78 Å resolution. J Mol Biol, 1999, 290(2): 505−514.
[30] Miyake O, Hashimoto W, Murata K. An exotype alginate lyase in Sphingomonas sp. A1: overexpression in Escherichia coli, purification, and characterization of alginate lyase IV (A1-IV). Protein Expr Purif, 2003, 29(1): 33−41.
[31] Yoon HJ, Choi YJ, Miyake O, et al. Effect of His192 mutation on the activity of alginate lyase A1-III from Sphingomonas species A1. J Microbiol Biotechnol, 2001, 11(1): 118−123.
[32] Hashimoto W, Miyake O, Ochiai A, et al. Molecular identification of Sphingomonas sp. A1 alginate lyase (A1-IV') as a member of novel polysaccharide lyase family 15 and implications in alginate lyase evolution. J Biosci Bioeng, 2005, 99(1): 48-54.
[33] Yamasaki M, Ogura K, Moriwaki S, et al. Crystallization and preliminary X-ray analysis of alginate lyases A1-II and A1-II ' from Sphingomonas sp. A1. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2005, 61(Pt 3): 288-290.
[34] Ogura K, Yamasaki M, Mikami B, et al. Substrate recognition by family 7 alginate lyase from Sphingomonas sp. A1. J Mol Biol, 2008, 380(2): 373−385.
[35] Preiss J, Ashwell G. Alginic acid metabolism in bacteria. J Biol Chem, 1962, 237(2): 309−316.
[36] Sawabe T, Ohtsuka M, Ezura Y. Novel alginate lyases from marine bacterium Alteromonas sp. strain H-4. Carbohydr Res, 1997, 304(1): 69−76.
[37] Nelson N. A photometric adaptation of the somogyi method for the determination of glucose. J Biol Chem 1944, 153: 375−380.
[38] Hu XK, Jiang XL, Hwang HM. Purification and characterization of an alginate lyase from marine bacterium Vibrio sp. Mutant strain 510-64. Curr Microbiol, 2006, 53(2): 135−140.
[39] Song K, Yu WG, Han F, et al. Purification and characterization of aginate lyase from marine bacterium Vibrio sp. QY101. Acta Biochem Biophys Sin, 2003, 35(5): 473−477.
[40] Wang YH, Yu GL, Wang XM, et al. Purification and characterization of alginate lyase from marine Vibrio sp. YWA. Acta Biochem Biophys Sin, 2006, 38(9): 633−638.
[41] Fu XT, Lin H, Kim SM. Purification and characterization of a Na+/K+dependent alginate lyase from turban shell gut Vibrio sp. YKW-34. Enzym Microb Technol, 2007, 41(6/7): 828−834.
[42] Lange B, Wingender J, Winkler UK. Isolation and characterization of an alginate lyase from Klebsiella aerogenes. Arch Microbiol, 1989, 152(3): 302−308.
[43] Ertesvag H, Erlien F, Skjak-Braek G, et al. Biochemical properties and substrate specificities of a recombinantly produced Azotobacter vinelandii alginate lyase. J Bacteriol, 1998, 180(15): 3779−3784.
[44] Heyraud A, Colin-Morel P, Girond S, et al. HPLC analysis of saturated or unsaturated oligoguluronates and oligomannuronates. Application to the determination of the action pattern of Haliotis tuberculata alginate lyase. Carbohydr Res, 1996, 291(23): 115−126.
[45] Sawabe T, Takahashi H, Saeki H, et al. Enhanced expression of active recombinant alginate lyase AlyPEEC cloned from a marine bacterium Pseudomonas elyakovii in Escherichia coli by calcium compounds. Enzyme Microb Technol, 2007(2), 40: 285−291.
[46] Matsubara Y, Kawada R, Iwasaki KI, et al. Extracellular poly (alpha-L-guluronate) lyase from Corynebacterium sp.: purification, characteristics and conformational properties. J Protein Chem, 1998, 17(1): 29−36.
[47] Davidson IW, Sutherland IW, Lawson CJ. Purification and properties of an alginate lyase form a marine bacterium. Biochem J, 1976, 159: 707−713.
[48] Tang JC, Taniguchi H, Chu H, et al. Isolation and characterization of alginate-degrading bacteria for disposal of seaweed wastes. Lett Appl Microbiol, 2009, 48(1): 38−43.
Advances in algae toolenzymes: alginate lyases
Liyan Li1, Huashi Guan1, Xiaolu Jiang1,2, and Jianjun Hao2
1 College of Medicine and Pharmaceutics, Ocean University of China, Qingdao 266003, China
2 College of Food Science and Engineering, Ocean University of China, Qingdao 266003, China
Marine can be considered as a rather unexplored source of biological material. Production of algal oligosaccharides by using valuable enzymes from marine origin has become an important way to utilize marine resources. As one of algal tool enzymes, the use of alginate lyases has been focused mainly on development and application of alginate oligosaccharides with bioactive function in recent years. In this paper, we reviewed the research of alginate lyases over the past decade in several aspects, including their origin, diversity, substrate specification, mode of action, structure and catalysis mechanism, assay of enzyme activity, enzyme characterization, as well as our own experience on this subject. At the end of the review, the application prospects of alginate lyases are presented.
alginate, alginate lyase, mannuronic acid, guluronic acid, oligosaccharide
海洋是一个巨大的资源宝库,又是一个潜力巨大的天然药源。PPS (藻酸双脂钠)、海立特等海洋药物的问世无疑极大地鼓舞了科学工作者们向海洋要宝的信心。褐藻胶作为源自褐藻植物细胞壁的水溶性酸性多糖,在食品、医药和化工等领域具有广泛的应用价值,而褐藻胶降解产物因其具有特殊的化学特性和生物活性,近年来成为一种新的潜在的功能产品不断受到人们的关注,其功能评价和开发研究得到不断深入,活性及药用价值研究已经成为新的热点。褐藻寡糖的酶法降解制备方法因其诸多优势代替化学降解已经成为一种必然。因此,作为一种海藻工具酶,褐藻胶裂解酶的研究对于推动褐藻寡糖的进一步开发有着深远的意义和应用价值。自1968年Kaiser等[1]首次报道分离自梭菌Clostridium alginolyticum的胞外褐藻胶裂解酶以来,对褐藻胶裂解酶的研究取得了长足的发展。本文从褐藻胶裂解酶的分类和来源、酶的性质、底物作用方式、酶活力及结构和机理等方面,结合我们的研究工作综述近十年来褐藻胶裂解酶的研究进展。
November 19, 2010; Accepted: January 26, 2011
Supported by: National Natural Science Foundation of China (No. 30901122).
Xiaolu Jiang. Tel: +86-532-82032290; Fax: +86-532-82032389; E-mail: jiangxl@ouc.edu.cn
国家自然科学基金 (No. 30901122) 资助。
