不依赖油水界面激活的黑曲霉脂肪酶突变体的构建
2011-02-10陈的舒正玉薛龙吟林瑞凤吴继光蒋咏梅李欣林跃鑫黄建忠
陈的,舒正玉,薛龙吟,林瑞凤,吴继光,蒋咏梅,李欣,林跃鑫,2,黄建忠
1 福建师范大学工业微生物教育部工程研究中心 福建省现代发酵技术工程研究中心 福建师范大学生命科学学院,福州 350108
2 宁德师范学院,宁德 352100
脂肪酶 (Lipase,EC3.1.1.3) 又称为甘油三酰酯水解酶,能有效地催化三酰基甘油酯水解为甘油和游离的脂肪酸。脂肪酶活性中心的催化部位通常是由天冬氨酸或谷氨酸、丝氨酸和组氨酸组成的催化三联体[1]。通常情况下,大多数脂肪酶的活性中心都被一种称为“盖子”的结构所覆盖,阻止了底物与活性中心的直接结合。盖子结构具有多样性,有的仅由1个单一的α-螺旋构成,有的由2个α-螺旋构成,还有的仅由1个Loop环构成,少数脂肪酶无盖子结构[2-3]。在油-水界面,脂肪酶盖子结构域构象发生改变,暴露出脂肪酶的活性中心,底物进入活性中心,脂肪酶的催化活性被激活。脂肪酶的催化活性在油水界面大幅度提高的现象称为界面激活[4-5]。受界面激活催化特性的影响,底物的团聚状态一定程度上影响脂肪酶的催化效率。为了获得不依赖界面激活的脂肪酶突变体,科研人员设计和构建了一系列“无盖型”或“开盖型”脂肪酶突变体。
根据已解析的枯草芽胞杆菌Bacillus subtilis脂肪酶A和南极假丝酵母Candida antarctica脂肪酶B的 3D结构,结合其催化特性[6-7],可以推测:具有“盖子”结构域和界面激活特性并不是鉴别脂肪酶和酯酶的恰当标准[8],脂肪酶在没有盖子结构域的情况下依然可以表现出催化活性。Miled等利用基因工程手段缺失人胃脂肪酶 (Human pancreatic lipase,HPL) 的“盖子”结构域后获得的脂肪酶突变体,其催化活性较野生型人胃脂肪酶显著降低[9]。此外,盖子结构域还与脂肪酶的其他酶学性质相关,如对映体选择性、链长特异性和热稳定性等[10-12]。
正因为脂肪酶盖子结构域对于维持其正常功能非常重要,因此利用基因工程手段构建“开盖型”脂肪酶突变体是十分必要的。Carrièr等利用人胃脂肪酶的盖子结构域置换猪胰脂肪酶 (Guinea pig pancreatic lipase related protein 2,GPLRP2) 对应结构域后获得的猪胰脂肪酶突变体,其盖子结构具有永久开盖型构型,同时该突变体不再依赖油水界面激活[13]。这一结果表明,脂肪酶具有的界面激活特性不仅与盖子结构域的存在有关,其他结构因子(如:盖子结构两侧铰链区的氨基酸残基等) 对于稳定盖子构型的开或关有着重要的作用。Brzozowski等报道疏棉状嗜热丝孢菌Thermomyces lanuginosa脂肪酶中位于盖子结构域第一个铰链区的 Arg84对于触发脂肪酶界面活性具有重要的作用[14]。类似的实验结果在米黑根毛霉Rhizomucor miehei脂肪酶中也得到了证实:位于该脂肪酶盖子结构域第一个铰链区的Ser84对触发该脂肪酶界面活性也具有重要的作用[15]。
微生物脂肪酶和酯酶均属于 α/β水解酶折叠家族,均能催化酯键的断裂,二者的三级结构具有高度的相似性,但仅脂肪酶表现出界面激活的催化特性。在先前的工作中,我们对比了黑曲霉脂肪酶(Aspergillus niger lipase,ANL) 和黑曲霉阿魏酸酯酶一级结构和3D结构的异同。在一级结构上,二者的氨基酸残基序列相似性达36%;在3D结构上,二者的盖子结构域存在显著差异。在活性中心的上方,黑曲霉脂肪酶和黑曲霉阿魏酸酯酶均可形成一段α-螺旋,但黑曲霉阿魏酸酯酶该α-螺旋的位置较黑曲霉脂肪酶对应结构的位置而言,更远离活性中心。我们预测这一结构差异可能与二者的催化特性差异之间存在一定的关联性。进一步分析该 α-螺旋两侧铰链区的氨基酸残基序列,我们预测位于ANL盖子结构域第一铰链区的 Ser84和第二铰链区的Asp99对于盖子结构域的开或闭构型可能发挥了决定性的作用[16]。本实验分别用黑曲霉阿魏酸酯酶多肽链中与黑曲霉脂肪酶的Ser84和Asp99相对应的氨基酸残基置换Ser84和Asp99,构建2个黑曲霉脂肪酶突变体:ANL-Ser84Gly和ANL-Asp99Pro,并测定了这2个突变体的部分酶学性质。
1 材料与方法
1.1 试剂与材料
1.1.1 菌株和质粒
克隆宿主 Escherichia coli DH5α,表达宿主Pichia pastoris GS115及质粒pPIC9K-lipanl均为本实验室保存 (lipanl为黑曲霉脂肪酶成熟肽的编码基因序列)[16]。
1.1.2 酶和试剂
高保真Pfu DNA聚合酶购自Sangon生工生物工程 (上海) 有限公司;各种限制性内切酶和 T4 DNA连接酶均购自 TaKaRa宝生物工程 (大连) 有限公司;系列4-硝基苯羧酸酯均购自Sigma公司;甲醇和甘油酯为市售分析纯。
1.2 方法
1.2.1 黑曲霉脂肪酶基因引入突变位点
通过重叠延伸聚合酶链式反应对黑曲霉脂肪酶基因进行定点突变,引入突变位点。基于毕赤酵母密码子偏爱性设计的用于 DNA定点突变的引物见表1。重叠延伸聚合酶链式反应中使用的PCR引物、模板、PCR扩增条件及产物大小见表2。用EcoRⅠ和 NotⅠ酶切 PCR扩增获得的引入突变位点后的lipanl全长基因,然后将其连接到经同样酶切后的pPIC9K上。重组 pPIC9K-lipanl-S84G和 pPIC9K-lipanl-D99P转化E. coli DH5α,DNA测序检验引入突变位点的正确性。

表1 本实验中使用的PCR系列引物Table 1 PCR primers used in this study
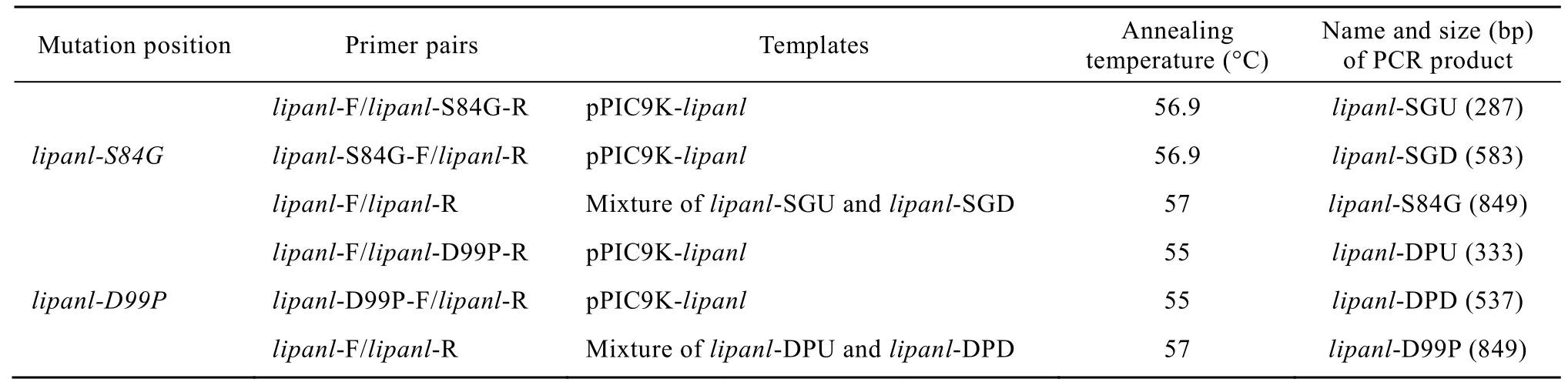
表2 重叠延伸PCR反应中使用的引物、模板、PCR扩增条件及产物大小Table 2 Primer pairs, templates and PCR programs used for lipanl mutagenesis and the resulting PCR products
1.2.2 P. pastoris GS115的转化、脂肪酶基因的诱导表达和脂肪酶的纯化
P. pastoris GS115的转化、脂肪酶基因的诱导表达和脂肪酶的纯化均参照文献[16]。重组脂肪酶经Endoglucosidase H处理、Sephadex G-75凝胶柱纯化及冻干。重组黑曲霉脂肪酶蛋白质浓度的测定参照Bradford法[17]。
1.2.3 脂肪酶活性的测定
脂肪酶活性的平板定性检测方法参照Hiol等的平板检测方法[18],本实验以三丁酸甘油酯作为脂肪酶活性的定性检测底物。
脂肪酶活性的分光光度计检测法参照Kordel等的检测方法[19],使用的缓冲液为0.05 mol/L His-HCl (pH 6.5),45 ℃进行测定。在该反应条件下,每1 min从对应4-硝基苯羧酸酯上释放出1 µmol对硝基苯酚作为1个脂肪酶活力单位 (U)。
为检测黑曲霉脂肪酶及其突变体界面激活效应,参照 Martinelle等所报道的方法[20],测定黑曲霉脂肪酶及其突变体水解不同浓度的 4-硝基苯丁酸酯的催化活力曲线。水解温度为25 ℃,使用的缓冲液为0.05 mol/L His-HCl (pH 6.5)。
1.2.4 温度对黑曲霉脂肪酶及其突变体稳定性的影响
55 ℃下温育黑曲霉脂肪酶及其突变体,在0~60 min的不同时间间隔内,分别检测残余脂肪酶酶活。将初始脂肪酶酶活定义为100%。
2 结果
2.1 脂肪酶编码基因的定点突变
微生物脂肪酶和酯酶具有相似的 α/β水解酶折叠结构,活性中心的催化位点均由 Ser-His-Asp/Glu三联体构成[21]。尽管脂肪酶和酯酶均能催化三酰甘油酯水解为脂肪酸和甘油,或是此反应的逆向合成,但只有脂肪酶具有独特的界面激活特性,而酯酶不具有此特性。在先前的研究中,我们对比了黑曲霉脂肪酶和黑曲霉阿魏酸酯酶的3D结构,并且预测黑曲霉脂肪酶盖子结构域两侧的铰链区影响脂肪酶盖子的构型。进一步分析黑曲霉脂肪酶盖子结构域两侧的氨基酸残基序列和黑曲霉阿魏酸酯酶对应的氨基酸残基序列,筛选出黑曲霉脂肪酶盖子结构域两侧的2个氨基酸残基:Ser84和Asp99,对脂肪酶盖子的构型可能具有决定性的作用[16]。为了验证该预测,将黑曲霉脂肪酶中的Ser84和Asp99分别用黑曲霉阿魏酸酯酶中对应的Gly和Pro残基置换,构建黑曲霉脂肪酶突变体ANL-S84G和ANL-D99P。
利用表1中的引物对表2中的PCR反应体系及反应条件进行PCR扩增。PCR扩增产物经限制性内切酶酶切后插入表达质粒 pPIC9K。重组质粒pPIC9K-lipanl-S84G和 pPIC9K-lipanl-D99P经测序验证阅读框的正确性及突变位点氨基酸残基的正确引入。突变氨基酸残基引入位点对应的碱基序列见图 1,测序结果表明在突变位点已成功引入置换的氨基酸残基。
2.2 黑曲霉脂肪酶及其突变体的诱导表达及纯化
将 pPIC9K-lipanl、 pPIC9K-lipanl-S84G 和pPIC9K-lipanl-D99P三个基因,分别导入到P. Pastoris GS115表达菌株中进行诱导表达,获得的重组脂肪酶具有相同的相对分子量,大约为35 kDa (图2A)。将20 µL重组脂肪酶用三丁酸甘油酯平板定性检测,在ANL、ANL-S84G和ANL-D99点样区周围,形成了清晰的水解圈 (图2B)。
2.3 黑曲霉脂肪酶及其突变体的底物特异性及热稳定性检测

图1 lipanl, lipanl-S84G和lipanl-D99P的序列比对Fig. 1 Sequence alignment of lipanl, lipanl-S84G and lipanl-D99P.
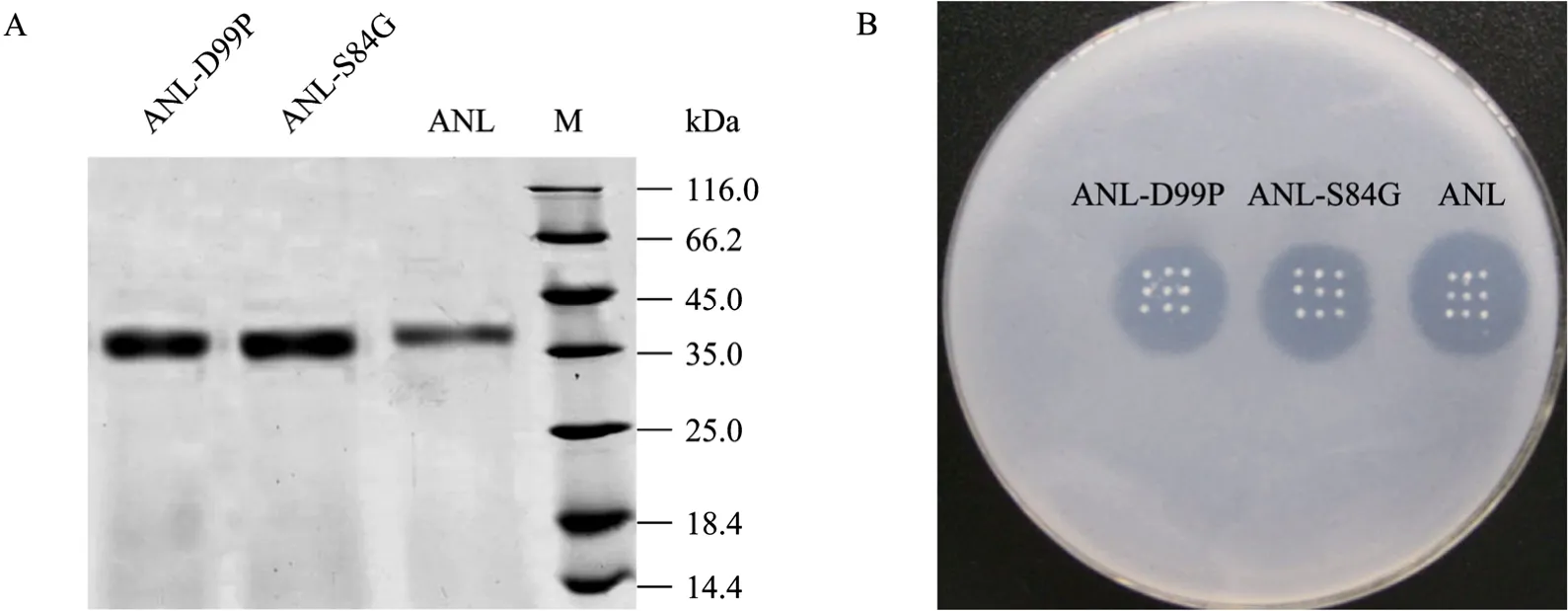
图2 黑曲霉脂肪酶及其突变体的纯化和平板定性检测Fig. 2 Purification and qualitative activity detection of A. niger lipase and A. niger lipase mutants. (A) SDS-PAGE analysis of the purified A. niger lipase and A. niger lipase mutants, M: protein markers. (B) Qualitative activity detection of A. niger lipase and A. niger lipase mutants on tributyrin-agar plate.

表3 黑曲霉脂肪酶及其突变体对系列4-硝基苯羧酸酯的水解活性Table 3 Specific activity of A. niger lipase and A. niger lipase mutants towards series of p-nitrophenyl esters
ANL-S84G 和ANL-D99P表现出了和ANL明显不同的底物特异性和热稳定性。水解系列 4-硝基苯羧酸酯实验结果表明:较ANL而言,ANL-S84G的比活力显著降低,降低幅度从29.8%到76.5%不等;而ANL-D99P水解4-硝基苯棕榈酸酯的比活力上升了2.2倍 (表3)。ANL-S84G的比活力显著降低可能是由于该突变导致重组脂肪酶 ANL-S84G形成了相对稳定的闭盖构型。Brzozowski等认为T. lanuginosa脂肪酶在闭盖构型状态下,Arg84与 Asp57可形成氢键。在油水界面,Arg84与结构重排后的 Cys268之间形成新的氢键,这一变化诱发了脂肪酶盖子结构的开启,并激活了脂肪酶的活性[14]。根据该假说可以推测,黑曲霉脂肪酶的 Ser84与T. lanuginosa脂肪酶上的Arg84在脂肪酶分子中所处的位置相同,因此,在诱导脂肪酶构型转变过程中,发挥了类似的功能。黑曲霉脂肪酶上的Ser84被Gly置换后,这一过程破坏了氢键的形成并将导致ANL-S84G突变体形成相对稳定的闭盖构型,在油水界面,ANL-S84G能形成有效激活构型分子的比率将降低,ANL-S84G的比活力也因此降低。较ANL而言,ANL-D99P催化4-硝基苯棕榈酸酯的比活力提高了2.2倍,而对4-硝基苯月桂酸酯和4-硝基苯肉豆蔻酸酯的比活力基本保持不变 (表 3)。黑曲霉脂肪酶上位于盖子结构域第二铰链区的Asp99被Pro置换后形成的脂肪酶突变体,扩大了盖子结构的伸展空间,使其活性中心能容纳更大的底物分子,因此,对长链脂肪酸酯的比活力有所提高。
上述分析,理论上解释了 S84G 和D99P的突变,增加了ANL-S84G与ANL-D99P盖子结构域及其两侧铰链区的柔韧性,导致其催化活性的降低或提高。此外,S84G和D99P的突变,可能会破坏突变体ANL-S84G与ANL-D99P的二级结构作用力,使突变体分子的二级结构域更趋不稳定,从而导致了其热稳定性的显著降低 (图3)。55 ℃下温育15 min,ANL-S84G和 ANL-D99P分别失去了 85.1%和94.0%的初始酶性,而 ANL在相同情况下仅失去15.6%的初始酶活。S84G和D99P的突变,对脂肪酶活性和热稳定性影响的分子机制还有待进一步深入研究。

图3 黑曲霉脂肪酶及其突变体的热稳定性检测Fig. 3 Effect of temperature on the stability of A. niger lipase and A. niger lipase mutants.
2.4 4-苯硝基丁酸酯浓度对于黑曲霉脂肪酶和黑曲霉脂肪酶突变体的影响
4-硝基苯丁酸酯 (p-nitrophenyl butyrate,pNPB)在水溶液中是部分可溶的,因此常用其作为研究脂肪酶界面激活的底物。为了将 pNPB的自身水解控制在最小程度内,本实验中使用的缓冲溶液为His-HCl (pH 6.5)。实验结果表明,ANL和ANL-S84G表现出了明显的界面激活效应 (图 4A和 4B),而ANL-D99P没有表现出界面激活效应 (图4C)。尽管ANL-S84G表现出界面激活效应,但其水解的动力学曲线和ANL相比,存在明显不同。正如上文所提到的,按照Brzozowski假说,ANL-S84G突变体的盖子结构应该保持相对稳定的“闭盖”构型,但实验结果依然具有界面激活特性。因此,除了 Arg84机制,ANL-S84G应该还存在其他机制触发盖子结构的开盖。D99P位点的突变可能改变了ANL-D99P盖子结构域第二铰链区的β折叠的相对位置,从而产生了不依赖于油水界面激活的催化特性。
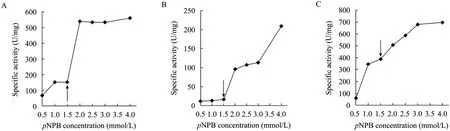
图4 pNPB浓度对黑曲霉脂肪酶及其突变体水解活性的影响. (A) ANL. (B) ANL-S84G. (C) ANL-D99PFig. 4 Influence of pNPB concentration on the specific hydrolytic activity of A. niger lipase and A. niger lipase mutants. The arrow indicates the solubility limit of pNPB. (A) ANL. (B) ANL-S84G. (C) ANL-D99P.
3 讨论
为了获得不依赖界面激活的脂肪酶突变体,先前开展了大量探索性工作,基本都集中在对组成盖子结构本身的氨基酸残基进行突变。Miled等通过缺失盖子结构域的方法获得的人胃脂肪酶突变体,其催化动力学虽然较天然脂肪酶分子表现出明显的差异,但并没有获得预期的开盖型突变体分子[9]。Carrièr等则通过脂肪酶分子之间相互置换盖子结构域,获得永久开盖的猪胰脂肪酶突变体[13]。本实验则通过蛋白质大分子结构与功能之间的对应关系,建立起脂肪酶与酯酶催化特性的差异性与对应结构的差异性之间的联系,结合生物信息学分析结果,对黑曲霉脂肪酶盖子结构两侧铰链区的氨基酸残基进行突变,获得了一个不依赖油水界面激活的黑曲霉脂肪酶突变体 (ANL-D99P)。ANL-S84G虽然依然保持界面激活的催化特性,但其催化动力学曲线与野生型ANL已发生显著性改变。本实验结果说明,脂肪酶的界面激活除了与脂肪酶的盖子结构本身有关外,盖子结构域之外的其他结构成分 (如盖子结构两侧铰链区的氨基酸残基) 也参与了油水界面脂肪酶构型的改变。进一步对脂肪酶盖子结构域更多的氨基酸残基进行定点突变和叠加突变,筛选更多的脂肪酶突变体,有可能获得性能更优良的脂肪酶突变体。同时,不依赖油水界面激活的脂肪酶突变体的构建,有利于进一步深入阐明脂肪酶界面激活的分子机制。
[1] Jaeger KE, Dijkstra BW, Reetz MT. Bacterial biocatalysts: molecular biology, three-dimensional structures, and biotechnological applications of lipases. Annu Rev Microbiol, 1999, 53(1): 315−351.
[2] Kim KK, Song HK, Shin DH, et al. The crystal structure of a triacylglycerol lipase from Pseudomonas cepacia reveals a highly open conformation in the absence of a bound inhibitor. Structure, 1997, 5(2): 173−185.
[3] Grochulski P, Li YG, Schrag JD, et al. Two conformational states of Candida rugosa lipase. Protein Sci, 1994, 3(1): 82−91.
[4] Cajal Y, Svendsen A, Girona V, et al. Interfacial control of lid opening in Thermomyces lanuginosa lipase. Biochemistry, 2000, 39(2): 413−423.
[5] Carrasco-López C, Godoy C, de Las Rivas B, et al. Activation of bacterial Thermoalkalophilic lipases is spurred by dramatic structural rearrangements. J Biol Chem, 2009, 284(7): 4365−4372.
[6] van Pouderoyen G, Eggert T, Jaeger KE, et al. The crystal structure of Bacillus subtili lipase: a minimal α/β hydrolase fold enzyme. J Mol Biol, 2001, 309(1): 215−226.
[7] Uppenberg J, Hansen MT, Patkar S, et al. The sequence, crystal structure determination and refinement of two crystal forms of lipase B from Candida antarctica. Structure, 1994, 2(4): 293−308.
[8] Verger R. ‘Interfacial activation’ of lipases: facts and artifacts. Trends Biotechnol, 1997, 15(1): 32−38.
[9] Miled N, Bussetta C, de Caro A, et al. Importance of the lid and cap domains for the catalytic activity of gastric lipases. Comp Biochem Physiol B: Biochem Mol Biol, 2003, 136(1): 131−138.
[10] Overbeeke PLA, Govardhan C, Khalaf N, et al. Influence of lid conformation on lipase enantioselectivity. J Mol Catal B: Enzymatic, 2000, 10(4): 385−393.
[11] Santarossa G, Lafranconi PG, Alquati C, et al. Mutations in the “lid” region affect chain length specificity and thermostability of a Pseudomonas fragi lipase. FEBS Lett, 2005, 579(11): 2383−2386.
[12] Secundo F, Carrea G, Tarabiono C, et al. The lid is a structural and functional determinant of lipase activity and selectivity. J Mol Catal B: Enzymatic, 2006, 39(1/4): 166−170.
[13] Carrière F, Thirstrup K, Hjorth S, et al. Pancreatic lipase structure-function relationships by domain exchange. Biochemistry, 1997, 36(1): 239−248.
[14] Brzozowski AM, Savage H, Verma CS, et al. Structural origins of the interfacial activation in Thermomyces (Humicola) lanuginosa lipase. Biochemistry, 2000, 39(49): 15071−15082.
[15] Derewenda U, Brzozowski AM, Lawson DM, et al. Catalysis at the interface: the anatomy of a conformational change in a triglyceride lipase. Biochemistry, 1992, 31(5): 1532−1541.
[16] Shu ZY, Duan MJ, Yang JK, et al. Aspergillus niger lipase: heterologous expression in Pichia pastoris, molecular modeling prediction and the importance of the hinge domains at both sides of the lid domain to interfacial activation. Biotechnol Prog, 2009, 25(2): 409−416.
[17] Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72(1/2): 248−254.
[18] Hiol A, Jonzo MD, Rugani N, et al. Purification and characterization of an extracellular lipase from a thermophilic Rhizopus oryzae strain isolated from palm fruit. Enzyme Microb Technol, 2000, 26(5/6): 421−430.
[19] Kordel M, Hofmann B, Schomburg D, et al. Extracellular lipase of Pseudomonas sp. strain ATCC 21808: purification, characterization, crystallization, and preliminary X-Ray diffraction data. J Bacteriol, 1991, 173(15): 4836−4841.
[20] Martinelle M, Holmquist M, Hult K. On the interfacial activation of Candida antarctica lipase A and B as compared with Humicola lanuginosa lipase. Biochim Biophys Acta, 1995, 1258(3): 272−276.
[21] Nardini M, Dijkstra BW. α/β hydrolase fold enzymes: the family keeps growing. Curr Opin Struct Biol, 1999, 9(6): 732−737.
