批次和流加培养中国仓鼠卵巢工程细胞的细胞周期相关基因转录谱分析
2011-02-09刘兴茂叶玲玲刘红李世崇王启伟吴本传陈昭烈
刘兴茂,叶玲玲,刘红,李世崇,王启伟,吴本传,陈昭烈
军事医学科学院生物工程研究所,北京 100071
应用全基因表达谱芯片考察哺乳动物细胞在培养过程中的基因表达谱,不仅可以反映调控细胞生长、代谢、增殖、凋亡的相关信号通路的关键基因表达状态,而且可以揭示细胞生长代谢动力学变化的内在原因,为细胞培养过程的优化控制提供理论依据[1-4]。近年来,随着基因组学和蛋白组学研究的拓展和深入,DNA芯片技术及蛋白组芯片技术越来越广泛地用于考察哺乳动物细胞自身及其在不同细胞培养工艺条件下的基因表达差异[5-8]。
批次培养和流加培养是哺乳动物细胞大规模培养技术体系中的两种最为常用的操作模式[9-10]。批次培养和流加培养过程中营养成分耗竭和代谢产物积累等环境因素的变化,必将影响到细胞周期调控基因的表达,进而对培养细胞的生长密度和细胞表达产物的生产效率产生负面影响[11]。基于此,本研究以表达人重组尿激酶原CHO工程细胞系11G-S为研究对象,试图运用基因芯片技术揭示CHO工程细胞在批次培养及流加培养不同生长阶段参与细胞周期调控相关基因的表达谱及调控细胞增殖相关信号通路的关键基因的表达状态,为探寻进一步提高细胞生长密度和细胞表达产物生产效率的可能途径提供依据。
1 材料与方法
1.1 材料
1.1.1 细胞株
表达人重组尿激酶原 CHO工程细胞系 11G-S (由军事医学科学院生物工程研究所构建、保藏)。
1.1.2 培养基
CHO工程细胞批次培养和流加培养所用无血清培养基为添加了胰岛素、腐胺、转铁蛋白、微量元素、Pluronic F-68及硫酸葡聚糖等成分的DMEM/F12 (1∶1,V/V)[12]。流加培养基为由葡萄糖、谷氨酰胺、氨基酸、微量元素、维生素、无血清添加成分、无机盐及次黄嘌呤等组成的混合液。
1.1.3 基因芯片
CHO工程细胞基因表达水平分析采用安捷伦公司 (Aglient Technologies) 的小鼠全基因组表达谱芯片小鼠全基因组芯片 (Whole mouse genome microarray kit),其性能参数见表1。
1.1.4 基因芯片分析样品
选取细胞接种时、无血清悬浮批次培养和流加培养的不同生长阶段的 CHO工程细胞用作基因分析样品,样品的选取和说明见表2。
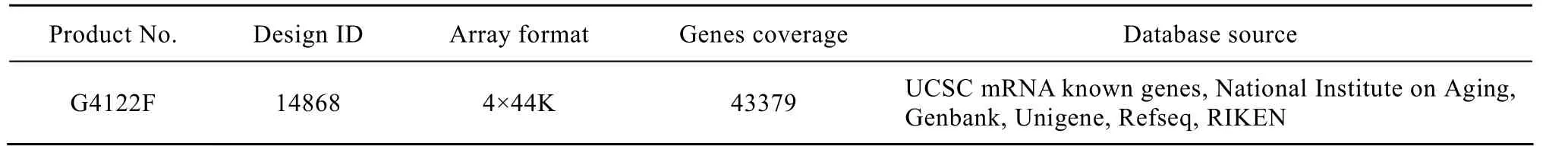
表1 Aglient小鼠全基因组表达谱芯片Table 1 Specifications of Aglient whole mouse genome microarray kit
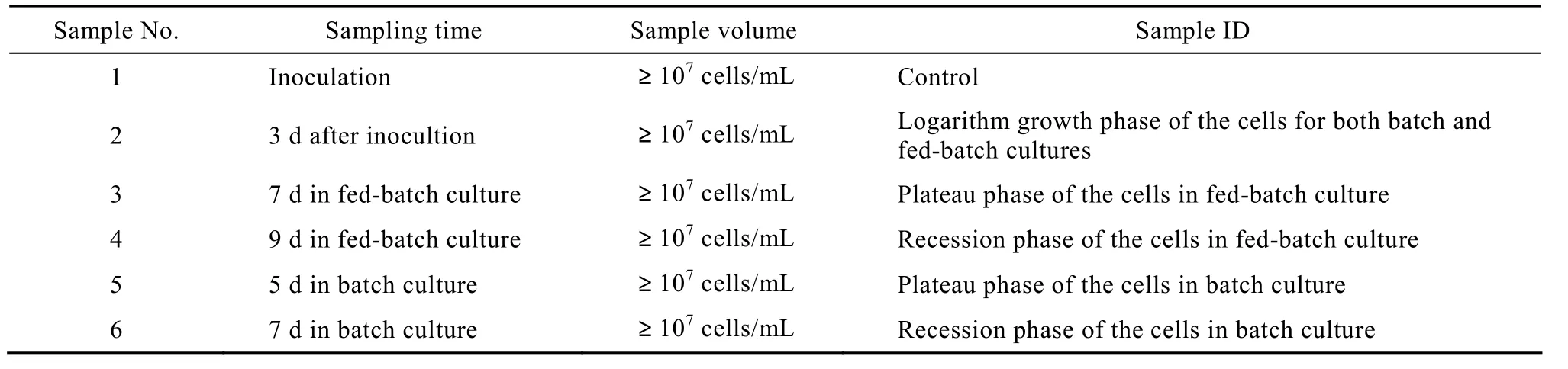
表2 用于基因芯片分析的样品Table 2 Samples for comparative transcriptional analysis by gene chip
1.2 方法
1.2.1 细胞培养
CHO工程细胞以3×105cells/mL接种于100 mL三角瓶内,培养体积 35 mL,培养时加入含有5 mmol/L谷氨酰胺、0.1% (W/V) Pluronic F-68及25 µg/mL硫酸葡聚糖的无血清培养基,37 ℃、90 r/min悬浮培养。流加培养于接种后72 h,每24 h加入3 mL流加培养基。
1.2.2 细胞周期的检测
分别于CHO工程细胞批次培养24、72、120、168 h和流加培养72、120、168、216 h各取细胞约1×106个,用75%的冰乙醇固定1 h,−20 ℃保存。流式细胞仪分析前先用PBS洗涤细胞,加入0.2 mL RNase A,37 ℃水浴消化30 min,再加入0.3 mL碘化丙啶 (Propidium iodide,PI) 置暗处染色20 min,用流式细胞仪 (BD FACSCalibur,USA) 检测细胞周期。
1.2.3 样品RNA抽提、基因芯片杂交及分析
取约1×107个细胞加入1.5 mL EP管中,采用Trizol 试剂盒提取RNA。采用苯酚/氯仿层相分离法纯化总RNA,将提取的RNA保存于−70 ℃超低温冷柜保存。用Nanodrop分光光度计测定RNA在260、280及230 nm的吸收值,以计算浓度并评估纯度。同时,用甲醛电泳试剂进行变性琼脂糖凝胶电泳,检测RNA的纯度及完整性。
用cy3荧光标记RNA样品,然后利用等量的探针进行杂交,杂交条件为42 ℃、16 h,55 ℃洗片。采用Agilent扫描仪对芯片进行扫描,读取数据,分辨率为5 µm、光电耦合装置 (Photoelectric multiplication tube,PMT) 电压100%,采用Gene spring软件进行标定处理分析,并将样品与对照比值取以2为底的对数,所得数值大于或等于1作为基因上调的筛选标准,所得数值小于或等于−1作为基因下调的筛选标准。
1.2.4 数据分析
根据测得的基因数据,首先将标准化的目标基因数据导入Genemapp软件中,并设定标准以界定上调或下调基因差异表达的程度,运用已知有关小鼠细胞周期及信号通路图,找出显著差异表达的基因。
2 结果与分析
2.1 CHO 工程细胞批次和流加培养时的生长周期分布
图1所示为CHO工程细胞无血清批次培养和流加培养过程中的活细胞密度变化。培养的前 3天,无血清批次培养和流加培养的 CHO工程细胞处于相同的培养条件,细胞处于对数生长期,活细胞密度呈现相同快速增加态势。批次培养CHO工程细胞于第4天达到41.5×105cells/mL的最大活细胞密度,随后活细胞密度呈现下降趋势,培养10 d后的活细胞密度减低至18.4×105cells/mL;流加培养CHO工程细胞的快速生长期持续至第5天,于第6天达到78.1×105cells/mL的最大活细胞密度,随后活细胞密度呈现下降趋势,培养10 d后的活细胞密度减低至50.4×105cells/mL (图1)。
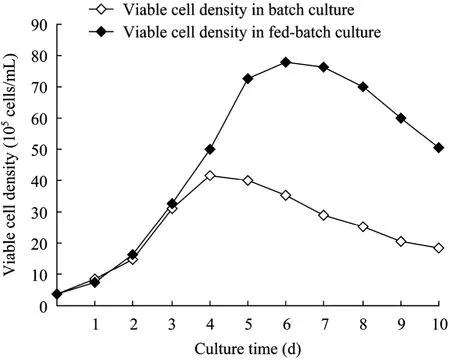
图1 CHO工程细胞在批次和流加培养过程中的活细胞密度变化Fig. 1 Profile of viable cell density of the CHO cells in both batch and fed-batch cultures.
依照 CHO工程细胞无血清批次培养和流加培养过程中的活细胞密度变化,选取批次培养 24、72、120和168 h分别代表CHO工程细胞批次培养的对数生长早期、对数生长晚期、平台期和衰退期;选取流加培养72、120、168和216 h分别代表CHO工程细胞流加培养的对数生长中期、对数生长晚期、平台期和衰退期。批次培养的CHO工程细胞随培养时间延长,G1期所占细胞比例明显升高(图2A);流加培养的 CHO工程细胞处于G2期的细胞比例随培养时间延长明显升高 (图2B)。批次培养和流加培养的CHO工程细胞中,处于S期的细胞比例均随培养进程呈现逐步下降的趋势 (图2A,B)。CHO工程细胞无血清批次培养和流加培养过程中的细胞周期分布变化与其各自的活细胞密度变化基本一致。
2.2 CHO工程细胞样品的RNA的质量鉴定
图3为各CHO工程细胞样品经RNA抽提处理后的变性琼脂糖凝胶电泳结果,各泳道均可见明亮的28S和18S核糖体RNA区带以及稍显暗淡和弥散tRNA和5S核糖体RNA区带,表明各细胞样品RNA抽提成功。表3为各提取RNA样品的紫外分光光度计检测结果,OD260/OD280比值介于1.8到2.0之间,OD260/OD280比值大于1.8,表明提取的总RNA样品质量完好。

图2 CHO工程细胞批次培养 (A) 和流加培养 (B) 过程中不同生长阶段的细胞周期分析Fig. 2 Cell cycle analysis of the CHO cells in different growth phase in both batch (A) and fed-batch (B) cultures.

图3 总RNA样品的变性琼脂糖凝胶电泳Fig. 3 Total RNA analysis of the CHO cells by denatured agarose electrophoresis. 1−6: total RNA of sample 1 to 6 respectively.
2.3 基因芯片实验数据分析
基因芯片实验数据来自于 CHO工程细胞批次培养及流加培养对数生长期、平台期及衰退期 3个阶段的样品,以培养起始接种的细胞作为对照,其他样品的每个基因标准化值与之相比,所得数值取以2为底的对数。图4A、B分别为CHO工程细胞在两种培养模式下比值大于1或小于−1及大于2或小于−2的基因上调及下调表达的差异状况。在基因芯片分析涉及的19 191个目标基因中,有差异表达的基因较多。总体而言,下调表达的基因数量多于上调表达基因数目,且上调表达基因其比值大多介于1和2之间,下调表达基因其比值大多介于−2和−1之间。在细胞生长的对数期及平台期,上调及下调表达的基因数目虽有差异,但总体上处于相对平衡的状态,说明细胞在较好的培养环境中,其细胞正负调控基因的表达相对平衡;在细胞生长的衰退期,下调表达的基因数目明显多于上调表达的数目,特别是显著下调表达的基因所占的比例显著升高,说明随着培养环境的恶化,细胞更多是以下调表达基因的方式,来适应不断变化的培养环境及维持自身的活力。对比两种培养模式的差异表达基因的方式及数量也有着明显的差异,特别是在流加培养的衰退期,下调表达的基因数量明显多于批次培养的衰退期,提示在流加培养模式下,随着培养体系中营养物、有毒代谢废物累积及渗透压的提高,细胞对环境的应激调控强度远远高于细胞在批次培养模式下由于少数营养物缺乏所产生的应激调控。

表3 用紫外吸光法测定RNA浓度和纯度Table 3 Concentration and purity of the total RNA from the CHO cells determined by ultroviolet absorption

图4 细胞无血清批培养及流加培养的基因表达差异Fig. 4 Number of differentially regulated genes of both batch and fed-batch cultures at different cell growth phases using two different significance criteria: fold change≥1 (A) or 2 (B) at P-value≤0.05. a: logarithm growth phase; b: plateau phase; c: recession phase of the cells.
2.4 两种培养模式下与细胞周期相关基因表达的差异
CHO工程细胞在批次培养和流加培养过程中,细胞周期调控基因虽然呈现基本相同的以下调表达为主的转录谱,但流加培养过程中参与细胞周期调控的上调表达基因仅为 Smad4,明显少于批次培养CHO工程细胞中的上调基因 (图5)。
在涉及细胞周期的86个主要基因中,批次培养对数期生长期细胞的差异表达基因包括,G1期上调表达的 Hdac7a基因及下调表达的 Tfdp1、Ccne2、Hdac5、E2f5基因;S期上调表达的Orc1l及下调表达的 Orc5l基因。细胞生长进入平台期时,有差异表达的基因明显增多,主要有 G1期上调表达的Gsk3b、Ccnd2、Tfjp1、Hdac7a基因及下调表达的Ccne1、Ccne2、Cdk6、Hdac5、E2f5基因;S期上调表达Trp53、Orc1l、Orc2l、Mcm2基因及下调表达的 Orc5l、Ywhag、Cdc45l、Pcna,M 期的基因Espl1。当细胞生长进入衰退期后,上调表达的基因明显减少,上调表达的基因主要有 G1期的 Smad4及Hdac7a基因;下调表达因与平台期相比没有太大的差异,下调表达的基因主要包括 G1期的基因Ccne1、Ccne2、Cdk2、Hdac5及E2f5;S期的Ywhag、Pcna、Cdk2、Cdc45l、Orc4l及Orc5l基因;G2期的Cdc2a基因和M期的Espl1基因。Hdac7a基因在CHO细胞批次培养的各阶段均上调表达,Ccne2、Orc5l、Hdac5及E2f5在批次培养的各阶段均下调表达,Ywhag、Pcna、Ccne1、Cdc45l及Espl1在批次培养的平台期及衰退期均下调表达。
细胞在流加培养模式下的流加培养阶段差异表达的基因以下调表达为主,且与批次培养模式下细胞的平台期及衰退期差异表达的基因大体相近,上调表达的基因仅有Smad4基因。在流加培养的平台期,下调表达的基因主要有G1期的Ccne1及E2f1,S期的Ywhag,G2期Cdc2a及Plk1,M期的Espl1;当细胞生长进入衰退期后,差异表达的基因明显增加,并且基本上都为下调表达,仅有 G1期的基因Smad4上调表达。下调表达的基因包括G1期的基因Ccne1、Ccne2、Hdac5、Tfdp1、Hdac7a及E2f1,S期的基因Ywhag、Ccnh、Orc5l、Cdc45l及Cdc7,G2期的基因Mad2l1、Cdc2a及Plk1,M期的基因Espl1。
3 讨论
细胞周期进程的实现有赖于各级调控因子对细胞周期精确而严密的调控。这些调控因子的核心是细胞周期蛋白依赖性蛋白激酶 (Cyclin dependent kinases,CDKs) 及其正、负性调控因子——细胞周期蛋白 (Cyclin) 和细胞周期蛋白依赖性蛋白激酶抑制剂 (CDI)[13-14]。CDKs属于丝氨酸/苏氨酸蛋白激酶家族,可在特定的细胞周期被激活,之后磷酸化相应的底物,从而引起后续事件的发生。Cyclin作为蛋白激酶复合体的调节亚基,对 CDKS起着正性调节作用。在高等真核细胞中,Cyclin 分为A、B、C、D、E和H六类。它们分别在细胞周期的不同时相中合成、积累,并与相应的CDKs结合,激活CDKs的蛋白激酶活,从而调节细胞周期进程。同时 CDKs的活性也可以被细胞周期抑制蛋白(Cell cycle inhibitory proteins,CKIs) 所抑制。
从依照细胞周期调控的信号通路图分析,批次培养对数期生长期的CHO工程细胞,仅Cyclin E2发生了下调表达,其他基因表达均没有明显差异。随着细胞生长进入平台期及衰退期,CDKs、Cyclin家族中的相应基因的数目在细胞的G1、S及G2期下调表达明显增加,主要有基因 Cdk6、Cdk2、Cdc2a、Ccne1及Ccne2,且细胞在衰退期下调表达基因多于平台期。同时TGF2β信号转导途径的中心分子Smad4在批次培养CHO工程细胞的衰退期上调表达。研究表明,Smad4的失活是引起细胞生长抑制丧失的一个重要原因[15]。批次培养CHO工程细胞在衰退期通过上调表达Smad4进一步抑制了细胞的生长。反映在细胞周期分布上,CHO工程细胞在对数期 G1期细胞所占的比例较低,S期细胞所占的比例较高,细胞可以快速增殖;在进入培养的平台期及衰退期后,G1期细胞所占的比例不断升高,S期细胞所占的比例却逐渐降低,大多数细胞停滞于G1期,细胞生长缓慢或停止。
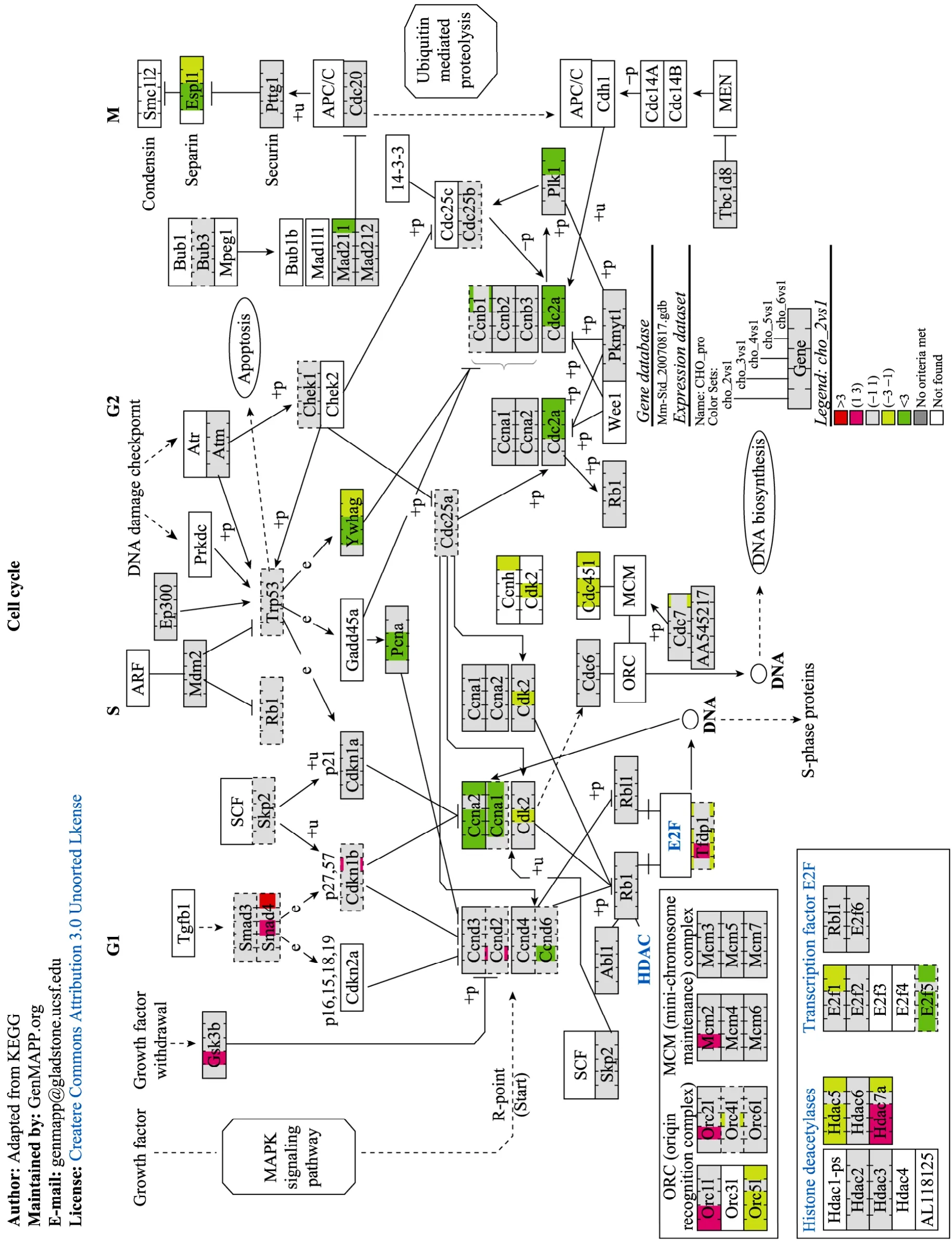
图5 批次和流加培养中细胞的细胞周期相关基因转录谱Fig. 5 Transcriptional profiling of cell cycle regulatory networks of cells in batch and fed-batch cultures.
流加培养 CHO工程细胞在细胞生长的平台期及衰退期,CDKs、Cyclin及CKI家族中的相应基因在G1、S及G2期下调表达基因的数量逐渐增加,且衰退期下调表达的基因多于平台期。与批次培养平台期及衰退期 CHO工程细胞下调表达的基因数目相比,流加培养相应阶段CHO工程细胞下调表达的基因少于批次培养。同时Smad4在培养衰退期也发生了上调表达,且强度高于批次培养。表明由于导致 CHO工程细胞在批次培养及流加培养模式下增殖减缓的原因不同,因此在细胞周期调控基因的表达也有所差异。反映在细胞周期分布上,CHO工程细胞在流加培养过程中,G1期细胞所占的比例呈现出先降后升的变化趋势,S期细胞所占的比例呈现出先升后降的变化趋势。
本研究获得的批次培养和流加培养 CHO工程细胞的细胞周期调控基因表达谱提示,CHO工程细胞在批次培养和流加培养过程中,主要是通过下调表达CDKs、Cyclin及CKI家族中的Cdk6、Cdk2、Cdc2a、Ccne1、Ccne2基因及上调表达Smad4基因,实现细胞增殖的调控。
REFERENCES
[1] Griffin TJ, Seth G, Xie H, et al. Advancing mammalian cell culture engineering using genome-scale technologies. Trends Biotechnol, 2007, 25(9): 401−408.
[2] Kantardjieff A, Nissom PM, Chuah SH, et al. Developing genomic platforms for Chinese hamster ovary cells. Biotechnol Adv, 2009, 27(6): 1028−1035.
[3] Charaniya S, Karypis G, Hu WS. Mining transcriptome data for function-trait relationship of hyper productivity of recombinant antibody. Biotechnol Bioeng, 2009, 102(6): 1654−1669.
[4] Yee JC, Gerdtzen ZP, Hu WS. Comparative transcriptome analysis to unveil genes affecting recombinant protein productivity in mammalian cells. Biotechnol Bioeng, 2009, 102(1): 246−263.
[5] Lee YY, Wong KTK, Nissom PM, et al. Transcriptional profiling of batch and fed-batch protein-free 293-HEK cultures. Metab Eng, 2007, 9(1): 52−67.
[6] Wong DC, Wong KTK, Lee YY, et al. Transcriptional profiling of apoptotic pathways in batch and fed-batch CHO cell cultures. Biotechnol Bioeng, 2006, 94(2): 373−382.
[7] Yee JC, de Leon Gatti M, Philp RJ, et al. Genomic and proteomic exploration of CHO and hybridoma cells under sodium butyrate treatment. Biotechnol Bioeng, 2008, 99(5): 1186−1204.
[8] djieff AK, Jacob NM, Yee JC, et al. Transcriptome and proteome analysis of Chinese hamster ovary cells under low temperature and butyrate treatment. J Biotechnol, 2010, 145(2): 143−159.
[9] Merten OW. Introduction to animal cell culture technology−past, present and future. Cytotechnology, 2006, 50(1/3): 1−7.
[10] Whitford WG. Fed-batch mammalian cell culture in bioproduction. BioProcess International, 2006, 4: 30−40.
[11] Wlaschin KF, Hu WS. Fedbatch culture and dynamic nutrient feeding. Adv Biochem Engin/Biotechnol, 2006, 101: 43−74.
[12] Liu XM, Liu H, Ye LL, et al. Serum-free medium for suspension culture of recombinant Chinese hamster ovary (11G-S) cells. Chin J Biotech, 2010, 26(8): 1116−1122.刘兴茂, 刘红, 叶玲玲, 等. CHO工程细胞 (11G-S) 悬浮培养的无血清培养基的设计. 生物工程学报, 2010, 26(8): 1116−1122.
[13] Loyer P, Trembley JH, Katona R, et al. Role of CDK/cyclin complexes in transcription and RNA splicing. Cell Signal, 2005, 17(9): 1033−1051.
[14] Morgan DO. Cyclin-dependent kinases: engines, clocks, and microprocessors. Annu Rev Cell Dev Biol, 1997, 13: 261−291.
[15] Attisano L, Wrana JL. Signal transduction by the TGF-beta superfamily. Science, 2002, 296(5573): 1646−1647.
