龙须菜UDP-葡萄糖焦磷酸化酶基因的克隆及其表达与琼胶产量的关系*
2011-01-10隋正红丁弘叶
张 杨,隋正红,丁弘叶,仲 洁
(中国海洋大学海洋生命学院海洋生物遗传育种教育部重点实验室,山东青岛266003)
龙须菜(Gracilaria leimaneiform is)属红藻门江蓠科江蓠属,是一种可用于琼胶提取的重要经济红藻。UDP葡萄糖焦磷酸化酶(UGPase)是合成UDP葡萄糖(UDPGlc)的重要酶,它以U TP和葡萄糖-1-磷酸(Glc1p)为底物合成UDPGlc。UDPGlc在红藻的碳代谢过程中扮演了重要的角色,它是半乳糖苷和红藻糖苷合成的前体物质[1-3]。半乳糖苷(或者称为半乳糖聚物)是多数红藻细胞壁也就是琼脂和角叉菜胶最主要的组成成分[4-6]。红藻糖苷是一种重要的光合产物,它的作用是作为短期的低分子量的碳水化合物的碳库。UDPGlc在某些红藻中还作为红藻淀粉生物合成过程中次级葡萄糖基的供体[7-10],这一过程与真核生物中糖原的合成途径类似[8,11-12]。
早在1960年代,UGPase便被分离出来,证明红藻中存在该酶[13],但到目前为止,只有少数几种红藻UGPase基因的研究报道。在细翼枝藻Pterocladia capillacea中UGPase的活性曾被阐述[14],在江蓠Gracilaria gracilis中有蛋白和基因水平的研究[15];日本凋毛藻Grif fithsia japonica UGPase的基因仅有部分序列在NCB I数据库中提交(GI:32401296)。迄今尚未见UGPase在龙须菜中的特征报道,也不清楚龙须菜中是否存在该反应过程。
鉴于UGPase在红藻碳储存物质尤其是在琼胶代谢中的重要作用,作者克隆了龙须菜中UGPase的cDNA和基因组DNA序列,采用荧光实时定量PCR的方法检测了不同琼胶含量藻株UGPase基因的表达变化,揭示了龙须菜的琼胶含量与UGPase基因表达的相关性,并推测在龙须菜中存在该酶催化的对应反应。
1 材料和方法
1.1 材料
实验材料龙须菜采自青岛湛山湾。将采集的新鲜藻体用灭菌水冲洗,去除藻体表面的泥沙,盐分和杂藻。用吸水纸将表面水分吸除,鲜活藻体用于核酸的提取,干燥后藻体用于琼胶的提取。
1.2 核酸的提取
龙须菜基因组DNA的提取使用TIANGEN公司的植物基因组DNA提取试剂盒。取鲜藻体100 mg经液氮研磨至粉末状,装入700μL GP1缓冲液混匀后于65℃水浴45 min。等体积酚氯仿抽提离心后,吸取水相至新的离心管并加入700μL GP2,混合充分后转入吸附柱离心、弃废液。加入500μL GD缓冲液于吸附柱,离心去除蛋白,用700μL PW漂洗液漂洗2次,离心并晾干残余漂洗液。最后滴加50μL洗脱液洗脱吸附柱上的DNA,收集DNA于-20℃储存。
总RNA的提取使用OM EGA生物技术公司的Plant RNA Kit。取新鲜藻体尖部100 mg,液氮中研磨至粉末状,加入到盛有500μL裂解液RB的离心管中,涡旋裂解1 min之后,依照试剂盒步骤提取总RNA。用TaKaRa公司的DNase I去除RNA中的DNA。将RNA于-80℃储存备用。
1.3 UGPase基因的克隆
1.3.1 UGPase基因部分序列的获得 采用PCR的方法获得UGPase基因的部分序列。引物序列参考文献[15],且在引物列表中列出(见表1,引物A和B)。以基因组DNA为模板,扩增获得了330 bp的龙须菜UGPase基因片段。PCR扩增在Thermo Electron公司的Thermal Cycler热循环仪上进行。反应体系25 μL中含有10μmol/L的正、反向引物各1μL,2.5 mmol/L的dN TP 2μL,5U的Taq DNA聚合酶和1μL基因组DNA。扩增条件为:94℃5 min,1个循环;94℃1 min;55℃1 min;72℃1 min,30个循环;72℃5 min,1个循环。PCR产物于1.5%的琼脂糖凝胶上进行电泳检测。
1.3.2 3’RACE 按照TaKaRa公司的3’-Full RACE试剂盒操作获得UGPase基因3’端序列。根据已知的UGPase基因部分序列设计特异引物3’213(见表1),与试剂盒中的锚定引物3’Site A dap to r Primer进行PCR扩增。反应条件为94℃3 min,1个循环;94℃1 m in;60℃1 m in;72℃1 min,30个循环;72℃5 min,1个循环。于1%琼脂糖凝胶电泳检测PCR产物的分子量。
1.3.3 5’RACE 5’端序列的获得使用Clontech的SMART RACE cDNA Amp lification试剂盒。设计特异的外侧引物5’288和内侧引物5’214(见表1),分别与试剂盒中的锚定引物10×Universal Primer A M ix和Nested Universal Primer A进行巢式PCR反应。外侧PCR反应条件为94℃30 s;65℃30 s;72℃1 min,25个循环;72℃3 min,1个循环。内侧巢式PCR反应以外侧PCR反应产物10倍稀释后取1μL作为模板,于50μL体系中进行,其中反应循环数为30,其它条件不变。
1.3.4 UGPase基因DNA序列的获得 根据UGPase基因的已知序列设计引物Qus和Qua(见表1),以基因组DNA为模板进行扩增,反应条件为94℃5 m in,1个循环;94℃1 m in;60℃1 min;72℃1 m in 30 s,30个循环;72℃5 min,1个循环。于1%琼脂糖凝胶电泳检测PCR产物。

表1 本研究中采用引物的特征Table 1 Characterization of primers employed in this study
1.3.5 回收和克隆 所有的PCR产物片段均通过琼脂糖凝胶的PCR产物回收试剂盒进行回收(OM EGA生物技术公司),将目标片段连接到质粒载体pMD18-T(TaKaRa公司)上,转入细菌DH5α,经过氨苄青霉素抗性筛选,挑取单菌落,分别以特异引物和通用引物M 13R/M 13F进行PCR反应,检测分子量,然后对克隆产物测序。
1.3.6 序列分析 基因序列特征通过网站软件进行分析(http://www.dna.affrc.go.jp/PLACE)。以Clustal W进行多序列对位分析,用于与龙须菜UGPase氨基酸序列进行比对的数据来自于NCBI数据库。
1.4 Southern blotting
Southern杂交实验过程均依照标准进行[16]。提取的DNA各5μg,用3种限制性内切酶EcoRI,、Xba I和Bam H I进行酶切消化后,于1.5%的琼脂糖凝胶电泳进行分离。凝胶经过变性和中和后,通过虹吸法将DNA转移到硝酸纤维素膜上,转移时间为20 h。采用以引物A和B[15]扩增所得的330 bp基因组序列,经Roche公司的Dig High-Prime labeling and detection starter试剂盒制备和标记探针。
1.5 琼胶的提取
参照文献[17]的方法略加改动。将收获的每株鲜藻体分为3份,每份2 g左右,于60℃烘干至恒重,称量记录每份藻体干质量。按照每0.1 g藻体干重添加4 m L NaOH(质量浓度2.5%)的比例向每份样品中添加NaOH溶液,放入恒温水浴中(85℃)加热碱处理2 h。用4层纱布过滤,蒸馏水洗涤藻体至p H=6.5左右,将水洗后的龙须菜放在锥形瓶中,按照每0.1 g藻体干质量添加6 mL蒸馏水的比例向每份样品中添加蒸馏水,温度保持120~125℃于高压锅中煮2 h。降温后用8层纱布过滤,过滤物在室温凝固后-20℃冷冻过夜。冷冻物解冻后用蒸馏水冲洗,于60℃24 h烘干至恒重[18],称量并记录琼胶干质量。琼胶含量=琼胶干质量/藻体干质量(g/g)。
1.6 实时定量PCR检测不同琼胶含量组藻体中U GPase基因的表达量
1.6.1 不同琼胶含量藻株的cDNA的合成 采用Promega公司的M-MLV Reverse Transcrip tase将龙须菜RNA反转录为cDNA。反应体系为:总RNA 1 μg,Oligo d T 0.5μg于70℃孵育5 min,冰上放置,在每个反应管中加入5×M-MLV Buffer 5μL,Inhibito r 1μL,dN TPM ix(10 mmol/L each)1μL,M-MLV Reverse Transcrip tase 1μL,添加RNAase free water至总体积25μL,在42℃金属浴中孵育60 min进行反转录,70℃孵育10 min终止反应,-20℃储存备用。1.6.2实时定量PCR Real-time RT-PCR选用TOYOBO公司的SYBR green Real time PCR Master M ix,于AB I公司的7500荧光定量PCR仪上进行实时定量PCR检测。反应体系为20μL,其中包含有10 μL的2×SYBR Green Real-time PCR Master M ix,荧光定量PCR正、反向引物各1μL,反转录所得的cDNA 1μL,其余的用ddH2O补足。反应程序为两步法:95℃60 s,1个循环;95℃15 s;60℃60 s,40个循环。以每株藻体的cDNA为模板做3个平行样,采集荧光信号。选择在龙须菜中表达较为恒定的gap dh基因作为内参,引物为gap dhs和gapdha[19],扩增可得到163 bp的gapdh基因的部分序列。设计U GPase基因的引物Yus和Yua(见表1),扩增可得到224 bp的UGPase基因部分序列。采用相对定量检测的2-△△CT法来分析目的基因的表达情况[20]。其中△CT=CTU GPase-CTgapdh;△△CT=△CT目标样品-△CT校正样品;2-△△CT=表达量的比值。
1.7 数据统计方法
琼胶含量和U GPase基因相对表达量的数据采用单因素方差分析和t检验的方法分析差异显著性。
2 结果与讨论
2.1 U GPase基因的克隆和序列分析
克隆得到的龙须菜(Gracilaria leim aneiform is)UGPase基因的DNA与cDNA序列一致。cDNA序列长度为2 200 bp,其中包含1个由1 485 bp组成的完整的开放阅读框(ORF),可翻译成494个氨基酸残基和终止密码子UAG,3’U TR长度为385 bp,5’U TR长度为330 bp(GenBank Accession No.GU 586070)。
龙须菜UGPase基因的494个编码氨基酸中,有59个酸性氨基酸,54个碱性氨基酸,168个疏水性氨基酸,138个极性氨基酸,编码了54.727 kD的蛋白,其等电点为6.096。其中氨基酸的数量与G.gracilis的495个氨基酸相差不大,龙须菜UGPase基因的氨基酸序列与其它物种的UGPase的氨基酸序列相似性较高(见图2)。其中与G.gracilis UGPase基因的相似性为91%,与G.japonica U GPase基因的相似性为74%,与其他物种的相似性为53%~57%。该酶含有5个赖氨酸残基的保守位点(见图2),这些残基对于与底物结合以及催化反应有着极其重要的作用[21-23]。以龙须菜序列为基准,其中K278,K344与磷酸盐结合或者与α-D-glucose-1-phosphate结合有关,K382与催化自身磷酸化作用有关[21]。氨基酸序列对比结果,在10个物种中都高度保守的氨基酸为“KLNGGLGT”和“KNSF”,在9个物种中都高度保守的氨基酸序列为“PPGHGD”和“DNLGA”,但还没有发现关于这些位点在红藻中所行使功能的报道。
本文利用SMART技术得到了龙须菜UGPase的完整5’端cDNA序列,因为该技术利用具有末端转移酶活性的5’-帽子结构依赖型反转录酶,仅对具有完整5’-帽子结构的m RNA模板进行反转录,并在反转录进行到m RNA 5’末端时,在反转录产物的3’端加上3个胞嘧啶核苷酸残基(CCC),使SM ART Oligo GGG引物结合上去进行第二链的延伸,通过模板转换,为引物提供结合部位,保证了反转录产物5’末端的完整性[24]。获得的5’U TR部分包含330 bp,该基因从起始密码子A TG前330 bp处的碱基A开始转录。5’侧翼序列的翻译初始位点序列为GCTA TG,与多数红藻的RCYA TG的翻译起始特征一致[25]。3’U TR部分包含385 bp,在终止密码子UAG之后261 bp处存在1个3’末端多聚A前的加尾信号:A TTA TT,与龙须菜半乳糖-1-磷酸尿苷酰转移酶的加尾信号相同[19],但不同于许多红藻基因中比较保守的TAAA加尾信号。在江蓠G.gracilis中几乎所有核基因的转录本的加尾信号都出现TAAA[25],它的U GPase基因3’非编码区多聚A前加尾信号为AA TAAA[15]。表明龙须菜G.lemaneifo rmis部分基因与江蓠G.gracilis等红藻的基因在转录调控机制上可能存在不同。
许多红藻中的核基因不含有内含子,例如江蓠G.gracilis中的磷酸丙糖异构酶[26]、龙须菜G.lem aneiform is的半乳糖-1-磷酸尿苷酰转移酶[19]和角叉菜Chondrus crispus中的gapdh基因[27]。与江蓠G.gracilis的UGPase基因相同,龙须菜的UGPase基因内也没有内含子。
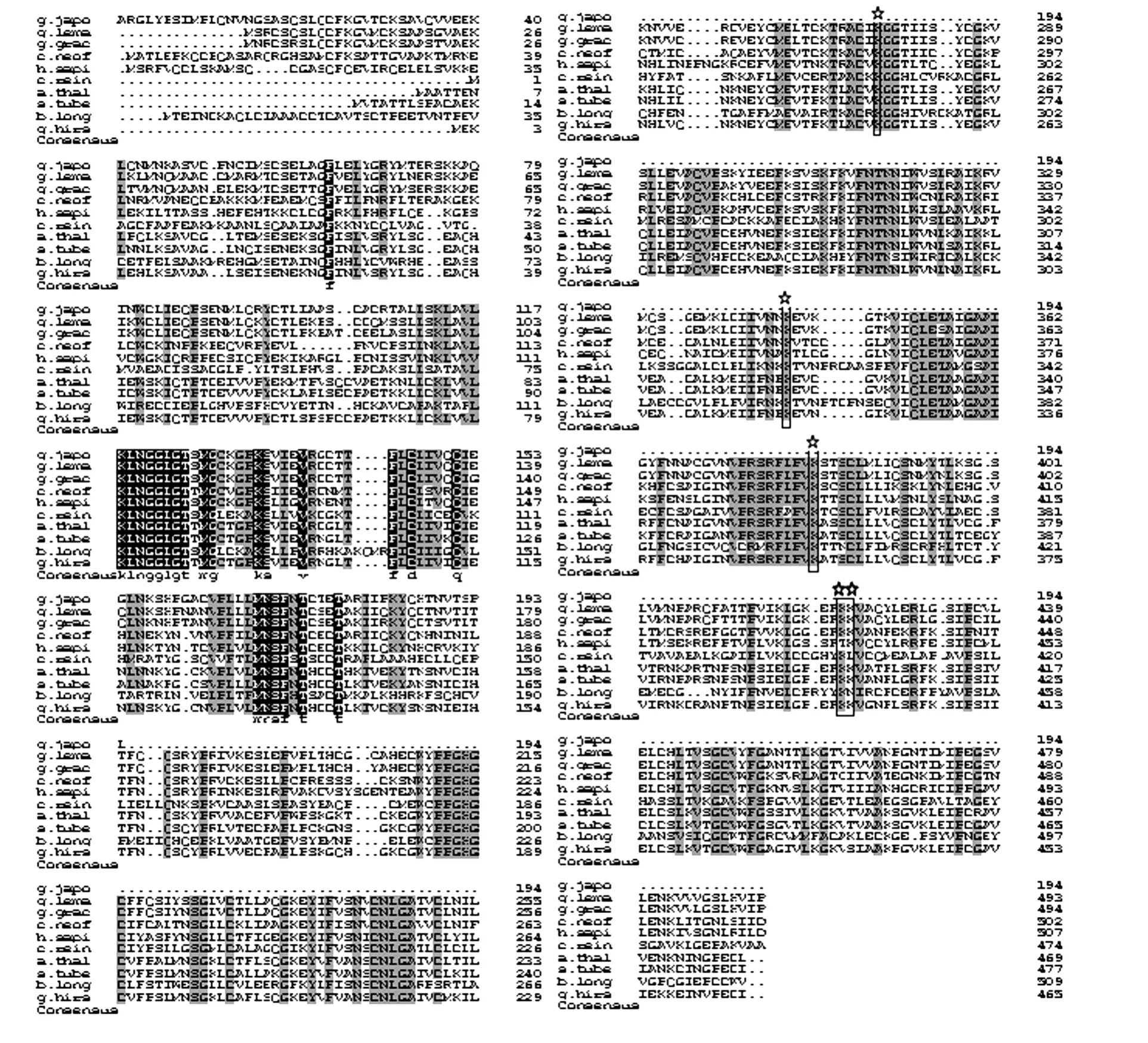
图1 UDP葡萄糖焦磷酸化酶(UGPase)的氨基酸序列比对结果Fig.1 The amino acid sequences alignment of UDP-glucose pyrophospho rylase(UGPase)
2.2 Southern blotting分析
Southern实验检测了龙须菜基因组中UGPase基因的拷贝数(见图3)。经检测UGPase基因是单一拷贝,表明在龙须菜中不存在UGPase的假基因,因此也排除了在后续的荧光定量分析中可能存在的假基因表达的干扰。
红藻中大多数的功能基因都是单拷贝。在Galderia su lphuraria中发现UDP-葡萄糖尿苷基转移酶基因是单一拷贝[28],龙须菜的UDP-葡萄糖尿苷基转移酶基因是单一拷贝[19],江蓠G.gracilis中UGPase基因是单一拷贝[15]。在土豆中发现UGPase基因也是单一拷贝[29]。
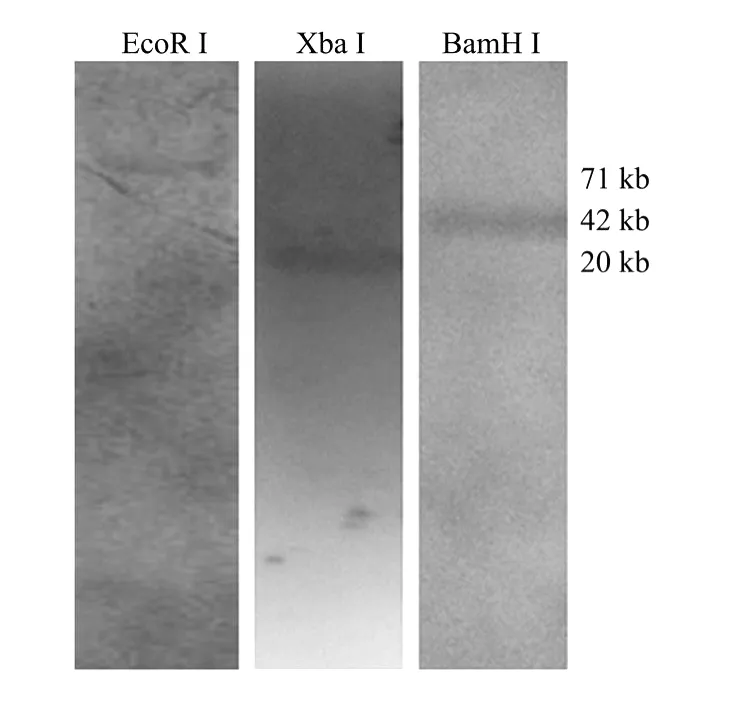
图2 UGPase基因的southern杂交结果Fig.2 Southern blo t hybridization of the UGPase gene
2.3 U GPase基因的表达与藻体琼胶含量的相关性分析
为研究UGPase基因在琼胶合成过程中的作用,文中测定了野生龙须菜的琼胶含量,选取其中的14株,对其UGPase基因表达进行了分析。根据不同藻株所提取的琼胶含量的高低,将这14个藻株划分为高琼胶含量和低琼胶含量两组(见图3),两组材料的琼胶含量差异显著(P<0.01)。
以实时定量PCR检测UGPase基因的相对表达(见图3),研究发现:UGPase基因的表达量在高琼胶含量藻株中都高于低琼胶含量藻株,高琼胶组藻株平均的2-△△CT值约是低琼胶组藻株的24倍。经统计学分析表明P<0.01,高琼胶组与低琼胶组的2-△△CT值间存在显著差异,证明了UGPase基因表达与龙须菜琼胶含量间存在显著相关性。

图3 不同藻株不同YIdeed in this study的琼胶含量及其对应的UGPase基因相对表达量。Fig.3 Agar contents of different algal strains and their exp ression levels of UGPase
UGPase基因在高琼胶组藻株中的表达量高,在低琼胶组藻株中的表达量低,说明UDP葡萄糖焦磷酸化酶是琼胶合成途径中的一种重要酶。然而,文中对龙须菜琼胶合成过程的了解还很有限。最近报道,从龙须菜中获得的与琼胶代谢相关的另一个酶—GAL T(Galactose-1-phosphate uridylyltransferase),发现该酶基因的表达与藻体琼胶含量有一定的相关性[19],涉及该途径的其它成分及其调控特征则没有报道,而琼胶代谢是龙须菜碳代谢的重要部分,因此从理论上有必要进一步加强相关研究。
另一方面,在同一海区中,藻体琼胶含量的差异主要是藻体本身遗传因素造成的。基因表达与琼胶含量的相关性揭示了基因表达情况可作为检测藻体琼胶含量的指标的潜力。龙须菜作为重要的产琼胶海藻,其人工栽培在本世纪取得了长足的发展[30],先后在福建、广东、山东、江苏等省取得成功,栽培面积超过13 000 hm2,年产量达到万t(干质量),产值逾十亿元,成为我国继海带和紫菜之后的第三大海藻栽培业,并促进了琼胶制造业的发展。产业的发展需要种质选育作为后盾,对于龙须菜来说选育的首要性状就是琼胶含量,因此相关分子标记或检测技术的开发对产业化具有重要意义。通过UGPase基因表达量与龙须菜琼胶含量的研究,研究发现UGPase基因的表达量可以作为衡量琼胶产量高低的一个指标,可对琼胶产量起指示作用,有可能作为种质筛选的分子标记而应用于产业化。
[1] Su J C,Hassid W Z.Carbohydratesand nucleotides in the red alga Po rphyra perforata II.Separation and identi?cation of nucleotides[J].Biochem istry,1962,1:474-480.
[2] Simon-Colin C,Kervarec N,Pichon R,et al.NMR13C-isotopic enrichment experiments to study carbon partitioning into organic solutes in the red alga Grateloupia dotyphora[J].Plant Physiology and Biochemistry,2004,42(1):21-26.
[3] Karsten U,Gors S,Eggert A,et al.Trehalose,digeneaside and flo ridoside in the Flo rideophyceae(Rhodophyta)a re-evaluation of its chemotaxonomic value[J].Cambridge:Phycologia,2007,46(2):143-150.
[4] Craigie J S.Cell walls Biology of the Red Algae[M].Cambridge:Cambridge University Press,1990,221-257.
[5] Marinho-Soriano E,Bourret E.Polysaccharides from the red seaweed Gracilaria dura(Gracilariales,Rhodophyta)[J].Bioresource Technology,2005,96:379-382.
[6] Marinho-Soriano E.Agar polysaccharides from Gracilaria species(Rhodophyta,Gracilariaceae)[J].Journal of Biotechnology,2001,89:81-84.
[7] Nagashima H,Nakamura S,Nisizawa K,et al.Enzymic synthesis of?oridean starch in a red alga,Serraticardia maxima[J].Pl.Cell Physiol,1971,12:243-253.
[8] Patron N J,Keeling PJ.Common evolutionary origin of starch biosynthetic enzymes in green and red algae[J].J Phycol.2005,41:1131-1141.
[9] Barbier G,Oesterhelt C,Larson M D,et al.Compartive genomics of two closely related unicellular thermo acidophilic red algae,Gracilaria sulphuraria and Cyanidioschyzonmerolae,Reveals the molecular basisof themetabolic flexiblility of Gracilaria sulphuraria and significant differences in carbohydrate metabolism of both algae[J].Plant Physiology.2005,137:460-474.
[10] Viola1 R,Nyvall P,Pedersen M.The unique features of starch metabolism in red algae[J].The Royal Society B.2001,268:1417-1422.
[11] Peat S,Turvey J R.Evans J M.The structure of foridean starch.II.Enzymic hydrolysis and other studies[J].Chem,1959,Soc.3341-3344.
[12] Patron N J,Keeling P J.Common evolutionary origin of starch biosynthetic enzymes in green and red algae[J].Phycol,2005,41:1131-1141.
[13] Su JC,Hassid W Z.Carbohydrates and nucleotides in the red alga Porphyra perforata II.Separation and identi?cation of nucleotides[J].Biochemistry,1962,1:474-480.
[14] Manley S L,Burns D J.Formation of nucleoside diphosphate monosaccharides(NDP-sugars)by the agarophyte Pterocladia capillacea(Rhodophyceae)[J].Phycol,1991,27:702-709.
[15] Lluisma AO,Ragan M A.Characterization of a UDP-glucose pyrophosphorylase gene from themarine red alga Gracilaria gracilis[J].Phycol,999,10:581-588.
[16] Sambrook J,Fritisch E F,Maniatis T.Molecular cloning:a laboratory manual,2nd edn[M].New York:Cold Sp ring Harbor Laboratory Press,1989.
[17] 薛志欣,杨桂朋,王广策.龙须菜琼胶的提取方法研究[J].海洋科学,2006,30(8):71-77.
[18] Marinho-Soriano E,Bourret E,De Casabianca M L,et al.Agar from the rep roductive and vegetative stages of Gracilaria bursapastoris[J].Bioresour Technol.1999,67:1-5.
[19] Li M,Sui Z H,Kang K H,et al.Cloning and analysisof the galactose-1-phophate uridylyltransferase(galt)gene of Gracilaria lemaneiform is(Rhodophyta)and correlation between gene expression and agar synthesis[J].Appl phycol.2009.DOI:10.1007/s10811-009-9435-8
[20] Livak K J,Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T))method[J].Methods,2001,25(4):402-408.
[21] Katsube T,Kazuta Y,Tanizawa K,et al.T.Exp ression in E.coliof UDP-glucose pyrophosphorylase cDNA from potato tuber and functional assessment of the five lysyl residues located at the substrate-binding site[J].Biochemistry,1991,30:8546-8551.
[22] Kazuta Y,Omura Y,Tagaya M,et al.Identification of lysyl residues located at the substrate-binding site in UDP-glucose pyrophosphorylase from potato tuber:affinity labeling with uridine diand triphosphopyridoxals[J].Biochemistry,1991,30:8541-8545.
[23] McCoy J G,Bitto E,Bingman C,et al.Structure and dynamics of UDP–glucose pyrophosphorylase from A rabidopsis thaliana with bound UDP-glucose and U TP[J].JMol Biol.,2007,366:830-841.
[24] Zhu Y Y,Machleder E M,Chenchik A,et al.Reverse transcriptase template switching:A SMARTMapp roach for fulllength cDNA library construction[J].Bio Techniques,2001,30:892-897.
[25] Zhou Y H,Ragan M A.Nuclear-encoded p rotein-encoding genes of the agarophyte Gracilaria verrucosa(Gracilariales,Rhodophyta)[J].Hydrobiologia,1996,326-327:429-436.
[26] Zhou Y H,Ragan M A.Cloning and characterization of the nuclear gene and cDNAs for triosephosphate isomeraseof themarine red alga Gracilaria verrucosa[J].Curr.Genet,1995,28:317-323.
[27] Liaud M F,Valentin C,Brandt U,et al.The GAPDH gene system of the red alga Chondrus crispus:p romotor structures,intron/exon organization,genomic complexity and differential exp ression of genes[J].Pl.Mol.Biol,1993,23:981-994.
[28] GrossW G,Schnarrenberger C.Purification and characterization of a galactose-1-phophate:UDP-glucose uridylyltransferase from the red alga Gald ieria sulphuraria[J].Eur J Biochem,2001,234:258-263.
[29] Borovkov A Y,M cClean P E,Secor GA.Organization and transcription of the gene encoding potato UDP-glucose pyrophosphorylase[J].Gene,1997,186:293-297.
[30] Zhang X C,Fei X G.Qualified breeding species by National Marine Origin Breed and Fine Breed Committee-an introduction to 981 Gracilariopsis lemaneiformis and its cultivation[J].Sci Fish Farming,2008,6:21-22.
