基于ND4和ND5基因序列分析的鳅超科鱼类系统发育关系
2010-12-25刘思情张家波唐琼英刘焕章
刘思情,张家波,唐琼英,刘焕章,*
(1. 华中农业大学,湖北 武汉 430070;2. 中国科学院水生生物研究所,湖北 武汉 430072)
基于ND4和ND5基因序列分析的鳅超科鱼类系统发育关系
刘思情1,2,张家波1,唐琼英2,刘焕章2,*
(1. 华中农业大学,湖北 武汉 430070;2. 中国科学院水生生物研究所,湖北 武汉 430072)
ND4和ND5是线粒体基因组中编码NADH脱氢酶亚基4和亚基5的两个蛋白质编码基因。该研究以鳅超科鱼类为研究对象,新测定了10个物种的ND4和ND5基因全序列以及中间的3个tRNA基因共212 bp的序列,结合从GenBank下载的15个物种的15条序列进行序列比较和系统发育关系分析。结果显示:鳅超科鱼类ND4基因全长1 380~1 387 bp,以ATG为起始密码子,终止密码子为不完全终止信号;ND5基因全长1 821~1 839 bp,同样起始密码子为ATG,终止密码子为TAA或TAG;ND4和ND5基因之间插入了3个tRNA基因,分别编码携带组氨酸、丝氨酸、亮氨酸的tRNA。ND4和ND5基因(包含3个tRNA基因)中A、T、G、C的平均含量分别为30.4%、27.3%、14.2%、28.1%,A+T (57.7%)的含量高于G+C (42.3%)的含量。转换与颠换比(Ti/Tv)平均值为1.586。选取斑马鱼和鲤鱼作为外类群,采用最大简约法(MP)、最大似然法(ML)和贝叶斯推断法(BI)进行系统发育树的重建。三种方法的系统发育分析结果都显示:花鳅亚科、条鳅亚科、沙鳅亚科、平鳍鳅科及Vaillantellidae分别构成单系;它们的系统发育关系为:(Vaillantellidae+(沙鳅亚科+(花鳅亚科+(条鳅亚科+平鳍鳅科)。这与线粒体全基因组和某些核基因(如RAG1基因)的研究结果类似,且支持率较高,表明ND4和ND5基因用于鳅超科鱼类的系统发育分析是可行的;但是该研究的结果有别于其他线粒体基因的分析结果,如基于cytb和D-loop基因进行的系统发育分析表明,条鳅亚科和花鳅亚科聚为姐妹群,再和平鳍鳅科聚在一起。这种差异可能是由于使用的基因长度差异造成的,长度越长,信息量越大,所反映的系统发育结果可能更加接近真实情况。
鳅超科;ND4/ND5基因;序列分析;系统发育
鲤形目是世界上最大的淡水鱼类类群,约有3 268种(Nelson,2006),在经济发展和科学研究中占有很重要的地位。传统的鲤形目鱼类研究多以形态特征为基础,其分类及系统发育关系还存在较多争议,为此,美国自然科学基金资助了“鲤形目生命树(Cypriniformes Tree of Life, CToL)”的国际合作项目,希望联合世界各国的鱼类学研究者通过综合鱼类化石、形态特征及分子等各个方面的数据资料对鲤形目鱼类的系统发育问题进行全面的研究。
鳅超科(Cobitoidea)是鲤形目中较为重要的一个类群,其物种数约占整个鲤形目的26%(Nelson, 2006)。关于鳅超科的分类,Sawada(1982)根据48个种及亚种的52个骨骼及外部性状特征,把鳅超科分为鳅科(Cobtidae)和平鳍鳅科(Balitoridae)两个科,其中鳅科包括沙鳅亚科(Botiinae)、花鳅亚科(Cobitinae),平鳍鳅科包括条鳅亚科(Nemacheilinae)和平鳍鳅亚科(Balitorinae)。而国内学者则大多数将条鳅亚科归为鳅科(Chen & Zhu,1984;Zhu,1995;各地方鱼类志等)。目前国际上广为接受的是Sawada(1982)的观点(Nelson,2006;Saitoh et al,2006)。近年来,一些学者基于形态和分子特征对鳅超科鱼类的系统发育关系进行了探讨,发现新的结果不同于以往的任何研究(Nalbant & Bianco,1998;Saitoh et al,2006;Tang et al,2006;Šlechtová et al,2007),沙鳅亚科是上述4个类群中最原始的类群,其他3个类群的关系不明朗。因此,鳅超科鱼类的系统发育关系还有待于进一步的分析。
ND4和ND5是线粒体基因组中编码NADH脱氢酶亚基4和亚基5的两个蛋白质编码基因。有研究显示,与其他线粒体基因相比,ND4、ND5基因进化速率适中,在重建亲缘关系较远类群间的系统发育关系时,能构建出更加可靠的系统发育树(Zardoya & Meyer,1996)。目前,将这两个基因联合运用于系统发育关系研究的报道还很少。就同一研究类群而言,与短片段序列数据集相比,长片段序列数据集具有更多的数据信息,能够构建出更可靠的系统发育树(Hofreiter,2008)。线粒体ND4、ND5基因及两个基因间的3个tRNA基因的联合序列(以ND4+5来表示)与应用广泛的cytb基因相比,具有更多的核苷酸位点,信息量相对大很多。本研究以鳅超科鱼类为研究对象,对其ND4+5基因序列的变异情况进行分析,重建鳅超科鱼类的分子系统树,探讨ND4+5基因序列中包含的系统发育信息,同时讨论鳅超科的系统发育问题。
1 材料与方法
1.1 研究用标本
本研究以斑马鱼(Danio rerio)和鲤鱼(Cyprinus carpio)作外类群,选择了鳅超科鱼类五个类群共23种25个个体进行序列分析,构建鳅超科鱼类的分子系统树。其中鳅超科10个物种的ND4+5基因为新测序列,其他15种鳅超科鱼类及外类群的同源序列从GenBank下载获得。DNA测序分析采用的样品均为95%的酒精固定标本,主要从国内各水系收集获得,标本保存于中国科学院水生生物研究所淡水鱼类博物馆。具体种类及其他详细信息见表1。
1.2 基因组DNA的提取
在本研究中,采用了两种提取DNA的方法,一种为常规的酚−氯仿抽提法;另一种为高盐抽提法。方法如下;
酚−氯仿抽提法,取鱼背部肌肉适量于1.5 mL的Eppendorf管中,用蒸馏水、TE(10 mmol/L Tris.HCl,1 mmol/L EDTA,pH8.0)缓冲液浸泡,直到组织中无酒精味。然后加入620 µL的STE(0.1 mol/L NaCl,10 mmol/L Tris-HCl,1 mmol/L EDTA,pH8.0)细胞裂解液,70 µL 10%的SDS,10 µL蛋白酶K(10 µg/µL),置于55℃的恒温箱中消化至透明。加入等体积的平衡酚抽提两次(pH值),再加入等体积的氯仿−异戊醇(24∶1)抽提,至无蛋白质。加入上清液两倍体积的无水乙醇沉淀2 h以上,离心后再用75%的乙醇洗涤沉淀,再离心去乙醇干燥后加入30~50 µL的灭菌双蒸水或TE缓冲液溶解,并存于−20℃的冰箱中备用。
高盐抽提法,取鱼背部肌肉适量于2 mL的Eppendorf管中,剪碎烘干,加入500 µL HOM Buffer和10~15 µL蛋白酶K,置于55℃的恒温箱中消化至透明。加入500 µL NaCl(4.5 mol/L),300 µL氯仿离心。加入600 µL异丙醇沉淀,于−20℃保存1 h以上,离心后再用70%乙醇洗涤,干燥后加入50 µL灭菌双蒸水溶解,存于−20℃的冰箱中备用。
1.3 PCR扩增
ND4+5基因序列全长3 387~3 443 bp。在mtDNA上,ND4基因左侧是ND4L基因,ND5基因右侧是ND6基因,ND4和ND5基因之间是3个tRNA基因。基于此结构,分别在ND4L、ND6基因的侧翼区及ND4、ND5基因内部设计引物。引物序列参考Miya et al(2006),并稍作改动。用于扩增的引物共5对:LArgd1+H11618d;L11427dA+H12632dB;L12328d+H13393d;L13058d+H13721d;L13559d+H14710d(图1)。
PCR反应的总体积为60 µL,反应体系包括:10×Buffer 6 µL,dNTPs 1.2 µL(每个碱基浓度为2.5 mmol/L),引物各1.5 µL(10 µmol/L),Taq酶3.0 U。PCR反应条件为:95℃预变性3 min,然后循环包括:94℃变性30 s,52℃退火30 s,72℃延伸1 min,共35个循环,最后再72℃延伸5 min。
1.4 PCR产物的纯化和测序
PCR产物用1.0%的琼脂糖电泳检测,并在紫外投射仪上观察。PCR产物用试剂盒进行割胶回收或直接过柱回收。回收产物送测序公司测序。
1.5 数据分析

表 1 研究中所用标本及来源Tab. 1 Specimens and their localities in the present study
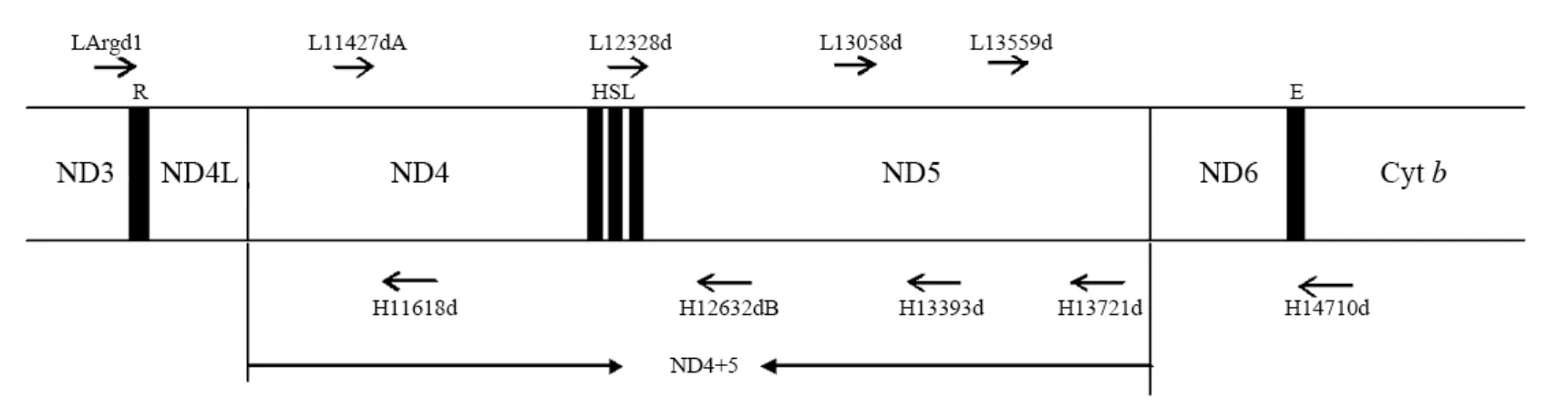
图 1 ND4、ND5基因位置图解及5对引物的相对位置Fig. 1 Schematic representation of the ND4/ND5 gene region and relative positions of 5 pairs of primers
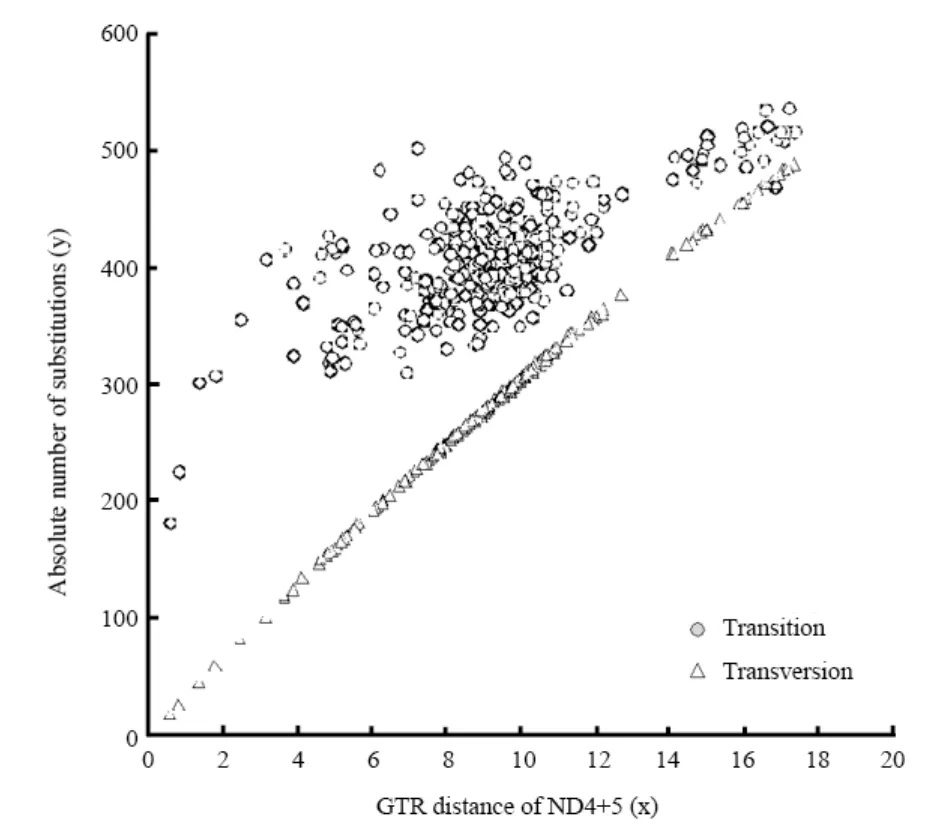
图 2 转换、颠换的绝对数目和基于GTR模型的本研究中所有个体ND4+5基因序列成对比较所得的遗传距离散点图Fig. 2 Scatter plot for the absolute number of transitions and transversions versus GTR distance of ND4+5 gene in pairwise comparisons between the species in the present study
DNA序列的排列使用ClustalX软件,并进行手工调整。从GenBank下载鳅超科鱼类和外类群鱼类的ND4+5基因序列。通过比对分析找出序列的起始密码子(ATG)和终止密码子,删除起始密码子前和终止密码子后的碱基。序列中各碱基的含量及变异情况用Mega4.0(Tamura et al,2007)中的Statistic命令进行分析。用PAUP*软件统计出序列中转换和颠换的绝对数目以及序列的变异情况(用GTR模型下的遗传距离,GTR distances),并在Statistica 6.0软件中进行作图分析序列中碱基替代的饱和程度。分子系统树的构建采用最大简约法(MP)、最大似然法(ML)和贝叶斯推断法(BI),其中MP和ML法采用PAUP*4.0b10(Swofford,2002)软件,BI法采用MrBayesV3.0(Ronquist & Huelsenbeck,2003)软件。MP和ML法各分支的置信度分别采用1 000次和100次自展分析(bootstrap analysis)进行重复检验。BI法以后验概率(posterior probability)来表示各分支的可信性。
2 结 果
2.1 鳅超科鱼类ND4+5基因序列与遗传变异分析
比对分析的结果表明,鳅超科鱼类ND4基因全长1 380~1 38 bp,以ATG为起始密码子,终止密码子为不完全终止信号;ND5基因全长1 821~1 839 bp,同样起始密码子为ATG,终止密码子为TAA或TAG;ND4和ND5基因之间插入了3个tRNA基因,分别编码携带组氨酸、丝氨酸和亮氨酸的tRNA,序列长度分别为70、68和73 bp。鳅超科鱼类ND4+5基因序列全长3 387~3 443 bp,序列中A、T、G、C的平均含量分别为30.4%、27.3%、14.2%、28.1%,A+T (57.7%)的含量高于G+C (42.3%)的含量。
基于Kimura双参数模型计算遗传距离,以确定类群间的遗传分化程度,结果显示类群间的遗传距离值为0.203~0.255。鳅超科ND4+5基因序列转换与颠换之比(Ti/Tv)的变异范围为0.983~9.579,平均值为1.586。 在供分析的3 462个位点中,共有1 899个变异位点,其中简约信息位点为1 525个。图1显示所有个体ND4+5基因序列的转换和颠换均未达到饱和,说明序列中的替代均具有系统发育意义,可用于数据分析。在所有的变异水平上,转换数明显大于颠换数。
2.2 分子系统树
基于目前的数据,我们分析了鳅超科鱼类5个不同的类群:条鳅亚科,花鳅亚科,沙鳅亚科,平鳍鳅科和Vaillantellidae的系统发育关系。其中Vaillantellidae只包含一个属Vaillantella,该属只分布于印尼及马来西亚一带(fishbase 2009年2月更新;网址http://www.fishbase.org/search.php)。使用最大简约法(MP)、最大似然法(ML)和贝叶斯推断法(BI)构建分子系统树。3种方法构建的分子系统发育树的拓扑结构基本一致(图3~5)。
以斑马鱼(Danio rerio)和鲤鱼(Cyprinus carpio)作为外类群,3种构树方法的结果一致显示,鳅超科的系统发育关系为:(Vaillantellidae+(沙鳅亚科+(花鳅亚科+(条鳅亚科+平鳍鳅科);条鳅亚科、平鳍鳅科、花鳅亚科、沙鳅亚科和Vaillantellidae这5个类群都各自独立形成单系,且都得到较高的支持率(均超过90;图3~5);Vaillantella处于所有鳅类的基部位置,成为一个独立的分支,其支持率较高(MP树中为78,ML树中为76,BI树中为100)。条鳅亚科和平鳍鳅科聚在一起构成一个分支(lineage),支持率均在50以上:MP树中为自展支持率(bootstrap value, BP)为50, ML树中BP为68,BI树中后验概率(posterior probability,PP)为94。该分支与花鳅亚科构成姐妹群,然后再与沙鳅亚科一起构成一个大的分支群。至于每个类群内部物种间的系统发育关系,MP树(图3)、ML树(图4)和BI树(图5)的拓扑结构几乎一致,只在部分类群(花鳅亚科中花鳅属的三个物种及条鳅亚科中的4个物种)的分支顺序上存在差异。
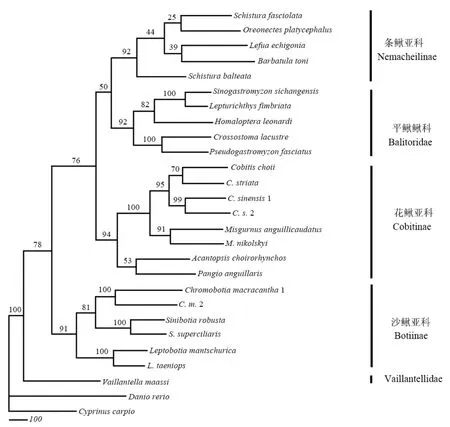
图 3 基于ND4+5基因序列构建的鳅超科鱼类的MP树Fig. 3 MP tree of Cobitoidea based on the ND4+5 gene sequences

图 4 基于ND4+5基因序列构建的鳅超科鱼类的ML树Fig. 4 ML tree of Cobitoidea based on the ND4+5 gene sequences
平鳍鳅科明显分为两个支系:平鳍鳅亚科(Balitorinae)和腹吸鳅亚科(Gastromyzoninae),它们分别构成单系群。腹吸鳅亚科构成单系的支持率在3种构树方法中都为100%,平鳍鳅亚科的支持率分别为82%(MP树)、68%(ML树)和98%(BI树)。条鳅亚科中,两个南鳅属(Schistura)的物种没有聚在一起。在M P树中,横纹南鳅(Schisturafasciolata)与平头平鳅(Oreonectes platycephalus)聚在一起。而在MP树和BI树中,横纹南鳅(Schistura fasciolata)与Barbatula toni聚在一起。花鳅亚科中,花鳅属(Cobitis)和泥鳅属(Misgurnus)的物种都能分别构成单系,且支持率很高(图3~5)。比较不同构树方法的结果发现,ML树和BI树中花鳅属各物种的聚类顺序跟MP树中的有所不同。沙鳅亚科在本研究中只包括两个属:沙鳅属(Botia)和薄鳅属(Leptobotia)(Chen,1980;Nelson,1994;Tang et al,2008),这两个属在3种树中各自构成单系,均得到较高的支持率。
3 讨 论
3.1 ND4和ND5基因的特点及在系统发育分析中的应用
mtDNA不同区段基因序列的进化速度不一样(Li & Shao, 2006)。因此,选取mtDNA上的不同基因或片段序列,可以进行不同分类水平上的进化研究。对于亲缘关系较近的物种,一般选取选择压力小、进化较快的基因序列;对于亲缘关系较远的物种,则选取选择压力大、比较保守的基因序列。
NADH作为线粒体呼吸链上的一种功能蛋白,它的基因序列一般很少被用来研究系统发育关系。但从本研究的结果来看,适合解决科级水平的系统发育关系,因而在进行同一个目内科间水平系统发育分析时,ND4和ND5基因也不乏为一个好的分子标记。
另外,在重建高阶元的系统发育关系时,序列长度是一个非常需要考虑的问题。约3 kb长的序列被认为是一个较为理想的长度(Miya et al,2006)。ND4+5基因全长3 387~3 443 bp,与cytb基因(1 140 bp)相比,包含更多的系统发育信息,将该标记用于分子系统发育分析,可能会得到更为准确的系统发育关系。

图 5 基于ND4+5 基因序列构建的鳅超科鱼类的贝叶斯树Fig. 5 Bayesian tree of Cobitoidea based on the ND4+5 gene sequences
就ND4和ND5基因而言,这两个基因单独用于系统发育关系的研究已有报道。Zhang et al(1999)分析12种的鲟形目鱼类的ND4基因的序列变异,推测了部分鲟鱼的亲缘关系。 Mou et al(2005)以ND4L和ND4基因为标记研究了黑腹果蝇的系统发育关系。Zhang et al(2006)对ND5基因进行克隆和序列分析,进一步确定了大黄鱼的分类地位。Makita et al(2000)将ND5应用于虎凤蝶属(Luehdorfia)的种间研究,结果显示,虎凤蝶属的种和外群蝴蝶种间ND5基因差异较大,同属内不同物种间ND5基因的差异相对要小。
本研究将ND4和ND5基因及两基因间的tRNA基因序列联合起来,对鳅超科鱼类的系统发育关系进行分析,得到的系统发育关系与线粒体基因组、核基因及形态学研究的结果较为一致,表明ND4+5基因序列应用于科之间系统发育关系的重建是可行的。至于该片段能否用于属间或种间系统发育关系分析还有待进一步研究。ND4+5基因易于扩增,通过5对引物联合扩增测序可获得其全序列。因此,对一些保存不太好的样本,无法扩增其核内基因,ND4+5基因可以作为一个很好的备选分子标记。
3.2 鳅超科鱼类的系统发育关系
鳅超科中关于鳅科和平鳍鳅科鱼类的系统发育关系问题,一致存在争议。Sawada(1982)和Siebert(1987)基于外部形态和骨骼特征的分析结果认为,鳅科只含两个亚科:花鳅亚科和沙鳅亚科,他们认为条鳅亚科与平鳍鳅科的成员有较近的亲缘关系,条鳅亚科应归为平鳍鳅科。Nalbant和Bianco(1998)认为Sawada(1982)研究中所用的骨骼性状,在条鳅亚科和平鳍鳅科中较为相似是因为两者趋同进化形成的。因此,Nalbant和Bianco(1998)将条鳅亚科作为一个独立的科对待。后来分子水平上的研究也认为条鳅亚科和平鳍鳅科均存在较大的差异,也支持将条鳅亚科独立为一个科;Sawada(1982)的鳅科(包括花鳅亚科和沙鳅亚科)并不构成单系。基于这两点,花鳅亚科和沙鳅亚科被认为应提升到科的水平(Nablbant & Bianco,1998;Tang et al,2006;Šlechtová et al,2007)。在系统发育关系上,Tang et al(2006)基于线粒体cytb和Dloop基因的分析结果认为,条鳅类和花鳅类构成姐妹群,再和平鳍鳅类聚在一起,沙鳅类位于更基部的位置;而核基因(Šlechtová et al,2007)及线粒体全基因组(Saitoh et al,2006)的结果却显示,条鳅类和平鳍鳅类首先聚在一起,再和花鳅类构成姐妹群,沙鳅类位于更基部的位置。
本研究以线粒体ND4+5基因作为分子标记所得出的结果与核基因(Šlechtová et al,2007)及线粒体全基因组(Saitoh et al,2006)作为分子标记所得出的结果一致,即条鳅类和平鳍鳅类构成姐妹群,再和花鳅类聚在一起,它们构成的大分支再和沙鳅类聚在一起,Vaillantellidae处于所有鳅类的基部位置。本研究的结果与基于线粒体cytb和Dloop基因所得出的条鳅类和花鳅类亲缘关系更近的结论不一致,这种差异可能是由于所使用的基因的长度差异造成的,长度越长,信息量越大,所反映的系统发育关系可能更加接近真实情况。
本研究中,Vaillantella处于所有鳅类的基部位置,成为一个独立的分支,其支持率较高(MP树中为78,ML树中为76,BI树中为100)。曾有研究对Vaillantella的系统发育位置持不同的观点。Sawada(1982)和Siebert(1987)基于形态数据分析研究认为,Vaillantella被分别归为两个不同科的物种。有人提出将这个属划分到一个独立的科Vaillantellidae(Nalbant & Bǎnǎrescu,1977;Šlechtová et al,2007)。我们的研究支持将其独立为一个科。
本研究中分子系统树显示:条鳅亚科中两个南鳅属(Schistura)的物种没有聚类在一起。在MP树中,横纹南鳅(Schistura fasciolata)与平鳅属(Oreonectes)的平头平鳅(Oreonectes platycephalus)聚在一起。而在MP树和BI树中,横纹南鳅(Schistura fasciolata)与须鳅属(Barbatula)的Barbatula toni聚在一起。由此推断,南鳅属可能不是一个单系,这与Bănărescu和Nalbant(1995)及Tang et al(2006)的研究结果是一致的,其对应的系统学问题需要进行进一步的研究。
总之,ND4+5基因作为一个优秀的分子标记,具有3 400 bp左右的序列长度,包含较多的系统发育信息。该基因序列不仅容易进行扩增、测序,而且能构建出可信度较高的系统发育树。本研究的结果进一步澄清了鳅超科鱼类之间系统发育关系的疑问,从分子水平上支持条鳅亚科与平鳍鳅科具有更近的亲缘关系,从而表明该基因应用于科之间系统发育关系的重建是可行并可信的。
Bănărescu PM, Nalbant TT. 1995. A generical classification of Nemacheilinae with description of two new genera (Teleostei: Cypriniformes: Cobitidae) [J].Trav Mus Host Nat “Grigore Antipa”, 35:429-496.
Chen JX. 1980. A study on the classification of the Botoid fishes of China [J].Zool Res, 1(1): 3-25. [陈景星. 1980. 中国沙鳅亚科鱼类系统分类的研究. 动物学研究, 1(1): 3-25.]
Chen JX, Zhu SQ. 1984. Phylogenetic relationships of the subfamily in the loach family Cobitidae (Pisces) [J].Acta Zootax Sin, 9(2): 201-207. Hofreiter M. 2008. Long DNA sequences and large data sets: investigating the Quaternary via ancient DNA [J].Quat Sci Rev, 27(27): 2586-2592. [陈景星, 朱松泉. 1984. 鳅科鱼类亚科的划分及其宗系发生的相互关系. 动物分类学报, 9(2): 201-207.]
Li YD, Shao JH. 2006. A study on whole mitogenome sequences and molecular phylogenetic relationships of animals [J].J Qinghai Med Coll, 27 (1): 68-70. [李耀东, 邵嘉会. 2006. 线粒体基因组全序列研究与动物分子系统发生的关系. 青海医学院学报, 27 (1): 68-70.]
Makita H, Shinkawa T, Ohta K, Kondo A. 2000. Phylogeny of Luehdorfia butterflies inferred from mitochondrial ND5 gene sequences [J].Entom Sci, 3(2): 321-329.
Miya M, Saitoh K, Wood R, Nishida M. 2006. New primers for amplifying and sequencing the mitochondrial ND4/ND5 gene region of the Cypriniformes (Actinopterygii: Ostariophysi) [J].Ichthy Res, 53(1): 75-81.
Mou SL, Zeng QT, Yang Y, Qian YH, Hu GA. 2005. Phylogeny of melanogaster species group inferred from ND4L and ND4 genes [J].Zool Res, 26(4): 344–349. [牟少亮, 曾庆韬, 杨 勇, 钱远槐. 2005.以ND4L和ND4基因为标记探讨黑腹果蝇种组的系统发育关系.动物学研究, 26(4): 344-349.]
Nalbant TT, Bǎnǎrescu P. 1977. Vaillantellinae, a new sub-family of Cobitidae (Pisces, Cypriniformes) [J].Zool Mededelingen, 52: 99-105.
Nalbant TT, Bianco PG. 1998. The loaches of Iran and adjacent region with description of six new species (Cobitoidea) [J].Ital J Zool, 65: 109-123.
Nelson JS. 1994. Fishes of the World[M]. 3rd ed. New York: John Wiley and Sons.
Nelson JS. 2006. Fisher of the World[M]. 4th ed. Hoboken: John Wiley and Sons.
Ronquist F, Huelsenbeck JP. 2003. MRBAYES 3: Bayesian phylogenetic inference under mixed models [J].Bioinformatics, 19: 1572-1574.
Saitoh K, Sado T, Mayden RL, Hanzawa N. 2006. Mitogenomic evolution and interrelationships of the Cypriniformes (Actinopterygii: Ostariophysi): The first evidence toward resolution of higher-level relationships of the world’s largest freshwater fish clade based on 59 whole mitogenome sequences [J].J Mol Evol, 63(6): 826-841.
Sawada Y. 1982. Phylogeny and zoogeography of the superfamily Cobitoidea (Cyprinoidei, Cyprinionfrmes) [J].Mem Fac Fisher:Hokkaido University, 28(2): 65-223.
Siebert DJ. 1987. Interrelationships Among Families of the Order Cypriniformes (Teleostei)[M]. New York: City University of New York.
Šlechtová V, Bohlen J, Tan HH. 2007. Families of Cobitoidea (Teleostei: Cypriniformes) as revealed from nuclear genetic data and the position of the mysterious generaBarbucca,Psilorhynchus,SerpenticobitisandVaillantella[J].Mol Phylogenet Evol, 44: 1358-1365.
Swofford DL. 2002. PAUP*: Phylogenetic Analysis Using Parsimony (*and other Methods). Version 4. [M].Sunderland, Massachusetts: Sinauer Associates.
Tamura K, Dudley J, Nei M, Kumar S. 2007. MEGA4: Molecular Evolutionary Genetics Analysis(MEGA)software version 4.0 [J].Mol Biol Evol, 24: 1596-1599.
Tang QY, Liu HZ, Mayden R, Xiong B. 2006. Comparison of evolutionary rates in the mitochondrial DNA cytochromebgene and control region and their implications for phylogeny of the Cobitoidea (Teleostei: Cypriniformes) [J].Mol Phylogenet Evol, 9: 347-357.
Tang QY, Yu D, Liu HZ. 2008.Leptobotia zebrashould be revised asSinibotia zebra(Cypriniformes: Botiidae) [J].Zool Res, 29(1): 1-9. [唐琼英, 俞 丹, 刘焕章. 2008. 斑纹薄鳅(Leptobotia zabra)应该为斑纹沙鳅(Sinibotia zabra). 动物学研究, 29(1): 1-9.]
Zardoya R, Meyer A. 1996. Phylogenetic performance of mitochondrial protein coding genes in resolving relationships among vertebrates [J].Mol Biol Evol, 13: 933-942.
Zhang SM, Zhang YP, Zheng XZ, Chen YJ, Deng Huai,Wang DQ,Wei QW,Zhang YW,Nie L,Wu QJ. 1999. The variation and the molecular systematics of 12 genera (Acipenseriformes) based on mt DNAND4L-ND4gene [J].Sci ChnSeries C:Life Sci. 29(6): 607-614. [张四明, 张亚平, 郑向忠, 陈永久, 邓怀, 汪登强, 危起伟, 张云武,聂龙, 吴清江. 1999. 12种鲟形目鱼类mtDNAND4L-ND4基因的序列变异及其分子系统学. 中国科学 C辑:生命科学, 29(6): 607-614.]
Zhang ZX, Li MY, Zhu JJ. 2006. Cloning and sequence analysis of mtDNA ND5 and cytbgene of large yellow croakerPseudosciaena crocea[J].Fish Sci, 25(12): 626-631. [张祖兴, 李明云, 朱俊杰. 2006. 大黄鱼mtDNA ND5和Cytb基因的克隆与序列分析. 水产科学, 25(12): 626-631.]
Zhu SQ. 1995. Synopsis of Freshwater Fishes of China [M]. Nanjing:Jiangsu Science and Technology Publishing House. [朱松泉. 1995. 中国淡水鱼类检索. 南京: 江苏科技出版社.]
Phylogenetic Relationships Among Cobitoidea Based on Mitochondrial ND4 and ND5 Gene Sequences
LIU Si-Qing1,2, ZHANG Jia-Bo1, TANG Qiong-Ying2, LIU Huan-Zhang2,*
(1.Huazhong Agricultural University, Wuhan430070,China; 2.Institute of Hydrobiology, the Chinese Academy of Sciences, Wuhan430072,China)
In the present study, we cloned and sequenced 10 new ND4 and ND5 gene sequences of Cobitoidea. These sequences were used to reconstruct phylogenetic relationships together with those of 15 other species downloaded from GenBank. The results showed that the length of ND4 gene sequence was 1 380−1 387 bp with ATG as starting codon and incomplete termination signal as terminated codon; the length of ND5 gene sequence was 1 821−1 839 bp with ATG as starting codon and TAA or TAG as terminated codon; three tRNA genes coding tRNAs that carry hisidine, serine and leucine respectively, were inserted between ND4 and ND5 genes. A, T, C and G accounted for 30.4%, 27.3%, 14.2% and 28.1% in ND4/ND5 gene (including intervening three tRNA genes). The content of A+T (57.7%) is higher than that of G+C (42.3%). The estimated Ti/Tv ratio was 1.586. WithDanio rerioandCyprinus carpioas outgroups, the phylogenetic relationships of Cobitoidea were analyzed using maximum parsimony (MP) method, maximum likelihood (ML) method and Bayesian analyses (BI). Results of all the three methods indicated that Cobitinae, Nemacheilinae, Botiinae, Balitoridae and Vaillantellidae were all monophyletic respectively, and their interrelationships were: (Vaillantellidae + (Botiinae + (Cobitinae + (Nemacheilinae + Balitoridae)))), which was consistent with the previous studies based on the whole mitogenome and some nuclear genes sequences (eg. RAG1 gene). However, the present results differ from those based upon other mtDNA genes sequences (e.g. the cytband D-loop showed that Nemacheilinae grouped with Cobitinaeforming sister-group, then they clustered with Balitoridae). The difference could be due to the phylogenetic information carried by different length sequence: Longer sequences carry more information and result in more reliable phylogenetic trees.
Cobitoidea; ND4/ND5 gene; Sequence analysis; Phylogeny
Q959.468; Q349; Q951.3
A
0254-5853-(2010)03-0221-09
10.3724/SP.J.1141.2010.03221
2009-06-02;接受日期:2009-10-30
国家自然科学基金资助项目(40432003;30700072);美国自然科学基金资助项目CToL
*通讯作者 (Corresponding author),Tel: 027-68780776,E-mail: hzliu@ihb.ac.cn
刘思情,女,硕士研究生
