SARS 3CL蛋白酶同源二聚化与底物结合互为别构调控因素
2010-11-30李春梅来鲁华
魏 平 李春梅 周 璐 刘 莹 来鲁华,*
(1北京大学化学与分子工程学院,分子动态与稳态国家重点实验室,北京分子科学国家实验室,北京 100871; 2北京大学理论生物学中心,北京 100871)
SARS 3CL蛋白酶同源二聚化与底物结合互为别构调控因素
魏 平1,2李春梅1,2周 璐1刘 莹1来鲁华1,2,*
(1北京大学化学与分子工程学院,分子动态与稳态国家重点实验室,北京分子科学国家实验室,北京 100871;2北京大学理论生物学中心,北京 100871)
研究了严重急性呼吸系统综合症(SARS)冠状病毒3C-Like蛋白酶(3CLpro)在存在底物或抑制剂时的二聚体形成情况.通过测定酶活性随酶浓度的变化,拟合出在底物存在下酶二聚体的解离常数约为0.94 μmol·L-1,小于纯蛋白酶的二聚体解离常数(14.0 μmol·L-1),表明底物对二聚体的形成具有增强作用.选用与底物具有类似结合方式的靛红类抑制剂N-萘甲基靛红-5-甲酰胺(5f),利用超速离心沉降速率方法定量测定了SARS 3CL蛋白酶单体和二聚体在不同浓度5f时的含量,发现5f同样具有诱导二聚体形成的能力.在3 μmol·L-1蛋白酶浓度下测定得到诱导二聚的EC50值(半数有效浓度)约为1 μmol·L-1,说明二聚体中只有一个单体与抑制剂结合.研究结果表明,随着底物浓度的升高,SARS 3CL蛋白酶会形成更多的二聚体,而二聚体含量的提高又反过来提高酶的活性,这种双向别构调控机制有可能是病毒用来调控多聚蛋白水解速率和组装时机的一种方法.
别构效应;SARS;3CL蛋白酶;分析型超速离心;底物增强的酶二聚化
严重急性呼吸系统综合症(SARS)冠状病毒3CL蛋白酶是病毒复制周期中的关键酶.由于该蛋白酶在人类细胞中没有同源分子,针对其设计的药物不易产生毒副作用[1].同时由于其在病毒复制中的突变率低,能够避免病毒突变过快形成的抗药性,因此被普遍认为是基于结构的抗SARS及其它由冠状病毒引发疾病药物设计的重要靶标[2-3].SARS冠状病毒3CL蛋白酶与其他几种冠状病毒3CL蛋白酶的序列相似程度很高.其三维结构与另外两种已知的冠状病毒3CL蛋白酶晶体结构基本相同,都含有三个结构域[2,4].N端两个结构域构成了催化结构域,其结构类似于丝氨酸蛋白酶,起催化作用的氨基酸残基位于这两个结构域间形成的裂隙中[2,5-6].C端的结构域是一个以α-螺旋为主的结构域.研究表明冠状病毒3CL蛋白酶在溶液中能够形成二聚体,C结构域对于二聚体的形成至关重要[7-9].
在生物系统中,蛋白质极少以独立的角色参与生命活动,往往是通过与其它分子结合形成大分子复合物的方式来实现各种功能的,其中相当部分是相同蛋白质分子间所形成的同源二聚或多聚体.在Brenda酶数据库(http://www.brenda.uni-koeln.de)中452个列出亚基构成的人类蛋白酶中,仅有约1/3 (141个)以单体形式存在,其余的蛋白酶均以聚合体形式存在,这其中有近一半(125个)为同源二聚体[10].聚集体的形成可以带来很多好处.例如:聚集过程能够使蛋白质产生随浓度变化而改变聚集状态的调控机制,只有在达到一定酶浓度时,聚合体蛋白才能形成,从而实现特定的功能;多聚化能够赋予蛋白酶新的结合界面,催化中心,别构位点等;多聚蛋白酶具有比单体酶分子更强的目标结合能力[10].蛋白质这种聚集能力也可以节约生物体内有限的基因资源.
现已报导的病毒蛋白酶中有相当一部分是以二聚体形式存在的.而且研究发现,这些病毒蛋白酶只有在二聚体中才有活性[11-12].这其中最著名的例子是人类免疫缺陷病毒HIV-1蛋白酶,其活性口袋位于二聚体界面上,活性中心由两个亚基各提供一个Asp-25组成,所以只有二聚体形成之后才能有完整的活性中心[13].同样以二聚来调控活性的还有人类肝炎病毒HCV蛋白酶,不同的是HCV蛋白酶的两个亚基互相提供活性中心残基,分别组成对称的两套催化中心[14].晶体结构研究显示冠状病毒3CL蛋白酶二聚体中的两个亚基分别拥有自己独立的活性位点,且远离其二聚体界面.我们的研究发现,SARS 3CL蛋白酶同样也需要形成二聚体才具有蛋白酶活性[15].SARS 3CL蛋白酶二聚体形成的原因,二聚体对于酶催化活性的调控作用等是非常有趣的问题.本研究小组在SARS爆发伊始,就运用理论计算与实验结合的方法研究了SARS 3CL蛋白酶的生物化学性质[16-18],酶催化反应机理[15,19-20],并开展了基于结构的抑制剂设计[21-22].
针对SARS 3CL蛋白酶二聚体的计算分析表明其二聚体的结合能力并不是很强[17],我们利用分析型超速离心方法,定量测出了纯SARS 3CL蛋白酶二聚体的解离常数为14.0 μmol·L-1[18].以此推测,在常用的体外SARS 3CL蛋白酶测活条件下(酶的浓度一般为1-3 μmol·L-1),二聚体的含量很低,这似乎与必须形成二聚体才能有活性的观点有所矛盾[15].为了解释此现象,我们通过测定酶活性随酶浓度的变化,推算出在底物存在下二聚体的表观解离常数明显变小,表明在测活条件下二聚体的含量有了显著提高.
为了证实以上推测,我们选取了一个本实验室在前期报导的与底物有类似结合的抑制剂分子N-萘甲基靛红-5-甲酰胺(5f)[22],采用分析型超速离心实验定量测定了在不同抑制剂浓度下二聚体含量的变化情况.分析型超速离心方法能够在天然溶液条件下有效分析生物大分子的相对分子质量分布情况[23-24].沉降速率实验能够直观灵敏地分析溶液中的大分子聚集状态的分布,而沉降平衡实验是目前最精确的测定大分子间同源或异源聚集体解离常数的手段之一[25-26].本文通过研究抑制剂5f对溶液中蛋白酶二聚体形成能力的影响,来分析说明分析型超速离心测得的纯SARS 3CL蛋白酶的二聚体与由蛋白酶活性随浓度变化研究拟合出的二聚体解离常数不一致的原因,探讨可能的SARS 3CL蛋白酶二聚化相互别构调控的机制.
1 材料与方法
1.1 蛋白酶的表达与纯化
将含有SARS 3CL蛋白酶表达质粒pET3CLpro-21h的大肠杆菌BL21〈DE3〉菌培养到对数生长期,向培养液中加入终浓度1 mmol·L-1的IPTG(isopropyl-1-thio-β-D-galactopyranoside),在 30℃下诱导表达3 h后,离心收集菌泥.用20 mL裂解液(40 mmol·L-1NaH2PO4/Na2HPO4,pH 7.3,100 mmol·L-1NaCl,1 mmol·L-1EDTA(乙二胺四乙酸),3 mmol·L-1DTT(硫代苏糖醇))重悬1 L培养液得到的菌泥,超声裂解细胞.16000 r·min-1转速离心,收集上清液.向上清液中加入终浓度100 g·L-1的硫酸铵,充分搅拌之后以16000 r·min-1转速离心,收集上清液.向上清液中继续加入硫酸铵至终浓度160 g·L-1,同样充分搅拌之后,再次以16000 r·min-1转速离心,收集沉淀.将沉淀溶于20 mL缓冲液A(20 mmol·L-1Tris-HCl,pH 8.5,1 mmol·L-1EDTA,1 mmol·L-1DTT),4℃下在1 L缓冲液A中透析充分.透析得到样品经阴离子交换色谱(HiTrap Q HP柱)分离纯化,收集到的样品经过适当浓缩后,通过凝胶过滤色谱(Sephacryl S-200 HR柱)再次纯化,得到的蛋白酶样品经SDS-PAGE电泳证实为95%以上的纯度.得到的产品保存在缓冲液C(40 mmol·L-1NaH2PO4/ Na2HPO4,pH 7.3,100 mmol·L-1NaCl,1 mmol·L-1EDTA,3 mmol·L-1DTT)中,并加入50%(φ,体积分数)的甘油,-20℃保存备用.
1.2 蛋白酶的活性测定
本文所述所有蛋白酶活性测定实验均使用本实验室黄常康博士等建立的显色底物活性测定方法[18].所采用的底物为SARS 3CL蛋白酶自切位点P1-P6序列,将自切位点P′位Ser用显色基团pNA代替(Thr-Ser-Ala-Val-Leu-Gln-pNA),酶催化水解Gln-pNA键,释放出显色化合物pNA,在390 nm波长下检测.pNA在体系中的吸光系数为6.33 OD· mmol-1·L-1.测活反应在37℃下进行.所使用的酶标仪为SpectraMax GeminiXS(Molecular Device).底物由上海吉尔生化有限公司提供.
1.3 蛋白酶聚集状态的测定
沉降速率实验在Beckman Optima XLA分析型超速离心机上进行,使用An60Ti转头和双孔样品池进行实验.新鲜纯化的高纯度样品经脱盐柱或透析处理后,使样品处于沉降缓冲液(40 mmol·L-1NaH2PO4/Na2HPO4,100 mmol·L-1NaCl,0.5 mmol·L-1EDTA,pH 7.3)中,其溶液环境与参照缓冲液完全一致.测得样品在沉降实验检测波长230 nm下的吸收值,配置样品浓度在该波长下吸收值的范围为0.5-1.0.不含抑制剂的对照组为3 μmol·L-1的SARS 3CL蛋白酶,蛋白溶液及参照液中加入相应量的不含抑制剂的纯DMSO;含有抑制剂的样品组,3 μmol·L-1的蛋白溶液与参照缓冲液中均加入浓度为3 mmol·L-1的抑制剂5f的DMSO(二甲基亚砜)储液.双孔样品池中,分别加入380 μL蛋白酶样品和400 μL参照缓冲液.样品池在转子中20℃,3000 r·min-1转速下平衡1 h后,设定检测波长为230 nm,在56000 r·min-1转速下收集沉降速率数据.实验数据用软件Sedfit v9.4(http://www.analyticalul-tracentrifugation.com/)分析处理.数据分析所需要的参数, SARS 3CL蛋白酶在280 nm波长下的分子消光系数(1.04 mg·cm-2),沉降缓冲液的密度(1.005 g·mL-1), SARS 3CL蛋白酶的偏摩尔体积(0.723 mL·g-1),以及蛋白酶的分子量(33914 Da)均根据该蛋白酶的氨基酸组成通过软 SENDNTERP(http://www.bbri.org/ rasmb/rasmb.html)计算得到.
2 结果与讨论
2.1 底物增强的SARS 3CL蛋白酶二聚
为了研究底物对于SARS 3CL蛋白酶二聚化的影响,我们测定了在不同蛋白浓度下(0.14-1.65 μmol·L-1)酶的活性.如果二聚体酶是酶的活性状态,那么体系中蛋白酶二聚体的含量会随着酶浓度的提高而增加.这意味着,单位酶的活性也会随着酶浓度的提高而增加,而这一值在一般蛋白酶中是不变的.我们假设蛋白酶的总浓度为CE,在溶液中存在着单体M和二聚体酶M2的平衡,二聚体的解离常数为Kd,单体和二聚体的组分都有结合底物S的能力,则有:

假定在测活条件下酶与底物结合形成的复合物浓度可以忽略不计,
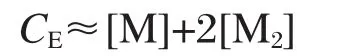
由此可以推出:


由酶活性实验可以推出蛋白酶的表观二级反应速率常数(kcat/Km)app.相关的研究表明,SARS 3CL蛋白酶的活性主要是由二聚体所贡献的[3,7-9,15-18],因此我们直接假设单体对于活性的贡献为零,定义酶的表观反应速率为

由此推出:

将酶的表观二级反应速率常数(kcat/Km)app对蛋白酶浓度CE作图(图1),由公式(1)拟合得到结果为
二聚体活性(kcat/Km)dimer=(35.8±1.1)mmol-1·L· min-1.
二聚体解离常数Kd=(0.94±0.08)μmol·L-1.
2.2 抑制剂诱导二聚体形成
我们在上节中通过测定酶活性随酶浓度变化的方法推算出在测活条件下二聚体的解离常数为0.94 μmol·L-1,与用分析型超速离心方法测定的纯蛋白酶的解离常数(14.0 μmol·L-1)有明显的差异[18].这可能是由于底物的存在提高了酶二聚体的结合能力.其它病毒蛋白酶(如HIV蛋白酶)中也存在着这种底物或抑制剂诱导二聚体形成的现象[27-28].为了更进一步研究SARS 3CL蛋白酶是否存在由底物或抑制剂诱导二聚的现象,我们选用了一个底物口袋结合抑制剂5f(图2),利用沉降速率实验研究了不同浓度的5f对于SARS 3CL蛋白酶聚集状态的影响.该抑制剂对浓度为1 μmol·L-1的SARS 3CL蛋白酶的半抑制浓度IC50为0.37 μmol·L-1[22].

图1 酶浓度依赖的活性曲线Fig.1 Concentration-dependent proteinase activity curve

图2 抑制剂5f的结构式Fig.2 Chemical structure of the inhibitor 5f
为了模拟测活反应的条件,使用较低浓度的蛋白酶(3 μmol·L-1),这一浓度条件下,蛋白酶几乎完全以单体形式存在[18].针对三组样品在同样条件下进行沉降速率分析,对照组仅有3 μmol·L-1的SARS 3CL蛋白酶;第二组含有3 μmol·L-1的蛋白酶和1 μmol·L-1抑制剂;第三组含有3 μmol·L-1的蛋白酶与6 μmol·L-1抑制剂.
如图3所示,对照组蛋白浓度为3 μmol·L-1的SARS 3CL蛋白酶在无抑制剂条件下沉降系数分布接近2.7 s,意味着溶液中的蛋白酶几乎均以单体形式存在[18];3 μmol·L-1的SARS 3CL蛋白酶溶液中加入1 μmol·L-1的抑制剂5f(摩尔比3∶1)后,沉降系数分布曲线中,约有近一半的蛋白酶处于二聚体的状态(4.1 s);仅加入2倍摩尔浓度的抑制剂5f(6 μmol· L-1)后,沉降系数分布曲线显示溶液中的SARS 3CL蛋白酶基本上全部以二聚体形式存在(4.2 s).该实验结果明确而直观地证明,随着抑制剂5f的浓度提高,SARS 3CL蛋白酶二聚体的形成能力增加,表明蛋白酶底物口袋结合小分子抑制剂后,改变了蛋白酶形成二聚体的能力.这与前面所得到的底物增强的酶二聚化现象一致.
进一步测定了3 μmol·L-13CL蛋白酶随抑制剂5f浓度变化的聚集状态分布情况,并由此估算出抑制剂5f对3 μmol·L-13CL蛋白酶诱导二聚的EC50值.测定了 5f浓度分别为 0、0.5、1、2、3、6 μmol·L-1时,3 μmol·L-13CL蛋白酶的聚集状态情况.如图4所示,在0.5-1 μmol·L-1的抑制剂浓度下,约半数蛋白酶处于二聚体形式,而蛋白酶与抑制剂在1∶1摩尔浓度下,90%以上的蛋白酶形成了二聚体,其对3 μmol·L-13CL蛋白酶的EC50值约为1 μmol·L-1.意味着只要溶液中约一半的蛋白酶分子与抑制剂分子结合后,几乎全部的蛋白酶分子都能形成稳定的二聚体.说明二聚体中的一个单体与抑制剂分子结合就可以提高二聚体的稳定性,不需要两个单体同时与抑制剂结合.我们的前期研究工作表明,SARS 3CL蛋白酶二聚体中只有一个单体是有活性的[17],但没有活性的那个单体是否也与底物结合尚不清楚.由于5f与底物具有类似的结合方式,从本工作的研究结果可以推测SARS 3CL蛋白酶二聚体中只有活性单体才能与底物结合.SARS 3CL蛋白酶形成二聚体的原因被认为是一种调控酶活性的方式,只有在足够高的酶浓度下才有活性可能是病毒为了等待有足够多的蛋白质表达出来再进行正确加工和组装的一种策略.我们的研究表明,即使只有较低浓度的酶,随着多聚蛋白底物浓度的升高,SARS 3CL蛋白酶会在底物的诱导下形成更多的二聚体,从而表现出更高的酶活性,这有可能是病毒用来调控多聚蛋白水解速度和组装时机的一种方法.

图3 沉降速率分析抑制剂5f诱导SARS 3CLpro的二聚体形成Fig.3 Sedimentation velocity analysis of the inhibitor 5f induced dimerization of SARS 3CL proteinase(A)the typical sedimentation velocity data of 3CLpro,(B)the residuals of fitting the upper panel A,(C)0 μmol·L-15f+3 μmol·L-13CLpro,(D)1 μmol·L-15f+3 μmol·L-13CLpro,(E)6 μmol·L-15f+3 μmol·L-13CLpro

图4 抑制剂5f浓度依赖的SARS 3CLpro聚集状态变化及EC50的测定Fig.4 Compound 5f concentration dependentsedimentation coefficient distribution and determination of EC50
3 结 论
利用酶活测定和分析型超速离心方法研究了SARS 3CL蛋白酶在底物和抑制剂存在下的二聚体形成情况.通过对蛋白酶活性随浓度变化曲线,拟合出在底物存在下二聚体的解离常数为0.94 μmol·L-1.为了进一步研究这种底物诱导二聚的现象,选用小分子抑制剂5f作为模拟底物,利用沉降速率方法测定了不同抑制剂浓度对于蛋白酶聚集状态的影响.研究结果表明SARS 3CL蛋白酶在溶液中二聚体的含量随抑制剂浓度的升高而增多,表现出一种抑制剂诱导的蛋白酶二聚现象.进一步测定了该抑制剂诱导二聚体形成的EC50值,发现蛋白酶二聚体中可能只有一个单体与抑制剂分子结合.该研究结果有助于进一步揭示SARS 3CL蛋白酶活性的别构调控机理,为新型抑制剂的设计提供新的思路.
1 Shigeta,S.;Yamase,T.Antivir.Chem.Chemother.,2005,16:23
2 Yang,H.;Yang,M.;Ding,Y.;Liu,Y.;Lou,Z.;Zhou,Z.;Sun,L.; Mo,L.;Ye,S.;Pang,H.;Gao,G.;Anand,K.;Bartlam,M.; Hilgenfeld,R.;Rao,Z.Proc.Natl.Acad.Sci.U.S.A.,2003,100: 13190
3 Lai,L.;Han,X.;Chen,H.;Wei,P.;Huang,C.;Liu,S.;Fan,K.; Zhou,L.;Liu,Z.;Pei,J.;Liu,Y.Curr.Pharm.Des.,2006,12: 4555
4 Bartlam,M.;Yang,H.;Rao,Z.Curr.Opin.Struct.Biol.,2005,15: 664
5 Anand,K.;Ziebuhr,J.;Wadhwani,P.;Mesters,J.R.;Hilgenfeld, R.Science,2003,300:1763
6 Anand,K.;Palm,G.J.;Mesters,J.R.;Siddell,S.G.;Ziebuhr,J.; Hilgenfeld,R.EMBO J.,2002,21:3213
7 Chen,S.;Chen,L.;Tan,J.;Chen,J.;Du,L.;Sun,T.;Shen,J.; Chen,K.;Jiang,H.;Shen,X.J.Biol.Chem.,2005,280:164
8 Zhong,N.;Zhang,S.;Zou,P.;Chen,J.;Kang,X.;Li,Z.;Liang,C.; Jin,C.;Xia,B.J.Virol.,2008,82:4227
9 Shi,J.;Wei,Z.;Song,J.J.Biol.Chem.,2004,279:24765
10 Marianayagam,N.J.;Sunde,M.;Matthews,J.M.Trends Biochem. Sci.,2004,29:618
11 Cameron,C.E.;Grinde,B.;Jacques,P.;Jentoft,J.;Leis,J.; Wlodawer,A.;Weber,I.T.J.Biol.Chem.,1993,268:11711
12 Darke,P.L.;Cole,J.L.;Waxman,L.;Hall,D.L.;Sardana,M.K.; Kuo,L.C.J.Biol.Chem.,1996,271:7445
13 Prabu-Jeyabalan,M.;Nalivaika,E.;Schiffer,C.A.J.Mol.Biol., 2000,301:1207
14 Lorenz,I.C.;Marcotrigiano,J.;Dentzer,T.G.;Rice,C.M.Nature, 2006,442:831
15 Fan,K.;Wei,P.;Feng,Q.;Chen,S.;Huang,C.;Ma,L.;Lai,B.; Pei,J.;Liu,Y.;Chen,J.;Lai,L.J.Biol.Chem.,2004,279:1637
16 Liu,S.;Pei,J.;Chen,H.;Zhu,X.;Liu,Z.;Ma,W.;He,F.;Lai,L. Acta Scientiarum Naturalium Universitaties Pekinensis,2003,35 (Suppl):62 [刘士勇,裴剑锋,陈 浩,祝小雷,刘振明,马文喆贺风雷,来鲁华.北京大学学报,2003,35(增刊):62]
17 Chen,H.;Wei,P.;Huang,C.;Tan,L.;Liu,Y.;Lai,L.J.Biol. Chem.,2006,281:13894
18 Wei,P.;Fan,K.;Chen,H.;Ma,L.;Huang,C.;Tan,L.;Xi,D.;Li, C.;Liu,Y.;Cao,A.;Lai,L.Biochem.Biophys.Res.Commun., 2006,339:865
19 Fan,K.;Ma,L.;Han,X.;Liang,H.;Wei,P.;Liu,Y.;Lai,L. Biochem.Biophys.Res.Commun.,2005,329:934
20 Huang,C.;Wei,P.;Fan,K.;Liu,Y.;Lai,L.Biochemistry,2004, 43:4568
21 Liu,Z.;Huang,C.;Fan,K.;Wei,P.;Chen,H.;Liu,S.;Pei,J.;Shi, L.;Li,B.;Yang,K.;Liu,Y.;Lai,L.J.Chem.Inf.Model.,2005, 45:10
22 Zhou,L.;Liu,Y.;Zhang,W.;Wei,P.;Huang,C.;Pei,J.;Yuan,Y.; Lai,L.J.Med.Chem.,2006,49:3440
23 Dam,J.;Schuck,P.Biophys.J.,2005,89:651
24 Schuck,P.;Millar,D.B.Anal.Biochem.,1998,259:48
25 Schuck,P.Anal.Biochem.,2003,320:104
26 Laue,T.M.;Stafford III,W.F.Annu.Rev.Biophys.Biomol. Struct.,1999,28:75
27 Schramm,H.J.;Nakashima,H.;Schramm,W.;Wakayama,H.; Yamamoto,N.Biochem.Biophys.Res.Commun.,1991,179:847
28 Marnett,A.B.;Nomura,A.M.;Shimba,N.;de Montellano,P.R. O.;Craik,C.S.Proc.Natl.Acad.Sci.U.S.A.,2004,101:6870
November 30,2009;Revised:March 15,2010;Published on Web:March 17,2010.
Substrate Binding and Homo-Dimerization of SARS 3CL Proteinase are Mutual Allosteric Effectors
WEI Ping1,2LI Chun-Mei1,2ZHOU Lu1LIU Ying1LAI Lu-Hua1,2,*
(1Beijing National Laboratory for Molecular Science,State Key Laboratory of Structural Chemistry of Unstable and Stable Species,College of Chemistry and Molecular Engineering,Peking University,Beijing 100871,P.R.China;2Center for Theoretical Biology,Peking University,Beijing 100871,P.R.China)
The 3C-like proteinase(3CLpro)of severe acute respiratory syndrome(SARS)coronavirus has been proposed to be a key target for anti-SARS drug discovery.It has been proposed and verified that the dimer was the active form of 3CLpro and only one protomer is active.In our previous work,we measured the dissociation constant(Kd)of the purified SARS 3CLpro using analytical ultracentrifugation at around 14.0 μmol·L-1.Using this Kdvalue,most of the SARS 3CLpro in the in vitro activity assay(1-3 μmol·L-1)might be in the monomer form and inactive.To explain this dilemma,we measured the enzyme activity change together with the enzyme concentration.By fitting the concentration dependent activity profile, the apparent dissociation constant was found to be 0.94 μmol·L-1,indicating a clear tendency toward substrate enhanced dimerization.This also explains why SARS 3CLpro was still active in the in vitro activity assay under a relatively low enzyme concentration.To further verify the substrate induced dimerization phenomenon,we selected a previously reported SARS 3CLpro isatin inhibitor,1-(2-naphthlmethyl)isatin-5-carboxamide(5f),which has similar binding interactions with the substrate and we studied its influence on SARS 3CLpro dimer formation using analytical ultracentrifugation.5f showed a strong ability to induce SARS 3CLpro dimer formation.By measuring the dimer and monomer distribution under different 5f concentrations,the EC50of dimerinduction was found to be about 1.0 μmol·L-1under an enzyme concentration of 3.0 μmol· L-1.This implies that only one protomer in the SARS 3CLpro dimer binds to the inhibitor or the substrate.As the apparent association constant and thus the enzyme activity of SARS 3CLpro increases with the concentration of the substrate,this may be a smart way to allosterically regulate the hydrolysis of the SARS viral polyproteins and the correct assembly of virons.
Allosteric effect;SARS;3CL proteinase;Analytical ultracentrifugation;Substrate enhanced enzyme dimerization
O641
*Corresponding author.Email:lhlai@pku.edu.cn;Tel:+86-10-62757486.
The project was supported by the National Natural Science Foundation of China(10721403,20473001)and the Ministry of Science and Technology of China.
国家自然科学基金(10721403,20473001)和国家科技部资助项目
