GLGI技术鉴定奶山羊乳腺中的κ-酪蛋白基因
2010-10-19孙瑞秋赵婧闫宏博李庆章高学军
孙瑞秋,赵婧,闫宏博,李庆章,高学军
(东北农业大学乳品科学教育部重点实验室,哈尔滨150030)
GLGI技术鉴定奶山羊乳腺中的κ-酪蛋白基因
孙瑞秋,赵婧,闫宏博,李庆章,高学军
(东北农业大学乳品科学教育部重点实验室,哈尔滨150030)
采用GLGI技术扩增本实验室构建的Long-SAGE标签库中的κ-酪蛋白基因。使用SMARTTM RACE cDNA Ampliication Kit通过RT-PCR把奶山羊乳腺组织中的mRNA逆转录成cDNA,分别用3’RACE和5’RACE扩增κ-酪蛋白基因的3’端和5’端。扩增产物经TA克隆后转化大肠杆菌,随机挑取阳性克隆后进行测序分析。通过序列比对、拼接,获得全长的κ-酪蛋白基因。应用GLGI技术成功获得了完整的奶山羊乳腺CSN3序列,为CSN3结构分析和功能研究奠定了重要基础。
GLGI,RACE,κ-酪蛋白基因
0 引言
Long-SAGE技术是一种高通量分析基因表达谱的方法,但长度仅为17 bp的标签序列很难精确地定位到单基因。采用GLGI(generation of longer cDNA fragments from SAGE tags for gene identification)技术可将Long-SAGE标签扩增至数百个碱基以上,较长的cDNA片段有利于基因结构分析和功能研究。
1988年Frohman等[1]发明了cDNA末端快速扩增技术(rapid amplification of cDNA ends,RACE),其实质是长距PCR,是一种应用通用引物和特异引物从低丰度转录本中快速扩增已知表达序列标签(expressed sequence tags,ESTs)5′和3′末端的简单有效的方法。
Shekar等[2]发现κ-酪蛋白基因在哺乳动物的泌乳过程中是必不可少的[3]。CSN3基因与产奶量、乳脂率和乳蛋白量显著相关[4]。本研究在优化GLGI条件的基础上,拟扩增并鉴定本实验室构建的奶山羊泌乳期乳腺组织的相关Long-SAGE标签库κ-酪蛋白基因。
1 材料和方法
1.1 材料
1.1.1 仪器
冷冻离心机(Z300-K),PCR仪(LabCycler,Senso-Quest),凝胶成像系统(ChampGel 6000)。
1.1.2 主要试剂
SMARTTM RACE cDNA Ampliication Kit,LA克隆试剂盒、EX Taq酶等,TOP10感受态细胞,胶回收试剂盒和质粒提取试剂盒,引物等。
1.1.3 LongSAGE标签
17bp的LongSAGE标签选自东北农业大学动物生物化学与分子生物学实验室建立的奶山羊乳腺上皮细胞泌乳期的LongSAGE标签文库,检索证实为高表达多重匹配标签并符合PCR引物设计要求。
1.2 方法
(1)总RNA的提取与检测。用Trizol法从奶山羊乳腺上皮细胞组织中提取总RNA,用甲醛变性电泳检测总RNA。
(2)cDNA的合成与验证。分别根据SMARTTM RACE cDNA Ampliication Kit逆转录合成3’cDNA和5’cDNA,以GAPDH作为内参照进行PCR检测cDNA。
(3)RACE条件扩增3’端。以RT-PCR产物3’cDNA为模板,κ-酪蛋白基因的特异标签序列(GSP)为上游引物CSN3:5’-CATGCGCAGGCTCATCAAAGA-3’。试剂盒中所含的通用引物(UPM)为下游引物。
PCR反应体系(25 μL总反应):
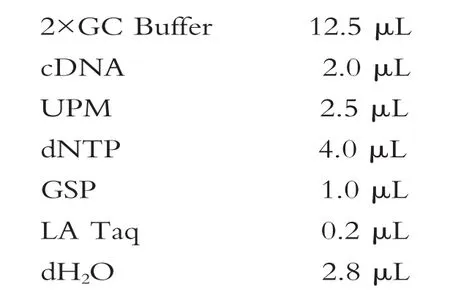
PCR反应条件:
(4)DNA克隆、鉴定和测序:PCR产物回收后,将其与pMDTM18-T载体连接。将4μL的连接产物转化大肠杆菌TOP10感受态细胞,并涂布于含有氨苄青霉素抗性的平板上,37℃培养10~16 h。随机挑取克隆菌落后增菌,提取质粒并进行质粒PCR鉴定,鉴定后的阳性质粒送到英俊公司测序。
(5)数据分析。测序结果经BLAST检索进行同源性分析。
(6)5’RACE引物的设计。用Primer5.0软件,在已测出的3’端序列中设计新的5’端引物。κ-酪蛋白基因引物为CSN3:5’-TTGGTCTCAGATGCACTCGCAATCG-3’。以5’cDNA为模板,UPM为下游引物,进行PCR反应,反应体系和条件同上。挑选阳性质粒测序。
(7)序列分析。将克隆测序得到的3’端序列与5’端序列进行拼接,得到完整cDNA序列,将所得序列在NCBI进行BLAST比对,以确认该序列与其它物种该基因的同源性。另外,在DNAstar软件上进行同源比对,并用Cluster W算法进行聚类分析得到系统发生树[5]。

2 结果
2.1 总RNA检测结果
电泳显示28S带亮度约是18S带的2倍,表明RNA的完整性(图1)。
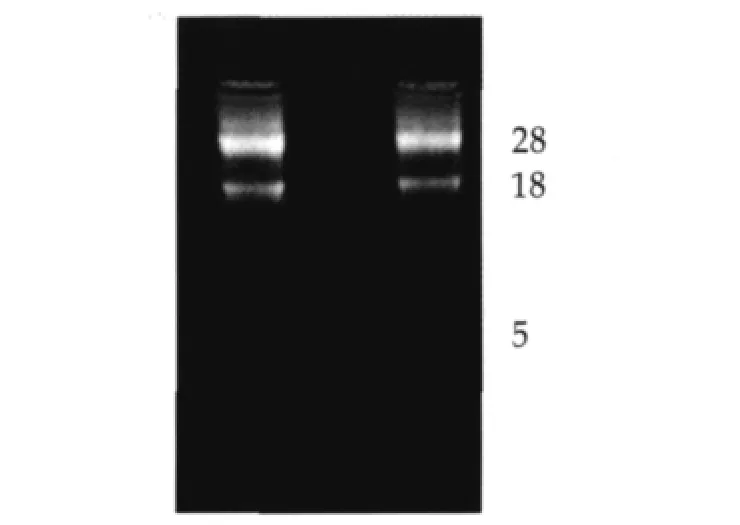
图1 奶山羊乳腺组织的总RNA电泳
2.2 RT-PCR检测结果
GAPDH检测cDNA的电泳显示269 bp处有单一清晰的条带(图2),表明cDNA完整。
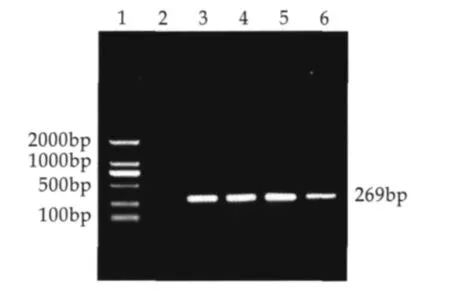
图2 RT-PCR检测结果
图2中,1为DNA Marker DL2000,2为空白对照,3和4为GAPDH验证3’cDNA,5和6为GAPDH验证5’cDNA。
2.3 κ-酪蛋白基因的3’RACE和5’RACE扩增结果
3’RACE的琼脂糖凝胶电泳图显示485 bp处出现一条单一的亮带(图3),5’RACE的电泳图显示672 bp处出现一条清晰的特异性条带(图4)。
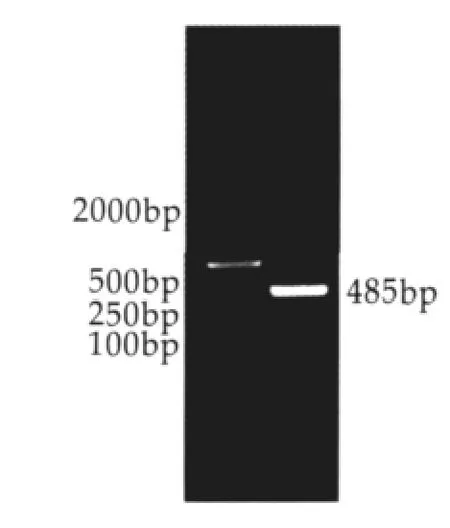
图33 ’RACE扩增结果
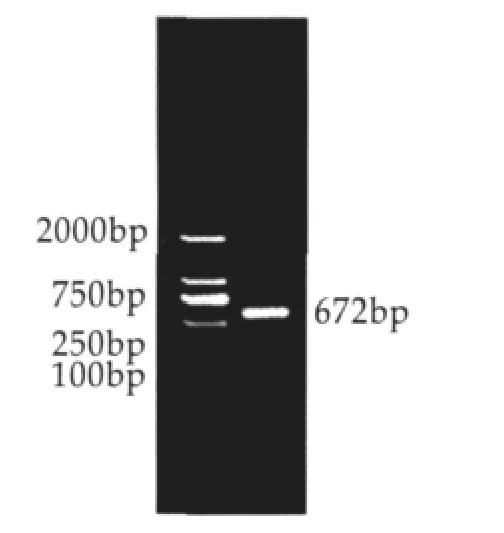
图45 ’RACE扩增结果
ACGCGGGGGGTTCCTTACAGTTGAAAGGC CAACTGAATCTACTGCCAAGCAAGAGCTGACG GTCACAAGGAAAGGTGCAATGATGAAGAGTTT TTTCCTAGTTGTGACTATCCTGGCATTAACCC TGCCATTTTTGGGTGCCCAGGAGCAAAACCAG GAACAACCGATATGCTGTGAGAAAGATGAAAG ATTCTTCGATGACAAAATAGCCAAATATATCC CAATTCAGTATGTGCTGAGTAGGTATCCTAGT TATGGACTCAATTACTATCAACAGAGACCAGT TGCACTAATTAATAGTCAATTTCTGCCATACC CATATTATGCAAAGCCAGTTGCAGTTAGGTCA CCTGCCCAAACTCTTCAATGGCAAGTTTTGCC AAATACTGTGCCTGCCAAGTCCTGCCAAGACC AGCCAACTACCCTGGCACGTCACCCACACCCA CATTTATCATTTATGGCCATTCCACCAAAGAAA GATCAGGATAAAACAGAAATCCCTGCCATCAAT ACCATTGCTAGTGCTGAGCCTACAGTACACAGT ACACCTACCACCGAAGCAATAGTGAACACTGTA GATAATCCAGAAGCTTCCTCAGAATCGATTGCG AGTGCATCTGAGACCAACACAGCCCAAGTTACT TCAACCGAGGTCTAAAAACTCTAAGGAGACATC AAAGAAGACAACGCAGGTCTAGCTGAAACCAAA TGACTACTTCAAACTTTCCTTTGGCCAGTTGTC TGCCTTCAGTGAACAGAGAGTATGATTTTCACA GATCCAGCTCCTTTCTCATCTCCTCTTACATTT TACATTTATGCCGCATTTAGTTTTTTGATTCCT GCATAATAAAGCCAATCAAATAAAAAAAAAAAA AAAAAAAAAAAAAAAAAAAA
2.4 κ-酪蛋白基因的3’端序列和5’端序列拼接后的结果
经克隆测序,将所得序列进行拼接得到1条长894 bp的cDNA片段。
2.5 比对结果
mRNA序列在NCBI上进行同源比对,发现所得CSN3序列与大羚羊的κ-酪蛋白mRNA(99%)、绵羊(95%)、牛(96%)、山羊(92%)高度同源。
用DNAstar软件将该基因序列进行同源性分析(图5),并将该基因各物种的核苷酸序列进行多序列比对,然后作系统发生树分析,发现奶山羊CSN3基因在分子进化上首先与山羊聚为一类,然后与绵羊聚在一起,最后与牛和大羚羊聚在一起(图6)。

图5 5个物种CSN3基因核酸序列同源性比较

图6 Cluster W作系统发生树分析
3 结果与讨论
本研究利用GLGI技术[6,7],将17 bp的SAGE标签前面加识别的碱基位点CATG作为5’端引物,3′端使用的是单一碱基dA、dG或dC与Oligo-dT结合的锚定引物进行第1次PCR扩增,只有与SAGE标签相匹配的序列才能退火并扩增。在第2次循环中,只有5′端OligodA的前一碱基与锚定的Oligo-dT相匹配的序列才能扩增,而单纯的Oligo-dA与Oligo-dT结合的片段因处于锚定状态则受抑制,因而最终得到的是含有SAGE标签的长片段DNA扩增产物,产物长度为200~1 000 bp[8,9]。
GLGI可以有效地解决tag特异性不足的问题,能够作出十分精确的全基因表达频谱;可以用来研究基因在转录过程中3’端的剪切修饰问题;对于一些实验方法不能得到的低丰度基因,若采用GLGI技术,则可以获得大量新基因。所以GLGI不但克服了SAGE技术[10]遇到的一些问题,而且又是SAGE的一个很好的补充,这两种方法的有机结合将是从整体水平分析基因表达的最有效的方法[11]。
SMART(switching mechanism at 5'end of the RNA transcript)技术的出现是一个新的里程碑。SMART法所需起始材料少,试验过程快速简单,全长比例也较高,目前已有商品化的试剂盒出售,国内大都采用这种方法构建cDNA SMART法文库[12-16]。其原理为:扩增标签的下游序列利用真核生物mRNA具有polyA尾巴的特性,在与其匹配的Oligo dT外侧加上特定的接头序列,逆转录后获得3’端连有特地接头的cDNA并,以其为模板,Long-SAGE标签加CATG识别序列作为基因特异性引物(GSP)和接头特异性引物分别为上下游引物扩增获得标签特异性cDNA片段。扩增标签的上游序列利用逆转录酶的末端转移酶活性,逆转录反应达到第一链cDNA的5’末端时会自动加3~5个dC,dC低温退火后与连有特定寡核苷酸序列(接头)的Oliogo dG配对,以其为模板,分别以与特定接头特异性引物和标签作为上下游引物扩增标签的5’cDNA片段。扩增产物经TA克隆后转化大肠杆菌,随机挑取阳性克隆后进行测序分析,通过序列比对、拼接即可获得较长甚至全长的cDNA片段。
降落PCR[17]是一种设计多循环以使相连循环的退火温度越来越低,从而达到最佳扩增条件的方法,反应过程中起始退火温度高于Tm值,随着循环的进行,退火温度逐渐降低到Tm值,从而确保第一个引物-模板杂交事件发生在最互补的反应物之间,退火温度最终低于Tm值。退火温度的范围可跨越15℃,从高于Tm(5℃)至低于Tm(10℃)左右,条件允许的话退火温度可以1℃或0.5℃递降。尽管退火温度最终会降低到非特异性杂交的Tm值,但此时目的扩增产物已开始几何扩增,在剩余循环中将超过任何非特异性产物的扩增量[18]。
本研究首次克隆得到奶山羊乳腺中CSN3的mRNA全长序列(894 bp)。完善了NCBI网站上公布的山羊CSN3序列,获得了完整的CSN3序列,为CSN3结构分析和功能研究奠定了重要基础[19,20]。
[1] FROHMAN M A,DUSH M K,MARTIN G R,et al.Rapid Production of Full-length cDNA,from Rare Transcripts:Amplification Using a Single Gene-Specific Oligonucleotide Primer[J].Proc Nat Acad Sci USA,1998,85:8998-9002.
[2] SHEKAR P C,GOEL S,RANI S D S,et al.Kappa-Casein Deficient Mice Fail to Lactate[J].Proceedings of the National Academy of sciences of the United States of America,2008,103(21):8000-8005.
[3] 李玲,李广,安小鹏,等.CSN1S2和CSN3基因表达量与西农萨能奶山羊产奶性状的关系[J].西北农林科技大学学报,2010,38(1):35-40.
[4] 陈宏,蓝贤勇,李瑞彪,等.CSN1S2、CSN3、β-lg基因对西农萨能奶山羊产奶性能的影响[J].遗传学报,2005,32(8):804-810.
[5] 伍晓雄,唐中林,李勇,等.猪PGAM2基因的克隆、序列特征及表达分析[J].畜牧兽医学报2008,39(9):1183-1189.
[6] CHEN J J,ROWLEY J D,WANG S M.Generation of Longer cDNA Fragments from Serial Analysis of Gene Expression Tags Forgene Identification[J].Proc Natl Acad Sci U S A,2000,97(1):349-353.
[7] CHEN J,LEE S,ZHOU G,et al.High-Through put GLGI Procedure for Converting a Large Number of Serial Analysis of Gene Expression Tag Sequences into 3′Complementary DNAs[J].Genes Chromosomes Cancer,2002,33(3):252-261.
[8] 李永平,周村建,郝飞.利用SAGE标签产生长片段cDNA应用于白念珠菌基因识别[J].第三军医大学学报,2005,27(20):2028-2030.
[9] 王惠琳,邓军,都飞,等.GLGI技术初步验证系统性红斑狼疮患者CD4+、CD8+T细胞基因表达谱[J].中华皮肤科杂志,2006,39(9):521-523.
[10] Velculescu V E,Zhang L,Vogelstein B,et al.Serial Analysis of Gene Expression[J].Science,1995,270(5235):484-487.
[11] 黄健华,苗雪霞,黄勇平.基因表达研究新方法SAGE和GLGI及其在家蚕功能基因组研究中的应用前景[J].蚕业科学,2004,30(4):395-398.
[12] 李长春.藏鸡腿肌全长cDNA文库构建与其ESTs的生物信息学分析和验证[D].武汉:华中农业大学,2005.
[13] 陆涛峰.鄂伦春马cDNA文库构建及CCL2基因的克隆与表达研究[D].北京:中国农业科学院,2008.
[14] 刘琳玲,丛波,赵海平,等.利用SMART技术构建蓝狐脾脏cDNA文库[J].特产研究,2008(1):6-9.
[15] 魏东霞,邓艳,吴绍强,等.用SMART技术构建猪蛔虫雌虫cDNA文库[J].中国预防兽医学报,2007,29(3):180-184.
[16] 吴茜,田可川,刘武军,等.利用SMART技术构建新吉细毛羊皮肤组织全长cDNA文库[J].西北农林科技大学学报,2010,38(5):46-50.
[17] HELWEG-LARSEN J,JENSEN J S,BENFIELD T,et al.Diagnostic Use of PCR for Detection of Pneumocystis Carinii in Oral Wash Samples[J].J Clin Microbiol,1998,36(7):2068.
[18] 万兵,闫杰,季明春,等.一种优化的PCR方法——降落PCR扩增目的基因[J].江苏临床医学杂志,2002,6(2):127-129.
[19] 纪英卓,白文林,张世伟,等.辽宁绒山羊和内蒙古绒山羊CSN3基因遗传多态性的比较分析[J].遗传育种,2007,43(7):4-6.
[20] 蓝贤勇,陈宏,潘传英,等.CSN3、CSN1S2和β-1g基因多态与西农萨能奶山羊产羔数的相关性研究[J].中国农业科学,2005,38(11):2333-2338.
Identification of κ-casein gene by GLGI technique in long-sAGE tags of dairy goat mammary gland
SUN Rui-qiu,ZHAO Jing,YAN Hong-bo,LI Qing-zhang,GAO Xue-jun
(The Key Laboratory of Dairy Science of Ministry of Education,Northeast Agricultural University,Harbin 150030,China)
An optimized procedure of GLGI was adopted to amplify κ-casein gene sequence in Long-SAGE tags of dairy goat mammary gland.SMARTTM RACE cDNA Ampliication Kit was used to mRNA of dairy goat mammary gland tissue reverse transcribe to cDNA by RT-PCR.3′and 5’cDNA fragments of κ-casein gene were extended by 3’RACE and 5’RACE respectively.Products of PCR were T/A-cloned and transformed to E.coli.Positive colonies were sequenced.Total length κ-casein gene was obtained by sequence contrasting and splicing.Complete CSN3 sequence of dairy goat mammary gland were successfully identified by applying this high-throughput procedure,which established a basis for further structural and functional analysis of CSN3.
GLGI;RACE;κ-casein gene
Q93-33
A
1001-2230(2010)12-0004-04
2010-07-12
黑龙江省国际合作项目,东北农业大学创新团队项目(CXT005-1-1)。
孙瑞秋(1985-),女,硕士研究生,研究方向为泌乳生物学与乳腺生物功能调控。
李庆章
