利用15个微卫星位点对荷斯坦奶牛进行亲子鉴定
2010-09-25钟美明中国海洋大学海洋生命学院山东青岛266000
钟美明 (中国海洋大学海洋生命学院 山东青岛 266000)
微卫星标记(microsatellite),又称为短串联重复序列(simple tandem repeats,STRs)或简单序列重复(simple sequence repeats, SSRs),是均匀分布于真核生物基因组中的简单重复序列,由2~6个核苷酸的串联重复片段构成,由于重复单位的重复次数在个体间呈高度变异性并且数量丰富,所以被广泛应用于亲子鉴定。RD Schnabel[1]等研究证实了利用12个微卫星位点足以对北美野牛和家养牛进行常规性的亲权鉴定,被测群体中野牛bison排除率达到0.9955,cattle的排除率达到0.9995;V Rehout[2]等利用10个微卫星位点对荷斯坦奶牛进行亲权鉴定,位点的杂合度最低0.607,最高0.835,多态信息含量最低0.575,最高0.816,10个位点的联合排除率达到0.999;根据2004年9月11~16日在东京举行的第29届ISAG国际会议上,其中一篇文献中提到,随机抽取了58头斯洛伐克荷斯坦牛的血液样品,进行11个微卫星位点的检测,联合排除率达到0.999[3]。国内在利用微卫星进行亲子鉴定方面的研究也很多,例如高爱保等[4]利用多重PCR扩展20个微卫星标记,对凉山半细羊毛进行亲权鉴定,双亲未知时,累积排除率达到0.998666;在父系半同胞基础上进行母子亲权鉴定,累积排除率达到0.999994;贾名威等[5]采用6个STR基因座对7头奶牛进行鉴定,父权概率达到0.99993;管峰等[6]应用4个STR位点对2份冻精和4头可疑种公牛进行个体鉴定,鉴别能力达0.9999。除此之外,微卫星标记在兔、犬、虎和熊猫等动物中都有非常广泛的应用[7-9]。
北美荷斯坦奶牛具有产奶量高、奶质好等特点,引进和繁育荷斯坦奶牛有利于提高中国奶品的质量。准确的系谱记录对现代奶牛育种是十分重要的,但实际中有很多原因会导致系谱记录错误,例如精液编号错误,混合精液授精,放牧方式饲养等。本试验中检测的奶牛均属于混合精液体外受精,因此对这些奶牛进行亲子鉴定检测具有十分现实的意义。本文通过检测荷斯坦奶牛的15个微卫星位点的多态性,旨在建立一种快速准确检测荷斯坦奶牛的方法。
1 材料与方法
1.1 试验材料
本试验检测的350头奶牛是经过体外受精,再胚胎移植到其他牛体内发育的。卵细胞来自屠宰场的卵巢,35支精液属于荷斯坦种公牛,体外授精时同时有3~4个父本存在。
1.2 主要试剂和仪器
试剂:10×PCR buffer、MgCl2、dNTP、TaqDNA聚合酶,引物,尿素,N,N-亚甲双丙烯酰胺,丙烯酰胺,三羟基氨基甲烷(Tris),十二烷基磺酸钠(SDS),去离子甲酰胺,四甲基乙二胺(TEMED)均来自上海生工,Marke为自制。
仪器:ABI PRISMTM377测序仪,PCR热循环仪等
2 试验方法
2.1 DNA模板提取
2.1.1 血液DNA的提取 取100µl左右的全血,剪碎凝块→加入全血DNA抽提液600ml及10mg/ml的蛋白酶K10µl,充分摇匀→在水浴锅中56℃隔夜消化→加入500ml苯酚,振荡30min→12000rpm离心10min→取上清,加入500ml苯酚,振荡30min→12000rpm离心10min→吸上清,加入500ml氯仿,振荡30min→12000rpm离心5min→吸上清,加入适量冻存无水乙醇,颠覆转动至DNA聚集成团→除去上清→加入70%的乙醇清洗DNA→12000rpm离心2min→除上清,干燥30min→加入100µl左右TE,溶解,留作模板。
2.1.2 冻精DNA的提取 精液取100µl入1.5ml tube→12000rpm,10min→200µl PBS重悬→12000rpm离心10min→500µl SMB重悬→12000rpm离心5min→加入500µl SMB,10% SDS 12.5µl,蛋白酶K 5µl, 重悬精子细胞,震荡混匀→37℃水浴过夜→500µl 苯酚氯仿异戊醇,12000rpm离心5min→移上清至新tube,加入500µl氯仿异戊醇,12000rpm离心5min→移上清至新tube→加入2M NaOAC 50µl和 500µl异 丙 醇 → –20℃ 3h→12000rpm 离 心15min→70% EtOH清洗沉淀→12000 rpm离心15min→55℃下用100µl 水溶
2.2 PCR反应体系
15组引物分成5组进行多重PCR,每对引物的反应体系如下:10×PCRbuffer2.5µl,MgCl21.5µl,dNTP0.5µl,去离子水6µl,正反引物各1µl,TaqDNA聚合酶0.2µl。PCR扩增程序:38 ℃ 2min ;95 ℃ 5min 预变性;94 ℃ 25min变性;55℃ 30min 退火;72℃ 45min延伸,循环35次;72 ℃ 2min最后延伸,分组情况见表1。
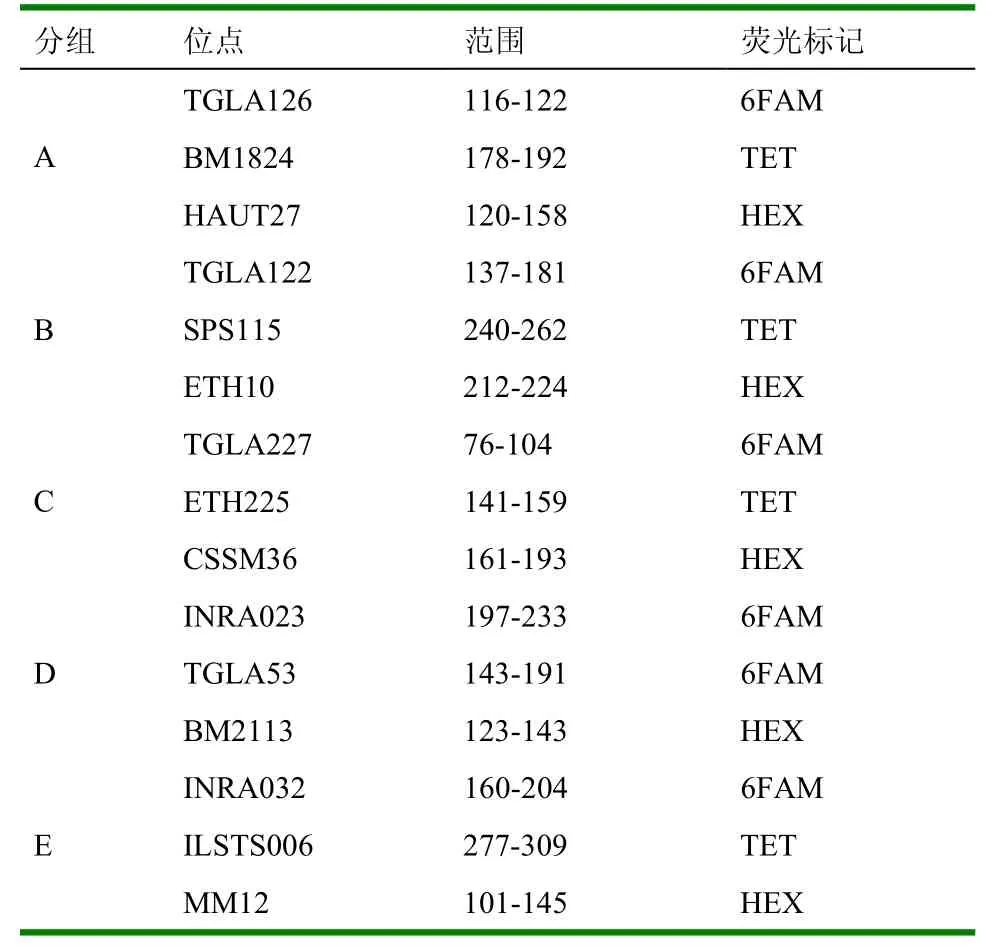
表1 复合PCR分组情况
2.3 微卫星标记与引物
微卫星位点的选择需要满足以下条件[10]:(1)多态性高。具体体现在等位基因数目多,多态信息含量和杂合度值高。(2)没有无效等位基因。(3)位点不连锁。即位点要求不在同一条染色体上或者同一条染色体上的位点距离大于40cM。例如本试验中ETH225(8cM)和MM12(77cM)在同一染色体上,但距离超过40cM[11]。(4)已知等位基因长度范围。(5)适合多重PCR。
依照以上条件,并且根据ISAG(the Interneta-tional Society for Animal Genetics)推荐的位点和自选位点,经试验最终确定15个微卫星位点,其中9个是ISAG推荐的核心位点组合(BM1824, BM2113, INRA023, SPS115, TGLA122,TGLA126, TGLA227, ETH10, ETH225),引物序列见表2。
2.4 产物检测及亲权鉴定
本试验PCR产物变性后经过ABI377测序仪电泳,得到的电泳图像由GenescanV3.7.1和Genotyper分析后得到微卫星位点的基因型,建立基因型数据库。用Cervus2.0利用基因型数据库,分析得到每一位点的等位基因频率,杂合度的观察值和期望值,多态信息含量(PIC)以及排除率,同时进行Hardy-Weinberg平衡和无效等位基因频率分析。
Cervus软件的亲权鉴定原理[12]是将候选父的基因型与子代基因型比较,如果在一个或多个位点出现不匹配那就排除,剩余不能排除的候选父再通过似然比确定。似然分析以一个数据为起点,评估给出这个数据的假设。似然值(likelihood)L在假设条件H下给出数据D,记作L(H│D)。似然值往往是与另一个似然值相比较的情况下给出的,即似然比L(H1,H2|D)=P(D|H1)/ P(D|H2),P(D|H)表示在H假设下得到数据D的概率,P(D|H1)一般指假设父是真正父的概率,P(D|H2)一般指假设父不是真正父的概率。

表2 各位点的染色体位置和引物序列
母本基因型不知道的情况下,似然比公式:L(H1,H2|ga,go)=T(go|ga)*p(ga)/P(go)*p(ga)=T(go|ga)/P(go),T(go|ga):根据假设父得到的子代基因型频率,P(go):子代基因型频率
一个大的似然比表示候选父是真正父比不是真正父的可能行更大。似然比是针对单个位点的,所有位点结合起来总的似然比相乘,取以e为底的对数就得到了LOD值。将LOD>0的值按从大到小排序,其中最大的LOD1,其次LOD2,Delta=LOD1-LOD2,即比较most-likely与second-likely候选父之间的LOD值。
2.5 统计分析
2.5.1 平均排除率 将所有位点的排除率通过基因型频率加权得到总的平均排除率。与以似然值为基础的亲权鉴定方法没有太大关系,尤其当错误率大于0时,但是可用来与前人或发表文献中的数据作比较。
2.5.2 可信度检验 LOD或Delta不能用标准的卡方分布来检验,所以Cervus用亲权模拟(simulation)来评估可信度。模拟的目的是通过比较大量随机后代的LOD,得到大量的Delta分布(模拟10,000次)。通过比较最可能父(most-likely parent)是真正父(true parent)时Delta分布与最可能父不是真正父时Delta分布,得到Delta关键值,假如这个关键值是在特定可信度水平如95%确定的,那么当分析真实数据的亲权关系时,最可能父的Delta值超过Delta关键值,其亲权鉴定的可信度即为可信度95%。
3 结果和讨论
3.1 Hardy-Weinberg平衡
Cervus检测结果显示,除INRA043和ILSTS006外,其他位点均符合Hardy-Weinberg平衡。Cervus中指出一两个位点的H-W不平衡并不影响似然比的计算,大量的不平衡就可能影响结果。INRA043和ILSTS006不平衡的原因不是因为分型错误或无效等位基因,而是因为群体繁育的父本局限在35头荷斯坦种公牛,因此可能造成了某些基因的频繁出现。这种情况下仍可以用Cervus软件分析亲权关系,但是应注意结果的可信度。
3.2 杂合度和多态信息含量
杂合度和多态信息含量结果见表3。杂合度的观察值最小为0.377(SPS115),最大为0.852(TGLA227),平均为0.612;杂合度的期望值最小为0.362(SPS115),最大为0.827(TGLA227),杂合度观察值的平均值为0.681。杂合度的期望值和观察值的最小差值为0.002(INRA023),最大差值为0.074(CSSM136)。差值小于0.2的位点有6个,差值在0.2~0.3之间的位点有4个,差值在0.3~0.5的位点有2个,差值在0.5~0.6的位点有2个。大部分位点的杂合度的观察值和期望值差异较小,说明等位基因分布比较合理。
多态信息含量在0.345-0.804之间变动,平均值为0.639。当PIC>0.5时,该位点具有高度多态性,当0.25
3.3 排除率
Excl(1)即排除率1,是指当父母双方均未知的情况下位点的排除率;Excl(2)即排除率2,指当父母一方已知的情况下位点的排除率,所有位点的平均排除率是各个位点的排除率通过基因型频率加权的方法计算所得。通过计算得出当父母双方均未知的情况下,15个位点的联合排除率为0.995492,当父母一方已知的情况下,15个位点的联合排除率为0.999936,因此这15个位点具有极高的排除率,足够用于holstein奶牛的检测。但是为了保证结果的准确性,当子代和候选父有2个或2个以上位点不同时,排除其亲缘关系。各个位点的排除率见表3。
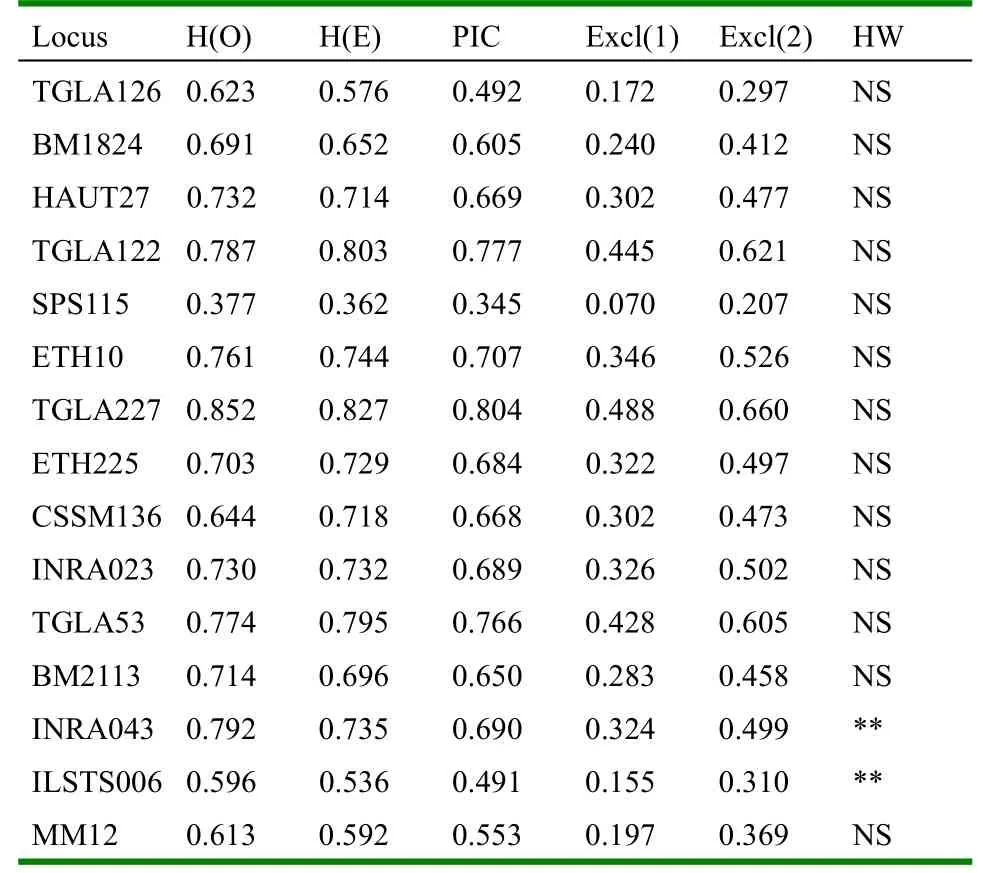
表3 15个位点的杂合度、多态信息含量、排除率和Hardy-Weinberg平衡
3.4 鉴定结果
本试验采用Cervus使用的似然值为基础检测亲子关系,不能用标准的卡方分布来检验,因此采用模拟(simulation)。分析结果中Delta最小值0.027,最大值7.19,LOD最小值0.0014,最大值9.7038,LOD值越大越可能是亲生父。最终289头被测小牛找到了亲生父,可信度达到95%,并且位点能完全匹配。但因为群体取样的局限性,造成其余61头虽然能找到完全匹配的候选父,但可信度达不到95%。所以为保证结果的准确性,只有所有位点匹配才能确认亲子关系,两个或两个位点以上的不匹配的排除亲子关系。
4 结论
在胚胎移植过程中容易因冷配产生奶牛和其他种群牛的杂交种,实际检测中荷斯坦奶牛与非荷斯坦奶牛的微卫星位点序列具有非常明显的差异(本文中未显示),因此这15个微卫星标记足够进行日常的荷斯坦奶牛的检测。微卫星标记方法对于大批量亲子鉴定检测具有快速、准确和经济的优点,对于未来国内发展大规模规模化养殖和繁育具有很重要的意义。
本试验的目的在于分析荷斯坦奶牛15个微卫星位点的遗传多态性,并以此为基础建立起快速准确的鉴定荷斯坦奶牛的方法。
[1] R D Schnabel, T J Ward, J N Derr. Validation of 15 microsatellites for parentage testing in North American bison,Bison bison and domestic cattle,Animal Genetics,2000,31,360-366.
[2] V.Rehout, E.Hradecka, J.Citek. Evaluation of population testing in the Czech population of Holstein cattle, Czech J.Anim.Sci.,51, 2006(12):503-509.
[3] Daniela Jakabova, Jozef Trandzik, etc. Microsatellite DNA polymorphism of Holstein cattle in Slovakia. Development of Genetic Research and Animal Production[C], 2004.
[4] 高爱保,吴登俊.利用微卫星标记进行凉山半细毛羊亲权鉴定的研究[J].遗传.2005,27(1):85-90.
[5] 贾名威,杨利国等. 应用6个STR基因座进行奶牛亲子鉴定[J]. 南京农业大学学报. 2004, 27(1): 74-77.
[6] 管峰,杨利国,艾君涛等. STR基因座在奶牛个体识别中的应用[J].畜牧兽医科学.2005, 21(2): 4-6.
[7] 韩春梅,张嘉保,高庆华等. 微卫星DNA在吉戎兔亲子鉴定中的应用研究[J]. 遗传. 2005,27(6):903-907.
[8] 叶俊华,麻俊武,杨前勇等. 利用10个STR-DNA的多态性进行警犬亲子鉴定[J]. 江西农业大学学报. 2005,27(1): 110-113.
[9] 张于光,李迪强,饶力群等. 东北虎微卫星DNA遗传标记的筛选及在亲子鉴定中的应用[J]. 动物学报.2003, 49(1): 118-123.
[10] R D Schnabel, T J Ward, J N Derr. Validation of 15 microsatellites for parentage testing in North American bison bison,Bison bison and domestic cattle.Animal Genetics, 2000,31:360-366.
[11]Joao F.C.de Oliveira, Jairo P. Nevesl, Eric A.Almeida, Clara Sabina Steigleder, Jose Carlos F.Moraes, Paulo Bayard D.Goncalves and Tania A.Weimer. Association between reproductive traits and four microsatellites in Brangus-Ibage cattle.Genetics and Molecular Biology,2005,28(1),54-59.
[12]T.C.Marshall, J.slate, L.E.B.kruuk, J.M.Pembrton. Statistical confidence for likelihood-based paternity inference in natural populations.Molecular Ecology.1998,7:639-655.
