肝脏肿瘤三维可视化方法研究
2010-09-11艾海明吴水才杨春兰高宏建
艾海明 吴水才 杨春兰高宏建 曾 毅
(北京工业大学生命科学与生物工程学院,北京 100124)
肝脏肿瘤三维可视化方法研究
艾海明 吴水才 杨春兰*高宏建 曾 毅
(北京工业大学生命科学与生物工程学院,北京 100124)
肝脏肿瘤三维可视化是肝癌微波热疗规划系统设计中的关键技术,其重建效果的好坏将直接影响到手术计划的可靠性和有效性。本研究提出一种基于自动分割的肝脏肿瘤三维可视化新方法。首先使用基于图论的方法自动分割出肝肿瘤,然后借助新的移动立方体算法重建出肿瘤表面,最后调用OpenGL库进行肿瘤模型绘制与显示。对6例肝癌患者CT图像进行肿瘤三维重建,实验结果表明;分割过程不需要人为干预,三维重建时程也较短,肿瘤模型的显示效果令人满意。
肝脏肿瘤;图像自动分割;三维重建
Abstract:3D visualization of hepatic tumor is a key technique in preoperative planning system of microwave hepatic tumor ablation,whose performance determines the reliability and effect of surgical planning result.In this paper,a new method for 3D visualization of hepatic tumor was proposed.Firstly,a segmentation method based on graph-theory was applied to automatically extract hepatic tumor.Secondly,a new marching cubes algorithm was employed toreconstruct tumor surface.Finally,surfacerenderingand displayingwere accomplished by 3D graphic library OpenGL.The proposed method was evaluated via CT images of 6 patients suffering from hepatic carcinoma.The results showed that the 3D reconstruction was fast and the visualization of the tumor model was satisfying without manual intervention.
Key words:hepatic tumor;image auto-segmentation;3D reconstruction
引言
肝癌是我国常见的恶性肿瘤之一,死亡率极高。微波热疗以其微创、操作方便和低并发症等优点受到人们的关注,在国内它已被用于治疗早期小型肝癌[1]。为了提高治疗的可靠性和安全性,使用“肝癌微波热疗手术规划系统”可在术前基于肝脏CT扫描图像,对肝肿瘤进行三维重建和可视化,通过对微波热疗温度场进行计算机模拟,从而制定出合理的手术方案[2]。基于CT扫描图像的肝脏肿瘤三维可视化是其关键技术之一,肿瘤重建效果的好坏直接影响系统输出手术计划的准确性。
医学图像自动分割一直是三维可视化技术中面临的一项关键技术难题,其主要原因是由于医学图像中相邻组织间灰度值相近,同一组织的灰度值不均匀和部分容积效应造成组织间边缘模糊[3]。文献[4]提出一种基于图论的有效分割算法,它的主要特点是能够保留低变化区域的细节,同时忽略高变化区域的细节。针对肝肿瘤CT图像的特点,借助该算法并将其成功地应用到肝肿瘤的自动分割中。
肝肿瘤三维可视化是通过对一系列肝肿瘤CT图像进行分割处理后,采用面绘制或体绘制算法重新还原出肝肿瘤的三维图像,它帮助医师对肝肿瘤大小、形状和空间位置获得定量描述。相比于体绘制技术,面绘制方法以其绘制速度快、便于实时交互、数据存储容量小等优点得到广泛的应用[5]。针对传统MC(marching cubes)算法存在的固有缺陷,文献[6]提出了一种结合图像分割与MC算法的新移动立方体(NMC)方法,它可以根据不同医学图像的特点采用恰当的分割方法,避免了MC只适合于阈值分割的局限性,同时采用一种基于区域增长的立方体检测方法,提高了表面跟踪的效率。借助NMC方法思想,将基于图论的分割算法与MC算法有机地结合,利用肝肿瘤分割结果构造等值面,表面跟踪时也采用基于区域增长的立方体检测方法,最后调用OpenGL库对肝肿瘤等值面进行显示。
1 基于图论的自动分割算法
1.1 区域比较谓词
区域比较谓词D用于判断分割过程中相邻区域间是否存在边界[4]。它通过对区域内部差异和区域间差异进行比较,使得图像分割结果能够自适应图像数据的局部特征。
设C是分离集森林V中某个集合,其最小生成树为MST(C,E),则集合 C的内部差异可定义为最小生成树中最大的权值

集合C1,C2⊆V,则两集合间差异为连接该两集合边的最小权重
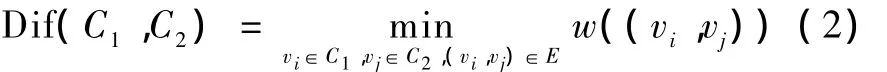
根据上述公式的定义,可引出区域比较谓词D
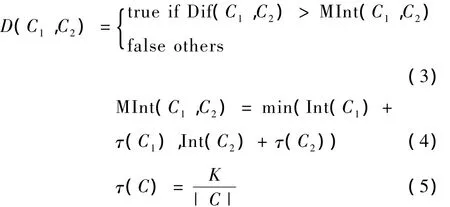
式中τ(C)为阈值函数,|C|为C的元素数目,K为常量参数,它用来控制过分割产生的小区域,参数K取值越大,抑制小区域的效果越显著。通常,肝肿瘤 CT图像分割中 K取值范围为[100,500]。
1.2 分割算法流程
输入图G=(V,E),图G中存在 n个节点,m条边,V的区域分割结果输出为S=(C1,C2,…,Cr),则分割算法过程如下[4]:
1)边集 E 分成 π(o1,o2,…,om),其中边的权重非递减进行排序
2)初始分割S0,这里每个顶点 vi对应某个子区域
3)重复第 3 步,其中 q=1,2,…,m
4)由动态分割集 Sq-1构造分割集 Sq:已知,;若,则合并Sq-1中两元素,子区域合并后 Sq-1⇒Sq;否则 Sq=Sq-1
5)返回S=Sm
借助区域比较谓词,该分割算法的本质是利用最小生成树寻找图像同质区域的过程,它能在近似线性时间O(nlogn)内完成图像分割,并且分割结果满足区域全局特性。
2 新移动立方体方法
新移动立方体方法(New Marching Cubes,NMC)算法综合了 Cuberille与 MC 算法[6],Cuberille算法将三维体数据分割成物体和背景两个部分,然后对物体数据的每一个采样点构建立方体,并求出表面法向量,把立方体体素的各个面投影到屏幕,利用Z-Buffer算法判断遮挡关系,显示出分割结果的等值面。MC算法通过相邻体数据切片间构建体素,根据经验设定分割阈值,利用线性插值求出每个立方体体素的三角剖分,将整个物体表面以三角面片的形式表示出来。本研究NMC算法描述如图1所示。

图1 新移动立方体算法步骤Fig.1 Algorithm steps of new marching cubes
法向量是通过对原始肝肿瘤CT序列图像进行中心差分计算获得,从而保证重建效果比较平滑。

式中,g(i,j,k)表示点(i,j,k)的灰度值。
NMC表面跟踪算法采用文献[6]提出的算法步骤,在此不再详述。
3 材料与方法
3.1 数据获取
实验CT图像数据集来源于中国人民解放军总医院提供的临床数据,共有6例肝癌患者的完整腹部序列图。原始肝肿瘤CT图像均为DICOM格式,采用自行开发的软件转换成通用图像格式(采用BMP格式),以便于后期数据处理。每组序列图包含53张腹部 CT图像,体素大小为0.703 mm×0.703 mm×2.5 mm,其中肿瘤图像断层数从16到28不等。
3.2 图像处理和肿瘤三维重建
1)平滑处理
由于医学图像存在噪声和运动伪影,使得图像质量变得模糊,使用高斯滤波器对原始CT图像进行平滑处理,其中高斯参数σ通常设置为0.8。
2)图像自动分割
定义一个无向加权图G(V,E),肝肿瘤CT图像的像素pi对应于图的节点vi,图的边 ei表示相邻两像素。这里每个像素采用8邻域系统,节点属性表示为像素灰度值,边属性表示像素间的灰度差值。另外,边权重函数w((vi,vj))定义为边上相邻像素灰度差值的绝对值,即

分割过程中主要有2个可调参数即 K和M,K在前面已有详述。M是小区域合并参数[4],其取值越大,抑制小区域的效果越显著,通常取值范围为100~200。
3)肝肿瘤区域提取
图像分割结果满足视觉全局特性,即不仅分割出肝肿瘤还包括其它组织如胃。由于分割后的每个子区域采用同一种伪彩色显示,因此人机交互式处理,在肝肿瘤的伪彩色区域选取一个种子点使用区域生长法提取出肝肿瘤。具体操作流程如图2所示。

图2 肝肿瘤区域提取算法流程图Fig.2 Algorithm flowchart of liver tumor extracting
4)边缘修正
由于医学图像边缘模糊,以及分割算法本身容易生成小区域,因此提取出的肝肿瘤边缘可能包含过于冗长的细小边缘。针对这种情况,借助数学形态学的方法进行边缘修正,主要操作算子包括膨胀、腐蚀、开运算和闭运算。
5)肝肿瘤三维重建
医学图像软件工具包MITK(Medical Imaging Toolkit)是建立在 VTK和 ITK的基础上,在统一的框架下实现医学影像的分割、配准、三维可视化算法[7]。肝肿瘤三维重建算法由 MITK 中mitkBinMarchingCubes类实现。它接受两个输入:一个是原始的肝肿瘤体数据,用于计算法向量;另一个是肝肿瘤经分割后得到的二值数据,用于计算等值点坐标。只要当前立方体的某边两端灰度值不同,则认为该边与等值面相交。
4 结果
肝肿瘤三维重建是在Windows XP操作系统下,利用Visual C++6.0编程实现的。对6例肝癌患者的CT图像进行了三维可视化实验,重建后的肝肿瘤模型均令人满意。图3为对其中某个患者的肝肿瘤,分别采用本方法和商业软件Amira进行三维重建的显示结果。图(a)为肝肿瘤CT序列图中一幅具有代表性的原始图像。图(b)是肝肿瘤图像的分割结果,其中分割参数K和M分别取值155、195,分割时程为2 s,共得到96个子区域,肝肿瘤分割结果显示为黄色区,该区域与原始图像的肝肿瘤在形状、大小均十分接近。图(c)为肝肿瘤提取后的二值图像,肿瘤区域灰度值为255。图(d)描述了肿瘤边缘修正结果,它先通过腐蚀运算消除冗余边缘,再通过膨胀算法得到修正后的肿瘤边缘,其中结构元素使用7×7像素的圆形像素块。图(e)为肝肿瘤三维模型,它共由26个肿瘤切片重建而成,三维重建算法执行过程耗时约5 s(在惠普工作站HP xw8400中)。为评价本方法重建肝肿瘤模型结果的准确性,借助商业软件Amria3.1进行了肝肿瘤模型重建,其中分割过程是在专业医师的指导下手工分割,并采用Amria提供的表面绘制方法进行肿瘤模型表面绘制。图(f)显示了基于Amira的肝肿瘤三维模型。由图(e)和图(f)比较可知,两者肿瘤模型的形状、大小均十分相似,只是商业软件重建的表面模型更加光滑,但这并不影响临床上定量评价该模型参数。

图3 肝肿瘤三维重建中各步操作结果与基于Amira的肿瘤模型。(a)原始肝肿瘤CT图像;(b)图像分割结果;(c)肿瘤二值图像;(d)肿瘤边缘修正结果;(e)肝肿瘤三维模型;(f)基于Amira的肝肿瘤模型Fig.3 The results of each step of 3D hepatic tumor reconstruction and hepatic tumor model based on Amira.(a)original CT image of hepatic tumor;(b)segmentation result;(c)binary image of hepatic tumor;(d)refinement of tumor boundary;(e)3D model of hepatic tumor;(f)3D model of hepatic tumor based on Amira
5 讨论和结论
医学对象三维可视化主要涉及2个关键技术即图像分割和三维重建算法的研究。传统的手动分割和交互式半自动分割工作量大、耗时长、易受人为主观影响且分割结果重复性差,不适合分割对比度低和边缘模糊的医学图像。本研究的图论分割算法能够解决上述不足,它可以快速、准确地分割出肿瘤组织,且分割过程不需要人为的干预。另外,它还可以应用到其它组织的分割如肝脏、胃等,由图3(b)可见,肝脏分割结果显示为绿色区,它与图3(a)中的肝脏形状、大小很相似。当然,基于图论的分割算法也存在一些不足,分割参数取值不同有时造成肿瘤分割结果差异较大,分割参数并不一定适用于肝肿瘤CT序列图中的其它图像。要克服这些不足,需要进行大量的肿瘤分割实验,统计分割参数,根据经验参数进行适当调整就可以节省寻找最佳分割参数的时间。
研究中采用的表面重建算法[6]克服了传统表面重建算法中分割时间长、不适合人机交互等缺点。它充分利用硬件加速,并采用OpenGL技术进行三维显示,缩短了绘制时间。另外,该重建算法在计算表面法向量时采用原始图像数据,因此重建出的肝肿瘤模型表面比较光滑(见图3(e)),避免了传统移动立方体算法中经常出现的“鱼鳞”现象。
基于图论的分割方法很好地解决了肝肿瘤自动分割的难题,是一种收敛性和健壮性较好的自动分割算法,可以分割出形状、大小各异的肿瘤,肿瘤边缘分割结果清晰。基于图论分割的新移动立方体表面重建算法可以精确地重建出肝肿瘤模型,其重建效果可与三维可视化商业软件Amira相媲美。当然,该模型表面并不十分平滑,主要有2方面原因:一是原始肝肿瘤CT图像数据量小,相邻断层图像间距过大,引起局剖细节信息丢失;二是肿瘤生长出现浸润现象,造成肿瘤形状极不规则。上述问题还需进一步改进和探讨。
本研究提出了一种基于自动分割的肝肿瘤三维可视化新方法,所提出的重建方法速度快,可靠性强,肝肿瘤模型显示效果令人满意,可用于肝癌微波热疗计算机辅助手术规划系统的设计中。
[1]Wang Zhili,Liang Ping,Dong Baowei,et al.Prognostic factors and recurrence of small hepatocellular carcinoma after hepatic resection on microwave ablation:a retrospective study[J].J Gastronintest Surg,2008,12:327-337.
[2]任新颖.肿瘤热疗中组织温度场测量方法及关键技术研究[D].北京:北京工业大学,2008.
[3]Campadelli P,CasiraghiE,LombardiG.Automaticliver segmentation from abdominal CT scans[A].In:Proceedings of 14th International conference on Image-Analysis and Processing[C].Piscataway:IEEE,2008.731-736.
[4]Pedrof FF,HuttenlocherDP.Efficientgraph-based image segmentation [J].International Journal of Computer Vision,2004,59(2):167-181.
[5]Cohen I,Gordon D.VS:a surface-based system for topological analysis,quantization and visualization of voxel data [J].Medical Image Analysis,2009,13(2):245-256.
[6]何晖光,田捷,赵明昌,等.基于分割的三维医学图像表面重建算法[J].软件学报,2002,13(2):219-226.
[7]Tian Jie,Xue Jian,Dai Yakang,et al.A novel software platform for medical image processing and analyzing[J].IEEE Transactions on Information Technology in Biomedicine,2008,12(6):800-812.
Research on 3D Visualization Methods for Hepatic Tumor
AI Hai-Ming WU Shui-CaiYANG Chun-Lan*GAO Hong-Jian ZENG Yi
(College of Life Science and Bioengineering,Beijing University of Technology.Beijing 100124,China)
TP391.41
A
0258-8021(2010)04-0504-05
10.3969/j.issn.0258-8021.2010.04.005
2009-12-22,
2010-04-21
北京市自然科学基金资助项目(3072004)
*通讯作者。 E-mail:clyang@bjut.edu.cn
