凝胶微球芯片实验数据分析方法的设计与应用
2010-09-11成黄欢孙啸周国华
李 成黄 欢孙 啸周国华
1)(生物电子学国家重点实验室 东南大学生物医学工程学院,南京 210096)2)(华东生物医学技术研究所,南京 210002)
凝胶微球芯片实验数据分析方法的设计与应用
李 成1,2黄 欢2孙 啸1*周国华2
1)(生物电子学国家重点实验室 东南大学生物医学工程学院,南京 210096)2)(华东生物医学技术研究所,南京 210002)
在一种使用凝胶微球芯片新技术进行基因突变分析的生物学方法中,芯片上红绿两种颜色微球个数的比例代表了正常基因与突变基因的比例。针对微球芯片扫描的图像,发展一种新的方法对图像进行处理和分析,自动识别不同颜色的微球,得到分别代表野生型和突变型的红色微球和绿色微球的各自个数,并开发了相应软件。取单色通道,然后灰度化,使用迭代法和大津法进行二值化的阈值选取,再用中值滤波算法进行噪声杂点消除,最后进行信号点识别及计数。通过对扫描图像的人工计数和软件计数结果的对比,新的方法相对于人工计数结果的偏差率小于10%。实践表明,新方法在处理和分析微球芯片图像时,目标识别结果达到要求,相应软件已在大肠癌早期诊断的生物学研究中应用,提高了分析效率。
图像分析;微球芯片;图像识别
Abstract:In hydrogel bead-array to analyze gene mutation,the proportion of red beads’number to green beads’represents the proportion of mutant genes to wild type genes.Based on the scanning images of a bead array,we developed a new analyzing method to recognize different color beads and count out the number of these beads representing different genes automatically.A corresponding software was also developed to improve the counting efficiency.A single color channel was selected first.Then the color image was transferred into gray one.The iterative and Otsu algorithms were used to calculate the threshold value which was used in binaryzation.Then median filtering was used to get rid of noises.Finally signal points were recognized and numbers were counted automatically.The deviation rate of the automatic results to manual counting results could be controlled less than 10% based on the result comparison of the software and manual work.The recognizing and counting results obtained by the proposed method meet the requirement.The software has been applied in early diagnosis of colon cancer.
Key words:image analysis;bead-array;image identification
引言
近年来,从基因突变角度进行疾病的早期诊断,正成为一个研究热点。然而,由于在癌症早期阶段,突变的基因丰度远低于正常的野生型基因丰度,给癌症的早期诊断带来了一定的难度[1]。到现在为止,已经有一些针对基因突变的检测手段,如DNA测序、数字化PCR等方法,但都存在一定的局限。例如:DNA测序不适用于突变量小于20%的突变样本的检测[2],灵敏度不高;数字化 PCR法则通过将DNA模板稀释分配到小孔中进行扩增[3-4],而每个小孔最多只能扩增一分子野生型或突变型基因,小孔数目的有限性局限了这种方法的灵敏度。如今,一种改进的微乳液 PCR法[5]得到应用,由于这种方法在油包水的微孔中进行单分子扩增,避免了小孔数量引起的局限性,从而大大提高了检测的灵敏度。但是,这种方法要用到流式细胞仪去获得检测结果,因而成本非常昂贵。最近,笔者采用了一种新的微乳液PCR和水凝胶微球芯片技术进行相关基因表达量分析的实验方法[6],这种方法结合了微乳液PCR方法,保证了检测的高灵敏度,同时又通过微球芯片技术,避免使用流式细胞仪分析结果,大大降低了成本。这种新的实验方法对于组织样本中的微量突变响应非常灵敏,同时成本很低,从而为癌症早期诊断的普及提供了一种可能。
这种方法的大概步骤为:首先,采用微球介导的微乳液克隆扩增技术,对样本进行扩增,最终实现在每个微囊中仅包含有一分子模板和一个微球,达到单分子扩增;接着,收集微球,并用水凝胶固定在玻璃芯片上,再通过盖玻片的压力,使微球均匀地平铺在芯片表面,制成单层微球芯片;然后,加入分别与野生型和突变型扩增产物特异性互补的荧光探针进行杂交;最后,扫描成像。
在扫描的微球芯片图像中,包括红绿两种颜色的点,分别代表结合有野生型和突变型DNA片段的微球,红点和绿点的比例就代表了野生型和突变型基因的比例,图像中这些代表微球的点统称为信号点。本研究的目的即通过图像处理的各种算法,自动识别图中红绿两种颜色的微球,并分别计数。计数前先要进行必要的预处理,图1就是经过图像预处理后仅显示单色信号点的灰度图。从中可见,凝胶固定的微球随机分布,大部分代表微球的信号点孤立且边缘清晰可见,这部分的微球识别较为容易。但是还有相当数目的微球并没有如实验所设想的成为完全的单层微球,而是出现部分叠加,有些甚至叠加成团,导致图像上的信号点出现粘连,这部分微球的识别和计数就成为需要主要解决的问题。另外,实验中选用的是直径为34 μm的琼脂糖微球作为引物结合载体,但由于实验过程中不可避免的误差,导致图像上代表微球的信号点大小不一。图像上还可以看到一些杂点或干扰条纹,由实验过程中不可避免的因素造成。所以,在图像识别和计数过程中,也应尽量去除这部分干扰。

图1 凝胶微球芯片的单色灰度图像Fig.1 Single-color grayscale image of a hydrogel beads array
1 方法设计
根据以上分析的实验图像特点,分预处理和目标识别两个模块进行图像处理,流程如图2所示。

图2 方法流程Fig.2 Flowchart of the method
1.1 预处理
1.1.1 颜色通道选择
因为图像上信号点主要有红色和绿色,为避免相互影响,应分别计数,先使图像呈单色显示。分析实验图像,为荧光扫描仪扫描后输出的24位的bmp格式,红、绿、蓝三种颜色通道各占8位,色阶值范围0~255。所以,若要单色显示,只要使另外两种颜色通道的色阶值为0即可。
1.1.2 灰度化
图像上主要分背景和信号点,预处理的目的是提取信号点并剔除噪声,转化为二值图像。在二值化以前,需要分析图像单色通道的色阶值分布特征,故先转化为灰度图,为下一步二值化的分析做好准备。将图像中的红、绿、蓝各色阶值统一用之前单色通道的色阶值代替,即可转化为灰度图。
1.1.3 二值化
将图像上的灰度值置为0或255,这样以后的图像不再涉及像素的多级值,使处理变得简单,而且数据的处理和压缩量较小。这里,把背景设为255(白色),而信号点的值设为0(黑色),并采用阈值分割,所有灰度大于或等于阈值的像素被判定为背景,其余像素为信号点内的有效像素。该方法的设计难点在于如何进行粘连信号点的分割,而阈值选择的恰当与否对分割的效果起着决定性的作用。笔者参考了一些常见的阈值分割算法,如双峰法、迭代法、大津法[7],根据图像特征,选择迭代法和大津法分别进行分割阈值的计算,并绘出了图像的灰度直方图,最终由用户在参考多种算法结果的基础上决定分割阈值。
1)迭代法选择阈值是基于逼近的思想,其实现步骤如下。
步骤1:求出图像的最大灰度值和最小灰度值,记为Zmax和 Zmin,令初始阈值为
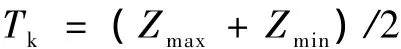
步骤2:根据阈值Tk将图像像素分为第1族和第2族,分别求出两者的平均灰度值Z1和Z2。
步骤3:求出新阈值Tk+1=(Z1+Z2)/2。
步骤4:若指定一个极小值ε,有|Tk+1-Tk|<ε,若逼近之值基本满足要求,则Tk+1即为最后的迭代结果,否则令Tk=Tk+1,重新执行上面的计算过程,直到|Tk+1-Tk|<ε。
2)大津法属于最大类间方差法,是自适应计算单阈值的简单、高效算法。其原理是对目标灰度图像的直方图进行分析,把阈值作为分界线将直方图分成两个部分,并从小到大不断移动,每次移动后就比较新分割的两部分到分界线的距离,当阈值使分界线在两部分之间的距离最大时,此时的阈值即所求阈值。对一幅灰度图用公式来表达,设t为分割阈值,前景点数占图像总像素的比例为w0,平均灰度为u0,背景点数为w1,平均灰度为u1。图像的总平均灰度为
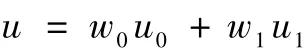
从最小灰度值到最大灰度值遍历t,当t使函数G=w0(u0-u)2+w1(u1-u)2的值最大时,t即为分割的最佳阈值。
1.1.4 中值滤波
该算法是基于排序统计理论的一种能有效抑制噪声的非线性信号处理技术,基本原理是把数字图像或数字序列中一点的值用该点的一个邻域中各点值的中值代替,让周围的像素值接近真实值,从而消除孤立的噪声点。在本软件中,将窗口大小设为3像素×3像素,进行逐个像素扫描,当前像素点的值则被以其为中心的9个像素点的中间值取代,从而一些独立的噪声点被去除,而有效信号点的边缘被保护。
1.2 信号点识别
完成预处理后,下面就开始识别信号点。如果信号点是一个个分开的无粘连连通域,对每个信号点内的像素递归寻找邻接的有信号值的像素,通过分析递归返回的变量,就可得到微球的个数和面积。但实际图像中的部分信号点是粘连在一起的,用上述方法无法准确统计个数。分析图像特征,由于选取的都是直径34 μm的微球,所以信号点大小比较均匀,使用一定大小的圆点标识信号点,进而转化为求圆点的个数,分割结果较好。
1.2.1 小面积区域标识
由于同一次实验选取的微球大小基本一致,在预处理过程中加入了统计分析功能,对所有的单连通区域求取平均面积供用户参考,再由用户确定一个面积参数,对像素数目小于此参数的单连通域都标识为单个微球,认为是一个独立且有效的信号点。
1.2.2 粘连微球中心定位
假设微球在图像中的形状都近似圆形,由于与荧光染料特异性结合的DNA片段在微球上的扩增一般都假设均匀,所以反映在平面图像上,越中心的位置染料聚集密度越高,从而信号强度应当越高。据此分析,同样建立了一个3像素×3像素的窗口进行逐个像素扫描,并挑选连通区域内信号值最高的像素点为中心点,循环找到信号值最高的像素,即认为是信号点的拟定中心。之后再使用圆形与当前连通区域进行形状匹配,不断微调圆心位置,直到匹配程度最高。
1.2.3 信号点标识
在假定的信号点中心处尝试绘制标识圆,且要满足两个判断条件:一是在所要绘制的标识圆区域内,均有信号值;二是对当前信号点区域绘制标识圆时,不与任意先前的标识圆发生粘连。这样绘制的每一个标识圆将代表一个有效的信号点,且将图像上粘连的信号点数目转化为无粘连的标识圆的数目。图3为图1中信号点的识别结果,其中空心黑圈代表识别出的标识圆边缘。可以看到,有效信号点几乎全部成功识别,且标识圆完全没有叠加,进而可以实现准确计数。

图3 信号点的识别结果Fig.3 The recognizing result of signal spots in the example image
1.2.4 标识圆计数
使用成熟的单连通域递归算法,即可完成计数任务。当逐行扫到有信号的像素时,开始8个方向的递归寻找,只要和此像素连通的其他信号像素均在辅助矩阵中进行标注。下面继续扫描信号像素点进行计数时,就不会将之前标注过的像素点计算在内,从而实现了标识圆的准确计数。
图3中标识符的自动计数结果为947,而对原图的人工计数结果为955。经过实验人员的验证,这两种方法所产生的结果差异是满足实验分析要求的。
1.2.5 信号点识别结果修正
由于微球的大小并不完全一样,而且实验过程中可能发生各种操作误差、荧光染料杂交、信号点粘连等情况,上面的算法识别结果不一定完全正确,因而适当的人工辅助识别修正是必要的。一些被错误绘制的标识符应当可以被操作者去除,而另一些没有被成功识别的信号点可以被操作者增绘标识符。如果操作人员要得到更准确的结果,可以修正后重新进行计数。
2 应用结果
采用水凝胶微球芯片技术,进行了粪便脱落细胞中大肠癌相关基因表达量检测的研究。大肠癌是最常见的消化道恶性肿瘤,筛查是早期发现大肠癌和提高大肠癌患者生存率的关键。目前用于普查筛选方法主要是结肠镜和大便隐血试验,而这两种方法对大肠癌的早期诊断均不十分理想;如今国际上粪便脱落细胞的检测为一种无创性检查,对大肠癌的诊断具有重要意义。笔者采用基于微球的微乳液“克隆”PCR法,同时扩增待测大肠癌相关基因COX2(或c-myc)的cDNA和一个看家基因betaactin的cDNA,并通过双色荧光技术,分别为扩增后的微球标记上红色或绿色,用以标识微球上被扩增的是待测癌症基因还是看家基因;再用水凝胶法,将这些克隆PCR产物(即表面包埋有DNA分子的微球)制备成微球芯片。由于看家基因beta-actin的表达量基本不变,故红色和绿色微球个数的比例高低表示待测基因的相对表达量,从而实现了一种新的大肠癌早期诊断的实验方法。这种方法产生了大量的结果图像,用人工进行计数显然不可能。所以,为了验证自动计数方法的实用性,从上述研究中挑选了10幅图像进行人工计数和自动计数的对比。本研究认为,经过人工识别修正后的再次计数为人工计数结果,且认为是准确结果,设为M;而原图不经修正的第一次自动识别结果可认为是自动计数结果,设为A。自动计数相对于人工计数的偏差率W为

同时,也计算了平均偏差率,有

具体结果见表1。
对10幅典型图像的计数结果的平均偏差率为3.94%。观察对比结果,第2组绿球(12.5%)和第4组绿球(20.7%)识别的偏差率较大。
3 讨论
3.1 关于偏差率大的原因
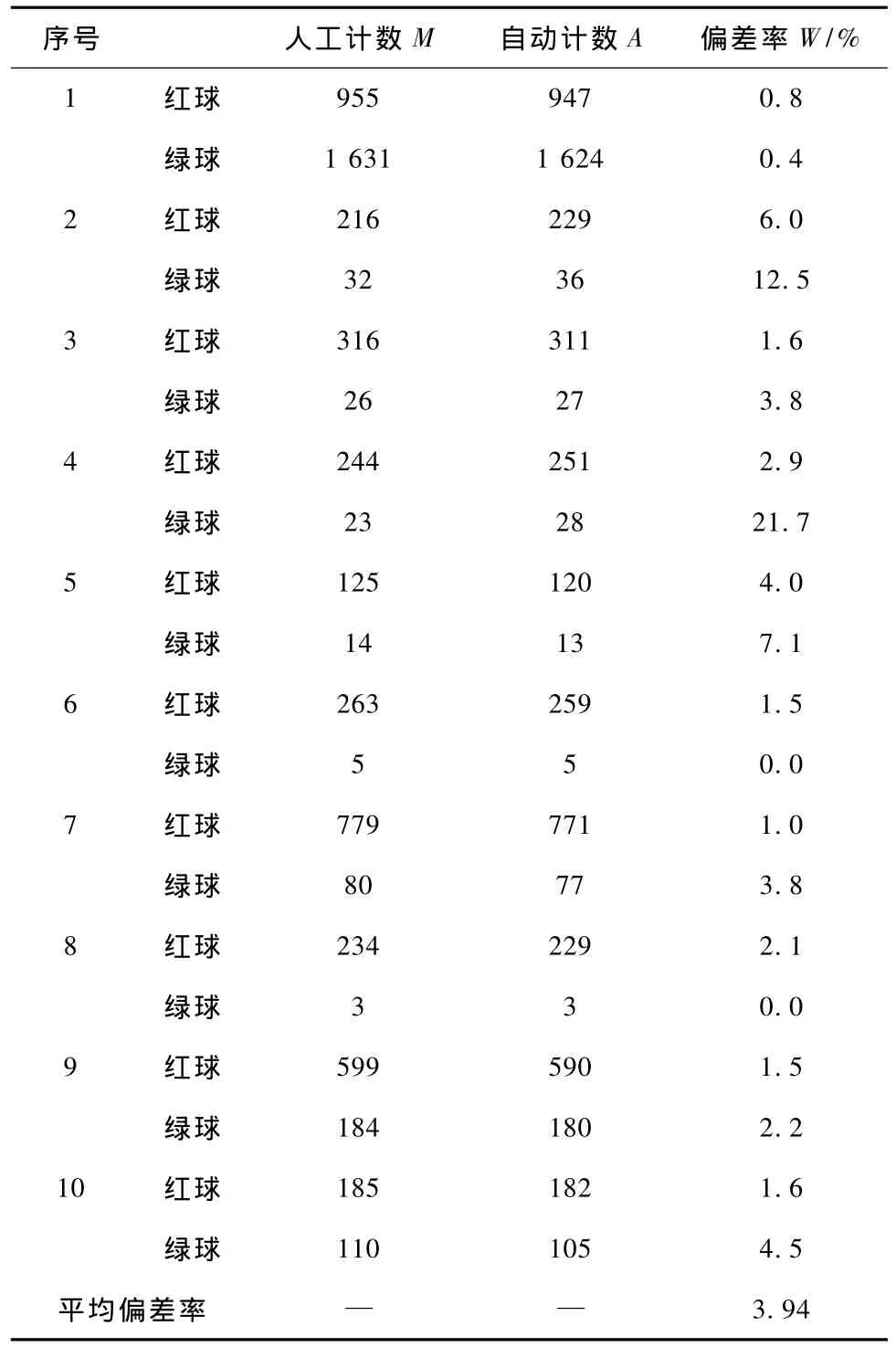
表1 人工和自动计数结果对比Tab.1 Comparison of manual and auto-counting results
对于第二组绿球和第四组绿球识别的偏差率较大的情况,主要是因为相应的扫描图像中出现了与其他图像相比比例较高的混色球。所谓混色球,是指实验时同一个微球上同时扩增了正常和突变的基因片段,没能达到真正的单分子扩增,导致两种颜色的荧光探针同时发光,人工识别时就出现漏查。在这种情况下,自动计数在计数绿球时将混色球计入的选择应当认为是正确的。通过图4中的示意,对第4组数据进行具体的原因分析。观察图4(b)中绿球的识别后图,右上角3个箭头所指的微球成功识别,但在原图中观察,对应微球均为混色球,并且由于红色强度较高,掩盖了较弱的绿色,导致人工识别时漏查3个绿球;同时,灰度图中左下方箭头所指的绿球发光面积较大,造成识别图中多标识了两个信号点,因而多查了2个绿球另外,而发光面积与实验所选微球是否大小不均、微球杂交受污染程度都会影响偏差率。由于此图本身的绿球数目比较少,所以偏差率超过了20%,大大超过了平均偏差率。
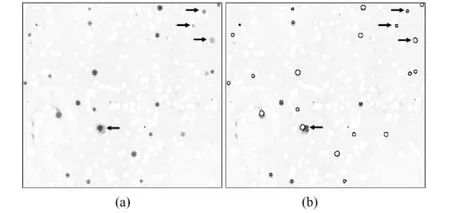
图4 较大差异结果组对应图像。(a)绿球对应灰度图;(b)识别后图Fig.4 Corresponding imagesofthedatasetwith comparatively big difference.(a)Corresponding grayscale image of green beads;(b)Image after recognizing
通过对其他各组数据的分析,偏差也大都与实验图像的质量相关,表明实验过程及扫描所成图像的质量是本方法能够正确应用的基础。另外,当实验中每次采用的微球大小不均导致图像中信号点大小不一时,面积较大的信号点内有可能满足同时绘制两个标识圆的条件,导致多查;面积较小的信号点又有可能满足不了半径确定的标识圆的绘制条件(即绘制范围内都有信号值,同时又不能与其他标识圆发生粘连),导致漏查。还有其他一些因素,如荧光染色不均匀、一个微球杂合了两种颜色的荧光染料、信号点粘连程度较深等,都会对自动计数方法的偏差率产生影响。但正如10组对比数据和图2的识别结果所示,其中占大多数的孤立信号点都可以准确识别,而数量其次的粘连程度不深的信号点群的识别效果也较好,只有极少数的粘连程度较深的信号点识别有误,因而所建立的自动计数方法的结果已经比较准确,可满足实验过程无大差错、扫描图像质量较好的结果分析要求。
另外,根据基因COX-2的表达量进行大肠癌早期诊断,有一个相对于看家基因比例的判断阈值,若COX-2基因与看家基因的表达量比例在此阈值上下10%的范围内,都建议去做更精准的医学检验。在本方法中,计数的平均偏差率达到3.94%,因而在凝胶微球芯片用于大肠癌早期诊断的临床应用中,需要扩大要求进行医学检验的范围,即两者表达量的比例在阈值上下15%的范围建议进行进一步的医学检验,这样即可满足初步筛查的要求。
3.2 关于阈值分割算法
由于阈值选择的适当与否对分割效果起到决定性作用,所以这里对迭代法和大津法这两种阈值分割算法进行专门讨论。迭代法所得阈值对图像的分割效果较好,能够区分出图像的前景和背景的主要区域,但对图像的细微区域区分度不高;而大津法适应性强,对图像的分割质量具有一定保障,是一种稳定和通用的阈值分割算法,但在两群物体的灰度差不明显的情况下,会丢失图像的一些整体信息[7]。为了验证两种方法对计数结果的影响,针对上面的10幅例图,对比了两种方法分割后的计数结果,如表2所示。
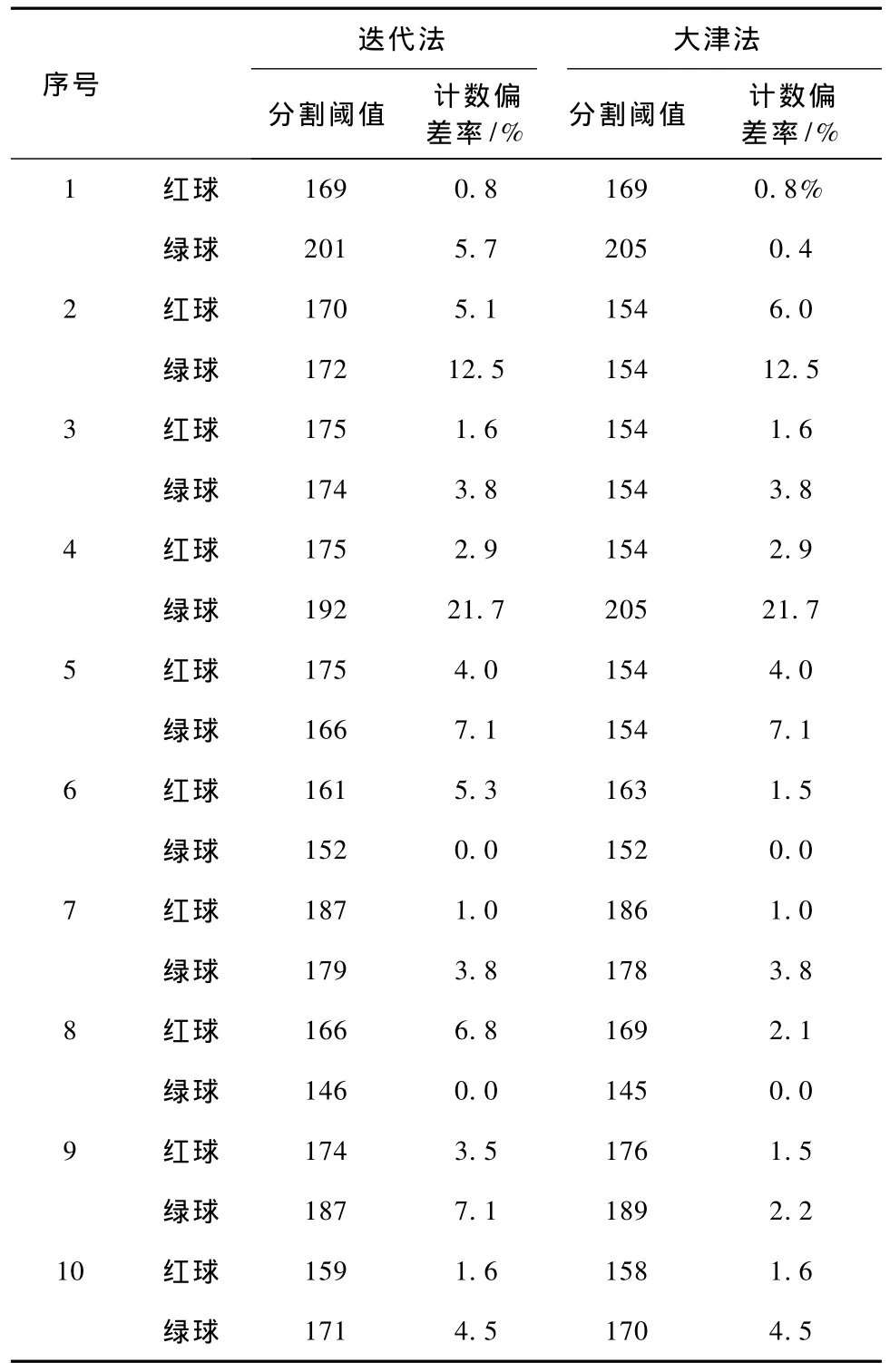
表2 迭代法和大津法计算阈值分割结果对比Tab.2 Comparison of segmentation results based on threshold values calculated by iterative and Otsu algorithms
分析上面的结果对比,可知这两种阈值分割算法的应用主要有两种情况。
1)在质量较好的图像中,由于目标和背景区别显著,所以两种算法分割后的计数结果差别不大。即使两种算法计算的阈值差别较大,计数结果仍然相近,如2组绿球、3组红球、3组绿球、5组红球等。在这种情况下,两种阈值分割算法的效果都满足要求。
2)对于目标和背景差别不大的图像,两种算法分割后计数的结果差别比较大。即使两者计算的阈值相近,但由于部分信号点强度低于计算的阈值,导致部分信号点丢失,计数结果偏低,如1组绿球、6组红球、8组红球、9组红球等。在这种情况下,大津法则体现了其优势,计算的阈值能够分割出更多的有效信号点,从而更加接近真实值。
分析质量较好的图像时,两种阈值分割算法的效果都较好,区别不大;而分析质量较差、目标和背景区别不大的图像时,大津法的分割效果更好。
4 结论
针对凝胶微球芯片图像,本研究提出了一种实用且高效的微球自动计数方法。此方法执行速度快,智能化程度高,大大提高了微球计数的效率,并已成功用于利用凝胶微球芯片进行大肠癌相关基因表达量的研究中,为微球芯片图像分析提供了一种新的思路。
[1]Lleonart ME,Cajal SR,Groopman JD,et al.Sensitive and specific Detection of K-ras mutations in colon tumors by short oligonucleotide mass analysis [J].Nucleic Acids Res,2004,32:e53.
[2]Bar-Eli M,Ahuja H,Gonzalez-Cadavid N,et al.Analysis of NRAS exon-1 mutations in myelodysplastic syndromes by polymerase chain reaction and direct sequencing[J].Blood,1989,73,281-283.
[3]Seung MD,Giovanni T,Constance J,et al.Detecting colorectal cancer in stool with the use of multiple genetic targets[J].Cancer Inst,2001,93,858-865.
[4]Vogelstein B,Kinzler KW.Digital PCR[J].Proc Natl Acad Sci USA,1999,96:9236-9241.
[5]Margulies M,Egholm M,Altman W,et al.Genome sequencing in microfabricated high-density picolitre reactors[J].Nature,2005,437,376-380.
[6]Huang Huan,Qi Zongtai,Deng Lili,et al.Highly sensitive mutation detection based on digital amplification coupled with hydrogel bead-array[J].Chem Commun(Camb),2009,4094-4096.
[7]蒋先刚.数字图像处理工程软件设计[M].北京:中国水利水电出版社,2006.
Design and Application of the Method for Analyzing Experimental Data from Hydrogel Bead-Array
LI Cheng1,2HUANG Huan2SUN Xiao1*ZHOU Guohua2
1(State Key Laboratory of Bioelectronics,Southeast University,Nanjing 210096,China)2(Huadong Research Institute for Medicine and Biotechnics,Nanjing 210002,China)
R318
A
0258-8021(2010)04-0492-06
10.3969/j.issn.0258-8021.2010.04.003
2010-03-01,
2010-04-23
国家自然科学基金资助项目(30470454);南京科学技术发展项目支持(200801087)
*通讯作者。 E-mail:xsun@seu.edu.cn
