肠球菌为主要菌群的腹泻标本的16S rRNA与PFGE分析*
2010-06-07周永运许彦梅崔志刚赵爱兰叶长芸徐建国
周永运,许彦梅,崔志刚,金 东,赵爱兰,叶长芸,徐建国
肠球菌是院内常见条件致病菌,腹泻标本有时也可分离到该菌,经常被认为是菌群失调的结果。为了解该类标本分离肠球菌菌株的克隆特征,进行了16S rRNA和PFGE方法以进一步分析。
1 材料及方法
1.1 样本特征及来源 与山西省临床检验中心合作,从太原市某医院采集儿童腹泻粪便标本,其中4例腹泻病患者的标本初步培养以肠球菌为优势菌群,腹泻儿童的病历材料见表1。
1.2 菌株来源及分离培养 所分析菌株包括以上4份腹泻病人粪便标本在厌氧和CO2环境中分离的各50株左右屎肠球菌和个别粪肠球菌;4个健康儿童粪便标本厌氧和CO2环境中分离的各10株肠球菌。取0.01mL粪便悬液,均匀涂布整个血平皿。在厌氧和 5%CO2两种条件下,用厌氧血平皿置37℃温箱培养24h,接种肠球菌的选择性培养基(含七叶苷的脑心培养基础),每份标本逐个挑选50个黑色菌落左右。
1.3 主要试剂 AmpliTaq Gold DNA聚合酶购自ABI公司;dNTP购于Promega公司;Sma I酶和DNA分子量marker购自Takara公司;DNA回收试剂盒Qiaquick®Gel Extraction kit购自Qiagen公司的;细菌基因组DNA提取试剂盒购自Promega公司;触酶、氧化酶购自梅里埃公司;溶菌酶购自Invitrogen公司。
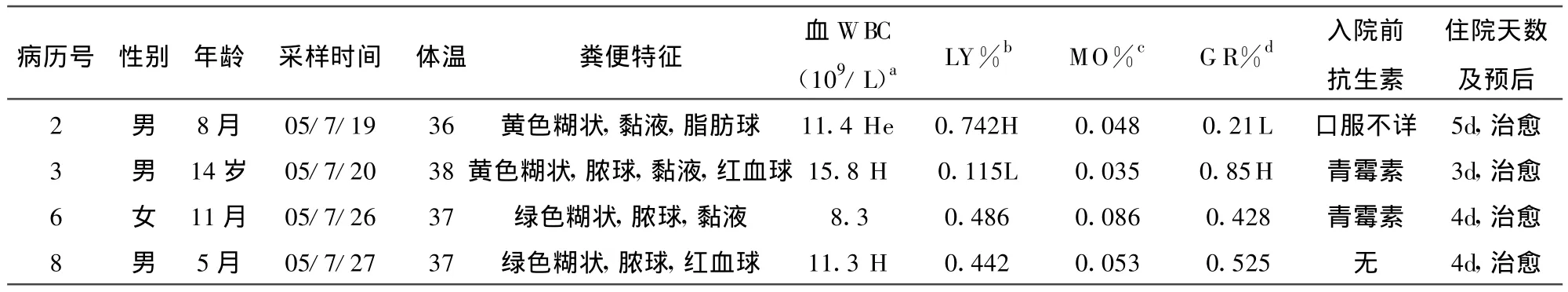
表1 4个腹泻儿童的病历材料Table 1 Medical record of 4 diarrhea children
1.4 主要仪器和设备 PCR仪为MG公司的PTC-200;凝胶成像系统为Bio-Rad公司Gel Doc XR型凝胶成像仪;细胞培养箱为Thermo forma;厌氧培养罐购自Oxoid公司;生化鉴定仪为DATE BEHRING公司Microscan WalkAway40 S1;PFGE为Bio-Rad公司 CHIF-Mappper脉冲场凝胶电泳仪。
1.5 16S rRNA基因序列分析方法
1.5.1 临床标本的采集和处理 无菌采集腹泻病人的粪便标本、称重,加10倍体积磷酸盐缓冲液(0.02mol/L)重悬。4℃,12 000r/min离心 2min,弃上清。然后加等体积的磷酸盐缓冲液重悬离心后得到的细胞沉淀可立即进行DNA提取或置-70℃冰箱冷冻保存。
1.5.2 总DNA的提取 标本中总DNA的提取参照已有的方法〔1〕。
1.5.3 16S rRNA基因保守区的扩增 使用16S rRNA通用引物〔2,3〕u9:5′-agagtttgatcytggyty ag-3′,907r:5'-ccgtcaattcmtttragttt-3'对标 本 中 总DNA进行扩增:反应体系为20μL,包括 10×buffer 2μL,2.5mmol/L MgCl2 2μ l,dNTP 1.6μL,上引物0.5μL,下引物 0.5μL,Gold 酶 0.2μL(1U),DNA模板 1μL,BSA(1mg/mL)2μL,三蒸水 10.2μL。扩增条件:预变性 95℃12min,后续 95℃50s,52℃30s,72℃1min30s,20个循环,72℃延伸10min。
1.5.4 PCR产物的回收及与载体的连接、转化和克隆子的筛选 PCR产物的纯化、回收按照试剂操作手册进行。纯化产物与T载体连接,以CaCl2法转化感受态细胞DH5α,进行蓝白斑筛选,每份样本选择约80个克隆子分别送上海生工生物工程公司、北京华大基因测序有限公司、北京擎科生物公司进行平行3次测序。测序仪为ABI公司的3730型生物测序仪。
1.5.5 序列的比对与统计分析 测得的序列首先使用Chromas软件观察测序质量,以CATGCAA作为起始序列,CCGGCTAA作为终止序列进行序列截取,将截取的序列用软件blast(http://www.ncbi.nlm.nih.gov/BLAST/Blast)在 NCBI和本室建立的核糖体共有序列数据库(www.RCSD.com)进行比对,比对的结果根据序列的相似性程度百分比进行分类统计分析。
1.6 PFGE 每份腹泻标本随机选择20株屎肠球菌,正常标本Y随机选择10株屎肠球菌进行PFGE分析。PFGE方法参照美国CDC李斯特菌的标准操作略有改动〔4〕。从血平皿上沾取适量细菌,均匀悬浊于TE中,用 BioMerieux Vitek colorimeter测其OD值并调整至6.1~6.3。取240μL细菌悬浊液于相应的 1.5mL Eppendorf管中,加入 60μL溶菌酶溶液,置于37℃水浴中孵育10min。配制细胞裂解液(CLB)时每5mL细胞裂解液加入37.5μL蛋白酶K,然后颠倒混匀。选用 SmaI(50U)酶切,37℃水浴中孵育至少2h。电泳预设温度在14℃,电泳参数为:初始脉冲时间2s;终止脉冲时间18s,电泳时间18h。图象用Gel Doc DR采集,采用Bionumarix软件分析。
2 结 果
2.1 4位腹泻患者粪便标本的16S rRNA基因序列分析 本研究检测了4份患儿粪便标本,每份标本检测3次。2号患儿标本总共检出6个种菌属,以 Enterococcus f aecium、Streptococcus sanguis为主,分别占总克隆量的68.6%(208/303)和25.1%(76/303),3次实验均有检出;另外 Rothia mucilaginosa也是3次实验均有检出,但数量较少,小于克隆量的 5%(13/303),Escherichia.coli、Actinomycesodontolyticus、Granulicatella三个菌属在不同的实验中有检出,小于总克隆的2%(3/303,3/303,1/303)。
3号患儿标本总共检出4个种属,含量较多的Enterococcus f aecium和Bacteroides sp 3次实验均有检出,分别占85.3%(250/293)和13.7%(40/13.7);Rothia mucilaginosa、Actinomyces odontolyticus数量较少,小于克隆量的 2%(2/293,1/293)。
6号患儿标本总共检出4个种属,含量较多的Enterococcus faecium、Streptococcus sp,3次实验均有检出,分别占总克隆的 95.9%(231/241)和2.1%(5/241);Rothia mucilaginosa、Escherichia.coli数量较少,小于克隆量的2%(3/241,2/241)。
8号标本的结果见表5。8号标本总共检出6个种属,含量较多的Enterococcus f aecium、Rothia mucilaginosa、Escherichia coli、Enterococcus f aecalis 3次实验均有检出,分别占总克隆的 91.1%(246/270)、2.2%(6/270)、3.0%(8/270)和 2.6%(7/270);Ochrobactrum lupini、Actinomyces odontolyticus数量较少,小于克隆量的2%(2/270,1/270)。
2.2 腹泻标本与正常标本的屎肠球菌分离菌株的PFGE图谱比对 分离健康儿童粪便标本:2个不满周岁的儿童粪便(X,Z),1个5岁儿童的粪便(Y),1个8岁儿童的粪便(K)。用选择性培养基结晶紫胆盐七叶苷筛选,每份在厌氧和CO2培养条件下共随机挑选10个菌。
腹泻患儿屎肠球菌分离菌株的染色体经过Sma I酶切后的PFGE图谱见图1。PFGE结果显示,2、3、6号标本中以一种 PFGE带型为主,在20个菌株的PFGE带型中均仅有一个不同带型,分别为2.36C、3.79C、6.2N,8号标本中存在5个带型,每个带型在 20个菌株中分别有:9、5、3、2、1个菌。Bionumerics软件的聚类结果与正常健康儿童粪便标本照的对PFGE结果一起显示,见图1。
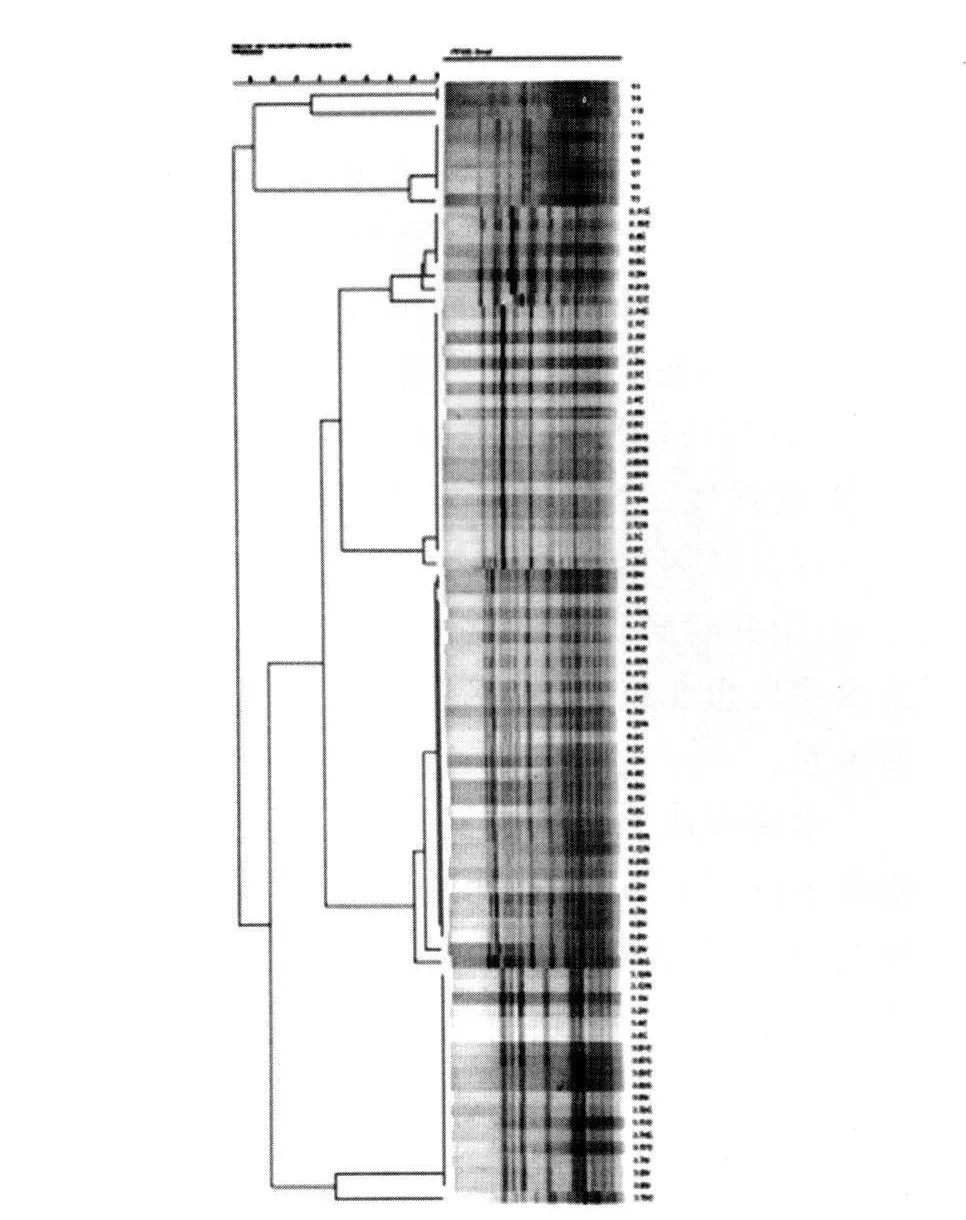
图1 腹泻标本与正常对照标本屎肠球菌的PFGE聚类分析结果Fig.1 PFGE dendrogram showing clustering of PFGE types recovered from diarrhea and normal specimens
3 讨 论
进行肠道微生态分析,使用传统方法进行病原菌的分离和鉴定,需要获得纯的培养物,但是能够获得纯培养的细菌比例少,还有大量的细菌不能得到培养和鉴定。仅仅依靠传统方法难以进行菌群分析,目前认为,以16S rRNA基因序列分析结合表型分析的鉴定方法,是细菌学检测和鉴定的发展方向〔5〕。PFGE是屎肠球菌流行病分型的可选方法,较其他方法分辨力更高,所有的菌株可以分型并且重复性好〔6〕。
本研究利用培养和16S rRNA序列分析方法发现,4个腹泻标本分离到的菌株均以屎肠球菌为主,而4个正常的健康儿童的粪便标本中仅Y一个以屎肠球菌为主。腹泻儿童的16S rRNA菌群分析显示菌群比例严重失调,常驻菌大大减少而肠球菌比例上升至68%以上,本结论与国外学者用DGGE电泳调查腹泻病人和无症状的人群的粪便菌群构成发现腹泻标本更多表现出细菌种类减少和某种细菌的过度生长的现象一致〔7〕。在荷兰调查的所有624位住院病人和200位住社区中病人的粪便标本中,屎肠球菌的分离率分别为43%和32%,明显低于本研究具有腹泻特征标本的分离率和百分比〔8〕。
利用SmaI酶切后进行PFGE电泳,聚类分析后发现腹泻标本中的除8号腹泻标本含有5个PFGE聚类克隆外,屎肠球菌中 2、3、6 PFGE聚类单一,表现为克隆性聚类克隆。正常的健康儿童Y标本,表现为屎肠球菌非克隆性,含有4种PFGE聚类克隆;其他健康儿童含有粪肠球菌或多种肠球菌,种类更加多样。
肠球菌是社区感染和院内感染的主要病原菌,可引发心内膜炎,菌血症,尿路感染,新生儿感染,中枢神经,腹腔和盆腔感染等〔9〕。美国国家医院感染监测系统(National Nosocomial Infections Surveillance,NISS)已将其列为医院感染的第2位病原菌〔10〕。但是国外尚无肠球菌引起腹泻的临床报道和流行病学资料。在我国部分省市陆续有肠球菌引发食物中毒的报道。2001年8月广东省东莞市常平镇某公司食堂发生了一起集体食物中毒,实验室证实为一起粪肠球菌引起的食物中毒〔11〕。本实验采用16S rRNA基因序列分析发现,肠球菌是优势菌群(>68%),次要菌群包括罗氏菌,链球菌,大肠杆菌,放线菌。这4份标本的菌群构成非常相似,没有发现其他可疑腹泻细菌。从临床治疗的效果来看,应用针对肠球菌敏感的抗生素后加上补液等辅助治疗后患儿痊愈,提示肠球菌可能与腹泻有着密切关系。
本研究从16S rRNA和PFGE的角度对腹泻粪便标本进行了分析,得到腹泻儿童肠球菌分布的初步规律,其致病性和相关机理还待今后实验范围的进一步扩大和研究的深入。
〔1〕丽鸿,赵勇,陈明杰,等.堆肥中微生物总DNA的高效提取〔J〕.微生物学报,2006,46:162-165.
〔2〕M cInnery JO,Wilkinson M,Patching JW,et al.Recovery and phylogenetic analy sis of novel archaeal rRNA sequences from deep-sea deposit feeder〔J〕.Appl Environ Microbiol,1995,61:1646-1648.
〔3〕Watanabe K,Kodama Y,Harayama S.Design and evaluation of PCR primers to amplify 16S ribosomal DNA fragments used for community fingerprinting〔J〕.J Microbiol Methods,2001,44:253-262.
〔4〕http://www.cdc.gov/PULSENET/protocols.htm.
〔5〕Weisburg WG,Barns SM,Pelletier DA,et al.16S ribosomal DNA amplification for phylogenetic study〔J〕.J Bacteriol,1991,173:697-703.
〔6〕Werner G,Klare I,Witte W.T he current MLVA typing scheme for Enterococcus f aecium is less discriminatory than M LST and PFGE for epidemic-virulent,hospital-adapted clonal types〔J〕.BMC Microbiol,2007,7:28.
〔7〕Mai V,Braden CR,Heckendorf J,et al.Monitoring of Stool Microbiota in subjects with diarrhea indicates distortions in composition〔J〕.J Clin Microbiol,2006,44(12):4550-4552.
〔8〕Endtz HP,van den Braak N,van Belkum A,et al.Fecal carriage of vancomycin-resistant enterococci in hospitalized patients and those living in the community in The Netherlands〔J〕.J Clin Microbiol,1997,35(12):3026-31.
〔9〕Murray BE.The life and times of the Enterococcus〔J〕.Clin MicrMicrobiol Rev,1990,3:46-65.
〔10〕Richards MJ,Edwards J R,Culver DH,et al.Nosocomial infections in combined medical2surgical intensive care units in the United States〔J〕.Infect Control Hosp Epidemiol,2000,21(8):510-515.
〔11〕单金华,袁钦华.一起粪肠球菌食物中毒的调查〔J〕.实用预防医学,2001,8:394.
