猪源粪肠球菌和屎肠球菌多重PCR快速鉴定方法的建立*
2010-03-07王亚宾陈丽颖段志刚崔保安
王亚宾,陈丽颖,胡 慧,段志刚,崔保安
(河南农业大学牧医工程学院,河南郑州450002)
肠球菌为革兰阳性球菌,是一种重要的条件致病菌[1],存在于肉品中的肠球菌,还是人食物中毒的主要原因[2-3]。近年来,肠球菌对人和动物感染不断增加,随着肠球菌耐药菌株的日益增多,感染后治疗也越来越困难,由于不同的肠球菌耐药性不同,肠球菌种的鉴定就显得尤为必要[4-5]。感染猪的肠球菌主要是粪肠球菌和屎肠球菌,它们也是医院内感染和各种环境介质中的优势菌群[6],快速、准确鉴定粪肠球菌和屎肠球菌对于控制肠球菌病具有重要意义。
目前,肠球菌的鉴定主要依靠传统的生化试验进行表型鉴定,这需要大量的时间进行生化管的准备和判断结果,一些商品化鉴定试剂盒API Rapid ID 32 Srep、BBL Crystal和Vitek-32等,虽能够快速将肠球菌鉴定到种的水平,检测结果也能在24 h后得到有效判读,但其鉴定的可靠性受到质疑[7]。基于PCR方法为肠球菌鉴定提供了可靠的手段,16 S rRNA、热休克蛋白60(HSP60)、延长因子EF-tu(tuf基因)、ddL和sodA等基因都可分别在种属水平上鉴定肠球菌,但同时能够在种属水平上对感染猪的粪肠球菌和屎肠球菌进行鉴定还没有报道。笔者利用NCBI公布的16 S rRNA基因序列设计属特异性引物,并根据文献[8]报道的sodA基因种特异性引物建立同时测定粪肠球菌和屎肠球菌多重PCR方法,以图为基层实验室肠球菌病诊断提供基础。
1 材料与方法
1.1 菌种
14株试验菌株中ATCC29212、CMCC(B)32223粪肠球菌参考菌株由广东环凯微生物科技有限公司提供;其余菌株(见表1)均为本室鉴定菌株,其中粪肠球菌(E.faecalis)和屎肠球菌(E.faecium)感染猪的临床菌株,这些菌株16 S rRNA基因序列均被NCBI收录。另外53株分离株(L1-L53)选自2006年—2008年期间分离自河南洛阳、济源、许昌等地送检病猪的肝、脾、肾猪的临床感染菌株(30株)、粪便分离菌株(13株)和郑州市零售鲜猪肉分离株(10株)。2株E.sanguinicola为分离于零售鲜猪肉菌株(Genbank号分别是FJ378700.1和FJ378705.1),链球菌(CVCC562)葡萄球菌(ATCC25923)为本室保藏菌株。这些菌株均于50%甘油中在-70℃下由本实验室保存。试验时,将其在BHI肉汤中活化,再划线接种到兔鲜血琼脂平板上,37℃、24 h培养后使用。
1.2 主要仪器和试剂
3K30型冷冻离心机,sigma公司生产;DYY-111-6B型电泳仪,北京六一仪器厂;PTC-200型PCR仪,MJ RESEARCH公司生产。2×PCR TaqMix(北京美莱博医学科技有限公司);Protein-ase K(Amresco),琼脂糖(西班牙进口分装),DNA Marker宝生物工程(大连)有限公司;酚∶氯仿∶异戊醇(25∶24∶1)(瑞兴科技有限公司);脑-心浸萃液态培养基(BHI)为广东环凯微生物科技有限公司产品。其他试剂均为国产分析纯产品。
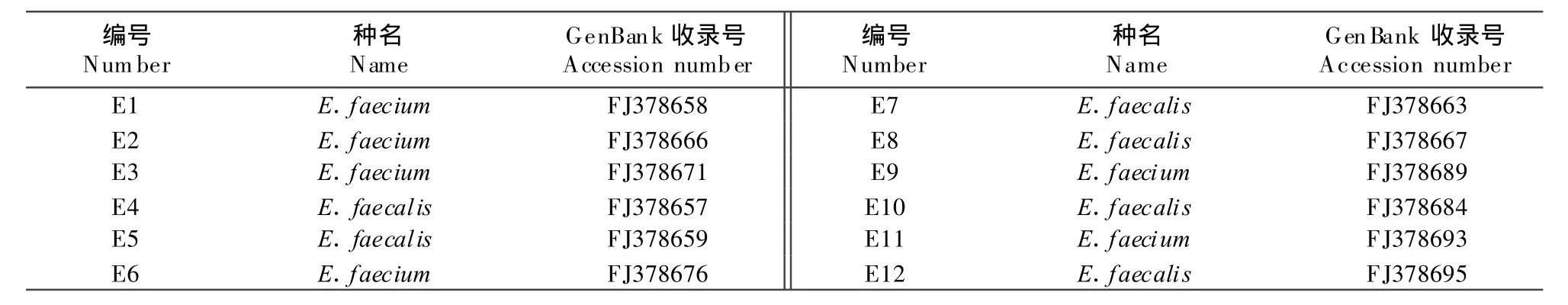
表1 试验菌株Table 1 Stains for test
1.3 引物选择与合成
属特异性引物参考GenBank公布的肠球菌16 S rRNA基因序列进行设计,种特异性引物参照文献[8]报道的粪肠球菌和屎肠球菌引物序列。所有引物由上海博尚生物技术有限公司合成。引物序列及预计扩增长度见表2。
1.5 多重PCR方法的建立
1.5.1 模板制备 将肠球菌、葡萄球菌和链球菌分别接种于BHI液体培养基,37℃摇床过夜培养,收集菌体,按照Messick报道[9]的方法进行常规酚/氯仿抽提,—20℃保存备用。
1.5.2 PCR扩增 反应体系为25 μ L,其中2×PCR TaqMix 8 μ L(0.2 U Taq DNA Polymerase/μ L,400 μ mol/L dNTP each,20 mmol/L pH 8.7 Tris-HCl,100 mmol/L KCl,3 mmol/L MgCl2);正向、反向引物(20 pmol/μ L)各1 μ L;模板DNA 2 μ L。最后补充双蒸去离子水(9 μ L)至25 μ L。扩增程序如下:95℃预变性5 min;94℃1 min,47℃45 s,72℃1min,共27个循环;最后于72℃延伸10 min。
1.5.3 PCR特异性试验 按上述反应体系与扩增条件,分别用E.sanguinicola、链球菌和葡萄球菌进行阴性对照,用双蒸水作为空白对照。
1.5.4 PCR敏感性试验 将提取的CMCC(B)32223和E1株的DNA模板经(Thermo)测定含量后,用双蒸水进行倍比稀释,设定含量分别为10 ng、1 ng、100 pg、10 pg、1 pg,按确定的扩增体系和扩增条件进行扩增,以出现特异扩增带的模板用量的最高稀释倍数计算可检出的肠球菌最低DNA含量。
1.6 临床分离株的检测
1.6.1 多重PCR方法检测 取53株猪的临床菌株、粪便菌株和鲜猪肉菌株按15 g/L方法进行扩增。取6 μ L PCR扩增产物,经含GoldView的15 g/L琼脂糖凝胶电泳后,进行结果判定。
1.6.2 16 S rRNA基因测序鉴定 按文献[10]报道的方法分别对53株分离菌株16 S rRNA进行PCR扩增、克隆,最终的重组菌送北京博尚生物技术有限公司完成测序,测定的DNA序列以双链碱基互补的结果为准。并将所测的肠球菌分离株的16 S rRNA基因序列提交GenBank数据库,与GenBank登陆的细菌的16 S rRNA序列进行BLAST,并同时将测定的肠球菌16 S rRNA基因序列与GenBank已登录的32种不同种类肠球菌菌株16 S rRNA序列进行同源性分析。
1.6.3 Vitek-32鉴定 取53株分离菌接种到BHI中增菌培养后,再接种到BHIA平板上,经24 h培养后挑取典型菌落用0.45%NaCl灭菌盐水配制成0.6~0.7麦氏单位,利用负压进样到革兰阳性细菌鉴定卡中,并放入Vitek-32全自动细菌鉴定系统的孵化箱中,进行鉴定。
2 结果
2.1 多重PCR扩增结果
12株标准株、2株粪肠球菌参考菌株、2株E.sanguinicola、葡萄球菌、链球菌以及双蒸水的PCR扩增结果见图1及图2。由图1、图2可见ATCC39212、CMCC(B)32223、E1-E12均有1 096 bp大小的特异性条带,与预计的肠球菌属特异性引物扩增大小片段相同;AT—CC39212、CMCC(B)32223、E4、E5、E7、E8、E10和E12株均有360 bp大小的片段,与设计的粪肠球菌种特异性引物扩增的大小相当;而E1、E2、E3、E6、E10和E11菌株均有215 bp大小特异性片段,与设计的屎肠球菌种特异性片段相似。E.sanguinicola也是肠球菌属的细菌,其也具有肠球菌属特异性1 096 bp条带,但在粪肠球菌和屎肠球菌种特异性引物扩增中,则没有出现相应的条带。链球菌和葡萄球菌是非肠球菌属的细菌,在肠球菌属和种的引物扩增中,与双蒸水一样,均未扩增出相应的特异性条带。
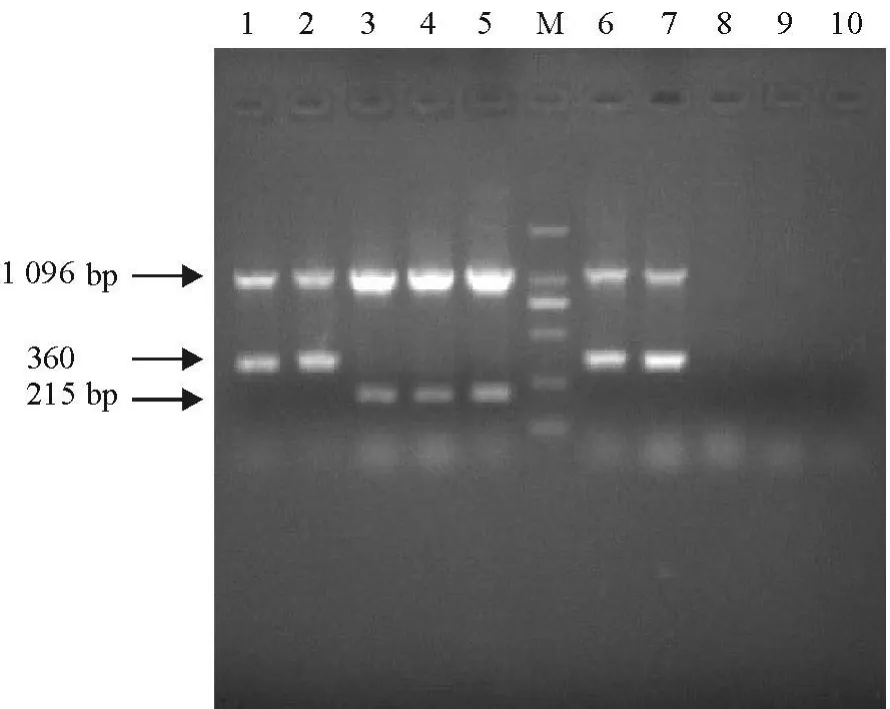
图1 多重PCR扩增结果Fig.1 M ultiple PCR results
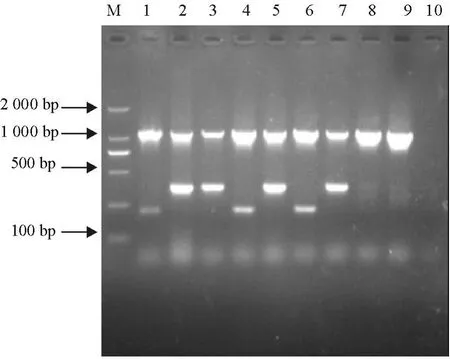
图2 多重PCR扩增结果 Fig.2 Multiple PCR results
2.2 多重PCR敏感性试验结果
分别取粪肠球菌和屎肠球菌各1株,提取DNA后用灭菌双蒸水分别稀释到含量为10 ng、1 ng、100 pg、10 pg、1 pg,进行敏感性试验,敏感性试验结果见图3。由图3可见,CMCC(29223)粪肠球菌在最终浓度为0.1 ng处仍有肠球菌属和种的特异性条带;而E1屎肠球菌在最终浓度为1 ng时有屎肠球菌属和种的特异性条带,而在浓度为0.1 ng时仅有屎肠球菌属特异性条带,没有种特异性条带。因此,建立的多重PCR方法对猪源粪肠球菌检测的敏感性为0.1ng,对屎肠球菌为1 ng。
2.3 临床分离株检测结果
53株分离于猪的临床菌株、粪便菌株和鲜猪肉菌株全部进行了多重PCR、Vitek-32和16 S rRNA基因测序鉴定,其中,部分菌株多重PCR检测结果见图4。由图可见,10株分离菌均具有肠球菌属和种特异性条带,据此可以判断L1-L4、L6、L7、L9、L10属于粪肠球菌E.faecalis(360 bp);L5、L8属于E.faecium(215 bp)。通过对53株分离菌的三种鉴定结果(见表3),发现多重PCR和16 S rRNA基因测序均能将53株分离菌鉴定到种的水平,而且二者符合率为100%。Vitek-32仅能将53株分离菌中的45株鉴定到种的水平(结果未报道),另有8株未能鉴定。在鉴定为肠球菌45株中,仅有33株与多重PCR和16SrRNA基因测序鉴定结果相符合,符合率为62.3%。在3种不同来源的肠球菌中,感染猪的临床菌株仅有14株与多重PCR和16 S rRNA基因测序鉴定结果一致,符合率为46.7%,尤其是感染猪的屎肠球菌,18株中仅有4株符合,符合率为22.3%,而对来源于鲜猪肉和粪便中的肠球菌,Vitek-32与多重PCR和16 S rRNA基因测序鉴定结果符合率较高,分别为80%和84.6%。
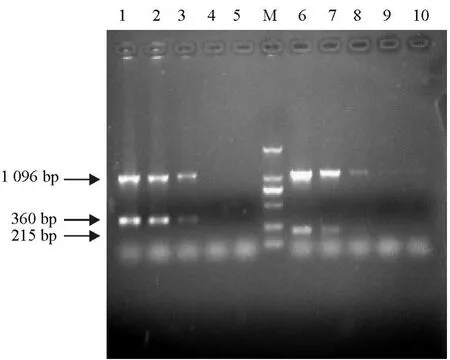
图3 PCR敏感性试验结果Fig.3 The results of PCR sensitivity test
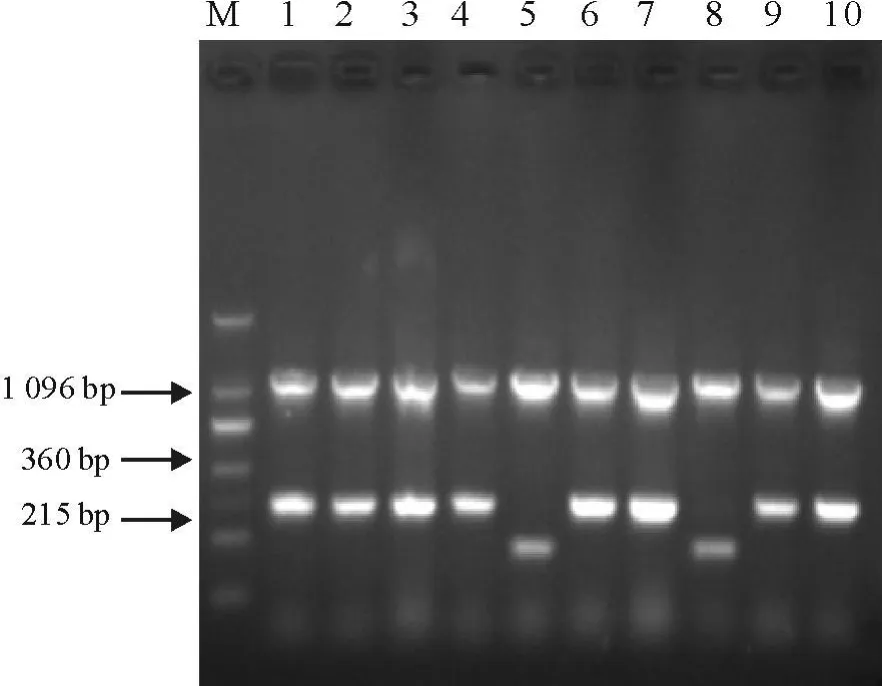
图4 PCR临床分离株检测结果Fig.4 PCR results of the isolated strains
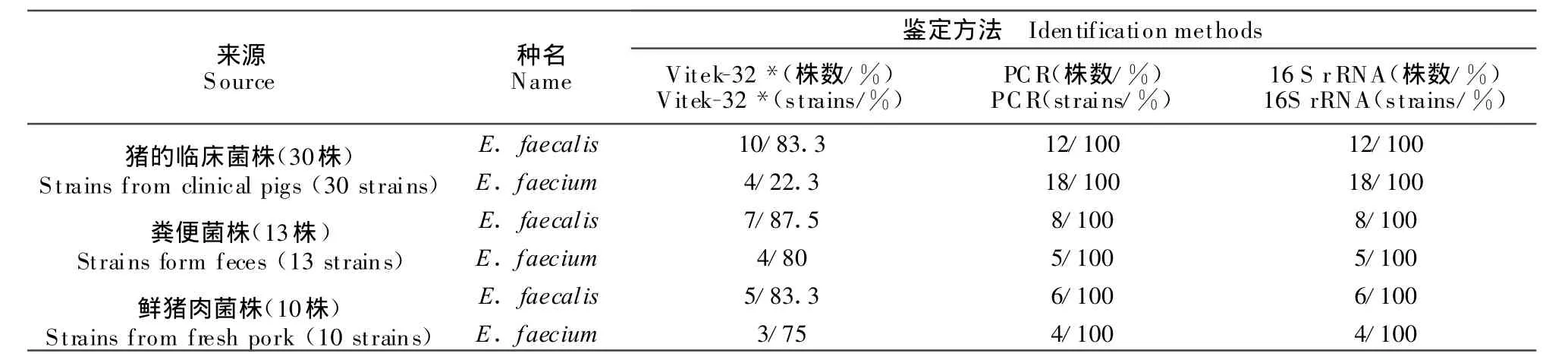
表3 临床分离株鉴定结果Table 3 results of identification for isolates
3 讨论
目前,肠球菌对人和动物的感染越来越多,耐药性越来越强,造成的损失也越来越大[11],这就迫切要求对感染的肠球菌进行快速、准确的鉴定。根据肠球菌16S rRNA基因序列及其他革兰阳性球菌的比较,设计肠球菌属特异性引物,并参考文献[8]报道的sodA的种特异性引物,通过优化反应条件,建立了能同时测定粪肠球菌和屎肠球菌的多重PCR方法,具有快速、灵敏、特异性强以及准确特点,为临床快速鉴定猪肠球菌感染奠定了基础。
16 S rRNA基因序列已被用作分子钟(molecular clock)去评估细菌间的相关性和鉴定未知细菌到属和种水平[12],多种细菌包括难于培养的细菌均成功用此方法进行了鉴定[13-14]。本研究建立的方法与6 S rRNA基因测序鉴定符合率100%,说明所建立的方法的可靠性;虽然,本方法与Vitek-32的符合率较低,据报道[15],Vitek-32对非人源肠球菌的鉴定结果常常受到质疑,特别是屎肠球菌。由于屎肠球菌表型差异较大,易已与屎肠球菌群其他细菌发生混淆[16],这也给生化鉴定方法增加了困难,本次测定的与感染猪的屎肠球菌符合率仅有22.3%,这可能是感染猪的屎肠球菌生化特性变异更大所致。
粪肠球菌和屎肠球菌是人、动物和各种环境介质优势菌群,也是感染猪的主要肠球菌种,本次使用的试验菌株均是分离于猪的临床菌株,并经过生化实验、WCPP、16 S rRNA、RAPD及毒力基因鉴定(资料未报),使用的阴性对照菌株葡萄球菌和链球菌均是感染猪临床菌株,临床上容易与肠球菌感染相混淆,乳球菌由于乳球菌很少引起猪的感染发病,其他的革兰阴性细菌可通过染色镜检可以区分,因此,本方法对于鉴定感染猪的粪肠球菌和屎肠球菌具有一定的特异性,有利于基层实验室对肠球菌病的诊断。
[1] 朱 波,强 华.肠球菌的毒力因子[J].检验医学与临床,2006,3(3):113-115.
[2] 袁钦华.一起粪肠球菌食物中毒的调查[J].实用预防医学,2001,8(5):394.
[3] 杨建忠,李凤荣,苑爱国.一起由肠球菌引起的食物中毒[J].内蒙古预防医学,1999,24(2):70.
[4] Carvalho M G S,Steigerwalt A G,Morey R E,et al.Characterization of Three NewEnterococcal Species,Enterococcus sp.nov.CDC PNS-E1,Enterococcus sp.nov.CDC PNS-E2,and Enterococcus sp.nov.CDC PNS-E3,Isolated from Human Clinical Specimens[J].Journal of Clinical Microbiololgy,2004,42(3):1192-1198.
[5] Danbing K E,Fracois J P,Fwancis M,et al.Development of a PCR assay for rapid detection of enterococci[J].Journal of Clinical Microbiololgy,1999,37:3497-3503.
[6] Klein G.Taxonomy,ecology and antibiotic resistance of enterococci from food and the gastro-intestinal tract[J].Int J Food Microbiol,2003,88:123-131.
[7] Garcia-Garrote F,Cercenado E,Bouza E,et al.Evaluation of a New System,VIT EK 2,for Identification and Antimicrobial Susceptibility Testing of Enterococci[J].Journal of Clinical Microbiology,2000,38(6):2108-2111.
[8] Charlene R J,Paula J F,John B B.Use of a Genus-and Species-Specific Multiplex PCR for Identification of Enterococci[J].Journal of Clinical Microbiology,2004,42(8):3558-3565.
[9] Messick J B,Cooper S K,Huntley M.Development and evaluation of a ploy merase chain reaction assay using the 16S rRNA gene for detection of Epery throzoon suis infection[J].J Vet Diag n Invest,1999,11(3):229-236.
[10] 程金平,王亚宾,刘 磊,等.应用16SrRNA基因序列分析鉴定猪源肠球菌[J].江西农业学报,2008,20(11):1-4.
[11] Fisher K,Phillips C.The ecology,epidemiology and virulence of Enterococcus[J].Microbiology,2009,155:1749-1757.
[12] Sacchi C T,Whitney A M,M ayer L W,et al.Sequencing of 16SrRNA gene:a rapid tool for identification of bacillus anthracis[J].Emerg Infect Dis,2002,8(10):1117-1123.
[13] William G W,Susan M B,Dale A P,et al.16 S Ribosomal DNA Amplification for Phylogenetic Study[J].Journal of bacteriology,1991,173(2):697-703.
[14] Cai H,Archambault M,Prescott J F.16S ribosomal RNA sequence-based identification of veterinary clinical bacteria[J].J Vet Diagn Invest,2003,15:465-469.
[15] 王亚宾,张红英,陈丽颖,等.16 S rRNA与Vitek-32对临床感染猪肠球菌鉴定结果比较[J].中国农学通报,2009,25(6):9-12.
[16] Hudson C R,Fedorka-Cray P J,Jackson-Hall M C,et al.A-nomalies in species identification of e-nterococci from veterinary sources using a commercial biochemical identification system[J].Lett Appl Microbiol,2003,36(4):245-250.
